Betalactamasa
| Serina betalactamasa | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
 Estructura de la betalactamasa de Streptomyces albus | ||||||||||
| Identificadores | ||||||||||
| Símbolo | dominio de β-lactamasa | |||||||||
| Pfam | PF00144 | |||||||||
| Clan pfam | CL0013 | |||||||||
| InterPro | IPR001466 | |||||||||
| PROSITE | PS00146 | |||||||||
| SCOP2 | 56601 / SCOPe / SUPFAM | |||||||||
| ||||||||||
| Metalo-beta-lactamasa | ||||||||
|---|---|---|---|---|---|---|---|---|
 Homotetrámero de metalo-beta-lactamasa L1, Stenotrophomonas | ||||||||
| Identificadores | ||||||||
| Símbolo | ? | |||||||
| Pfam | PF00753 | |||||||
| Clan pfam | CL0381 | |||||||
| InterPro | IPR001279 | |||||||
| ||||||||
| β-lactamasa | ||||||||
|---|---|---|---|---|---|---|---|---|
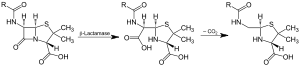 Acción de la β-lactamasa y descarboxilación del intermedio | ||||||||
| Identificadores | ||||||||
| CE no. | 3.5.2.6 | |||||||
| No CAS. | 9073-60-3 | |||||||
| Bases de datos | ||||||||
| IntEnz | Vista IntEnz | |||||||
| BRENDA | Entrada BRENDA | |||||||
| FÁCIL | NiceZyme vista | |||||||
| KEGG | Entrada KEGG | |||||||
| MetaCyc | camino metabólico | |||||||
| PRIAM | perfil | |||||||
| Estructuras PDB | RCSB PDB PDBe PDBsum | |||||||
| Ontología de genes | AmiGO / QuickGO | |||||||
| ||||||||
Los beta-lactamasas son enzimas ( EC 3.5.2.6 ) producidas por las bacterias que proporcionan multi- resistencia a antibióticos β-lactámicos , tales como penicilinas , cefalosporinas , cefamicinas , y carbapenems ( ertapenem ), aunque carbapenems son relativamente resistentes a beta-lactamasa. La betalactamasa proporciona resistencia a los antibióticos al romper la estructura de los antibióticos . Todos estos antibióticos tienen un elemento común en su estructura molecular: un anillo de cuatro átomos conocido como β-lactámico . Por hidrólisis, la enzima lactamasa abre el anillo β-lactámico, desactivando las propiedades antibacterianas de la molécula.
Los antibióticos betalactámicos se utilizan normalmente para tratar un amplio espectro de bacterias Gram positivas y Gram negativas .
Las betalactamasas producidas por organismos gramnegativos suelen secretarse, especialmente cuando hay antibióticos presentes en el medio ambiente. [1]
Estructura
La estructura de una serina β-lactamasa de Streptomyces viene dada por 1BSG . El pliegue alfa-beta ( InterPro : IPR012338 ) se asemeja al de una DD-transpeptidasa , a partir de la cual se cree que evolucionó la enzima. Las β-lactamasas de serina se agrupan por similitud de secuencia en los tipos A, C y D.
El otro tipo de beta-lactamasa es del tipo metalo ("tipo B"). La estructura de la metalo-beta-lactamasa 1 de Nueva Delhi viene dada por 6C89 . Se parece a una RNasa Z , de la que se cree que evolucionó.
Penicilinasa
La penicilinasa es un tipo específico de β-lactamasa, que muestra especificidad por las penicilinas , nuevamente al hidrolizar el anillo β-lactámico . Los pesos moleculares de las diversas penicilinasas tienden a agruparse cerca de los 50 kiloDaltons.
La penicilinasa fue la primera β-lactamasa en ser identificada. Fue aislado por primera vez por Abraham y Chain en 1940 a partir de E. coli Gram-negativas incluso antes de que la penicilina entrara en uso clínico, [2] pero la producción de penicilinasa se extendió rápidamente a bacterias que antes no la producían o que la producían sólo en raras ocasiones. Se desarrollaron betalactámicos resistentes a la penicilinasa, como la meticilina , pero ahora existe una resistencia generalizada incluso a estos.
Resistencia en bacterias Gram-negativas
Esta sección puede requerir una limpieza para cumplir con los estándares de calidad de Wikipedia . El problema específico es: mezcla confusa de clasificaciones estructurales y funcionales; Necesito un párrafo explicativo sobre lo que son estas clases . ( Septiembre de 2021 ) |
Entre las bacterias gramnegativas, la aparición de resistencia a las cefalosporinas de espectro extendido ha sido una preocupación importante. Apareció inicialmente en un número limitado de especies bacterianas ( E. cloacae , C. freundii , S. marcescens y P. aeruginosa ) que podían mutar para hiperproducir su β-lactamasa cromosómica de clase C. Unos años más tarde, apareció resistencia en especies bacterianas que no producían naturalmente enzimas AmpC ( K. pneumoniae , Salmonella spp., P. mirabilis ) debido a la producción de BLEE de tipo TEM o SHV (betalactamasas de espectro extendido). Característicamente, dicha resistencia ha incluido oxiimino- (por ejemplo , ceftizoxima ,cefotaxima , ceftriaxona y ceftazidima , así como el oxiimino-monobactam aztreonam ), pero no 7-alfa-metoxi-cefalosporinas ( cefamicinas ; en otras palabras, cefoxitina y cefotetan ); ha sido bloqueado por inhibidores como clavulanato , sulbactam o tazobactam y no involucró a carbapenémicos ni temocilina . Las β-lactamasas AmpC mediadas por cromosomas representan una nueva amenaza, ya que confieren resistencia a las 7-alfa-metoxi-cefalosporinas ( cefamicinas ) como cefoxitina o cefotetanpero no se ven afectados por los inhibidores de β-lactamasa disponibles comercialmente y, en cepas con pérdida de porinas de la membrana externa, pueden proporcionar resistencia a los carbapenémicos. [3]
Betalactamasa de espectro extendido (BLEE)
Los miembros de esta familia comúnmente expresan β-lactamasas (p. Ej., TEM-3, TEM-4 y SHV-1) que confieren resistencia a cefalosporinas de espectro expandido (espectro extendido). A mediados de la década de 1980, se detectó este nuevo grupo de enzimas, las β-lactamasas de espectro extendido (BLEE) (detectadas por primera vez en 1979). [4] La prevalencia de bacterias productoras de BLEE ha aumentado gradualmente en los hospitales de cuidados intensivos. [5] Las BLEE son betalactamasas que hidrolizan las cefalosporinas de espectro extendido con una cadena lateral de oxiimino. Estas cefalosporinas incluyen cefotaxima , ceftriaxona y ceftazidima , así como el aztreonam oxiimino-monobactam . Por tanto, las BLEE confieren multirresistenciaa estos antibióticos y oxiimino-beta lactámicos relacionados. En circunstancias típicas, derivan de genes para TEM-1, TEM-2 o SHV-1 por mutaciones que alteran la configuración de aminoácidos alrededor del sitio activo de estas β-lactamasas. Un conjunto más amplio de antibióticos β-lactámicos es susceptible de hidrólisis por estas enzimas. Recientemente se ha descrito un número creciente de BLEE que no son de linaje TEM o SHV. [6] Las BLEE son frecuentemente codificadas por plásmidos. Los plásmidos responsables de la producción de BLEE con frecuencia portan genes que codifican la resistencia a otras clases de fármacos (por ejemplo, aminoglucósidos). Por lo tanto, las opciones de antibióticos en el tratamiento de organismos productores de BLEE son extremadamente limitadas. Carbapenémicosson el tratamiento de elección para las infecciones graves debidas a organismos productores de BLEE; sin embargo, recientemente se han notificado aislamientos resistentes a carbapenémicos (principalmente resistentes a ertapenem ). Los organismos productores de BLEE pueden parecer susceptibles a algunas cefalosporinas de espectro extendido . Sin embargo, el tratamiento con tales antibióticos se ha asociado con altas tasas de fracaso. [ cita requerida ]
Tipos
TEM betalactamasas (clase A)
TEM-1 es la beta-lactamasa más común en bacterias Gram-negativas . Hasta el 90% de la resistencia a la ampicilina en E. coli se debe a la producción de TEM-1. [7] También es responsable de la resistencia a la ampicilina y la penicilina que se observa en H. influenzae y N. gonorrhoeae en cantidades cada vez mayores. Aunque las betalactamasas de tipo TEM se encuentran con mayor frecuencia en E. coli y K. pneumoniae , también se encuentran en otras especies de bacterias gramnegativas con una frecuencia creciente. Las sustituciones de aminoácidos responsables de la beta lactamasa de espectro extendido (BLEE)el fenotipo se agrupa alrededor del sitio activo de la enzima y cambia su configuración, lo que permite el acceso a sustratos de oxiimino-beta-lactámicos. La apertura del sitio activo a sustratos de betalactámicos también aumenta típicamente la susceptibilidad de la enzima a los inhibidores de la betalactamasa, como el ácido clavulánico. Las sustituciones de un solo aminoácido en las posiciones 104, 164, 238 y 240 producen el fenotipo BLEE, pero las BLEE con el espectro más amplio suelen tener más de una sustitución de un solo aminoácido. En base a diferentes combinaciones de cambios, actualmente se han descrito 140 enzimas de tipo TEM. TEM-10, TEM-12 y TEM-26 se encuentran entre los más comunes en los Estados Unidos. [8] [9] [10] El término TEM proviene del nombre del paciente ateniense (Temoniera) del que se recuperó el aislado en 1963.[11]
Betalactamasas SHV (clase A)
SHV-1 comparte el 68 por ciento de sus aminoácidos con TEM-1 y tiene una estructura general similar. La betalactamasa SHV-1 se encuentra con mayor frecuencia en K. pneumoniae y es responsable de hasta el 20% de la resistencia a ampicilina mediada por plásmidos en esta especie. Las BLEE de esta familia también tienen cambios de aminoácidos alrededor del sitio activo, más comúnmente en las posiciones 238 o 238 y 240. Se conocen más de 60 variedades de SHV. SHV-5 y SHV-12 se encuentran entre los más comunes. [8]
Betalactamasas CTX-M (clase A)
Estas enzimas se nombraron por su mayor actividad contra la cefotaxima que otros sustratos de oxiimino-beta-lactámicos (p. Ej., Ceftazidima , ceftriaxona o cefepima ). En lugar de surgir por mutación, representan ejemplos de adquisición de plásmidos de genes de beta-lactamasa que normalmente se encuentran en el cromosoma de la especie Kluyvera , un grupo de organismos comensales raramente patógenos. Estas enzimas no están muy relacionadas con las betalactamasas TEM o SHV, ya que muestran solo aproximadamente un 40% de identidad con estas dos betalactamasas comúnmente aisladas. Actualmente se conocen más de 80 enzimas CTX-M. A pesar de su nombre, algunos son más activos con ceftazidima que con cefotaxima.. Se han encontrado principalmente en cepas de Salmonella enterica serovar Typhimurium y E. coli , pero también se han descrito en otras especies de Enterobacteriaceae y son el tipo BLEE predominante en partes de América del Sur. (También se ven en Europa del Este) CTX-M-14, CTX-M-3 y CTX-M-2 son los más extendidos. CTX-M-15 es actualmente (2006) el tipo más extendido en E. coli en el Reino Unido y es ampliamente prevalente en la comunidad. [12] Se ha descubierto que un ejemplo de beta-lactamasa CTX-M-15, junto con IS Ecp1 , se ha transpuesto recientemente al cromosoma de Klebsiella pneumoniae ATCC BAA-2146. [13]
Betalactamasas OXA (clase D)
Las betalactamasas OXA fueron reconocidas durante mucho tiempo como una variedad de betalactamasas menos común, pero también mediada por plásmidos, que podía hidrolizar la oxacilina y las penicilinas antiestafilocócicas relacionadas. Estas beta-lactamasas se diferencian de las enzimas TEM y SHV en que pertenecen a la clase molecular D y al grupo funcional 2d. Las betalactamasas de tipo OXA confieren resistencia a ampicilina y cefalotina y se caracterizan por su alta actividad hidrolítica frente a oxacilina y cloxacilina y el hecho de que son poco inhibidas por el ácido clavulánico . Las sustituciones de aminoácidos en las enzimas OXA también pueden producir el fenotipo BLEE. Si bien la mayoría de las BLEE se han encontrado en E. coli ,K. pneumoniae y otras Enterobacteriaceae , las BLEE de tipo OXA se han encontrado principalmente en P. aeruginosa . Se han encontrado BLEE de tipo OXA principalmente en Pseudomonas aeruginosaaislamientos de Turquía y Francia. La familia de betalactamasas OXA se creó originalmente como un grupo fenotípico en lugar de genotípico para unas pocas betalactamasas que tenían un perfil de hidrólisis específico. Por lo tanto, hay tan solo un 20% de homología de secuencia entre algunos de los miembros de esta familia. Sin embargo, las adiciones recientes a esta familia muestran cierto grado de homología con uno o más de los miembros existentes de la familia de betalactamasas OXA. Algunos confieren resistencia predominantemente a ceftazidima, pero OXA-17 confiere mayor resistencia a cefotaxima y cefepima que resistencia a ceftazidima.
Otros
Se han descrito otras BLEE mediadas por plásmidos, como PER, VEB, GES e IBC beta-lactamasas, pero son poco frecuentes y se han encontrado principalmente en P. aeruginosa y en un número limitado de sitios geográficos. PER-1 en aislamientos en Turquía, Francia e Italia; VEB-1 y VEB-2 en cepas del sudeste asiático; y GES-1, GES-2 e IBC-2 en aislados de Sudáfrica, Francia y Grecia. PER-1 también es común en especies de acinetobacter multirresistentes en Corea y Turquía. Algunas de estas enzimas también se encuentran en Enterobacteriaceae, mientras que otras BESB poco comunes (como BES-1, IBC-1, SFO-1 y TLA-1) se han encontrado solo en Enterobacteriaceae.
Tratamiento
Si bien los organismos productores de BLEE se asociaban anteriormente con los hospitales y la atención institucional, estos organismos ahora se encuentran cada vez más en la comunidad. Las E. coli positivas para CTX-M-15 son una causa de infecciones urinarias adquiridas en la comunidad en el Reino Unido, [12] y tienden a ser resistentes a todos los antibióticos lactámicos β orales, así como a las quinolonas y sulfonamidas . Las opciones de tratamiento pueden incluir nitrofurantoína , fosfomicina , mecilinam y cloranfenicol . En caso de desesperación, también se pueden usar inyecciones de ertapenem o gentamicina una vez al día .
Β-lactamasas resistentes a inhibidores
Aunque las β-lactamasas resistentes a inhibidores no son BLEE, a menudo se comentan con las BLEE porque también son derivados de las enzimas clásicas de tipo TEM o SHV. A estas enzimas se les dio al principio la designación IRT para TEM β-lactamasa resistente a inhibidores; sin embargo, todos han sido posteriormente renombrados con designaciones TEM numéricas. Hay al menos 19 β-lactamasas TEM resistentes a inhibidores distintas. Se han encontrado β-lactamasas TEM resistentes a inhibidores principalmente en aislados clínicos de E. coli , pero también en algunas cepas de K. pneumoniae , Klebsiella oxytoca , P. mirabilis y Citrobacter freundii . Aunque las variantes de TEM resistentes a inhibidores son resistentes a la inhibición por ácido clavulánicoy sulbactam , mostrando así resistencia clínica a las combinaciones de betalactámicos-inhibidores de lactamasa de amoxicilina - clavulanato ( co-amoxiclav ), ticarcilina - clavulanato ( co-ticarclav ) y ampicilina / sulbactam , normalmente siguen siendo susceptibles a la inhibición por tazobactam y posteriormente la combinación de piperacilina / tazobactam , [ cita requerida ]aunque se ha descrito resistencia. Esta ya no es una epidemiología principalmente europea, se encuentra a menudo en las partes del norte de Estados Unidos y debe analizarse con infecciones urinarias complejas. [9]
Β-lactamasas tipo AmpC (clase C)
Las β-lactamasas de tipo AmpC suelen aislarse de bacterias gramnegativas resistentes a cefalosporinas de espectro extendido. Las β-lactamasas AmpC (también denominadas clase C o grupo 1) se codifican típicamente en el cromosoma de muchas bacterias Gram negativas, incluidas las especies de Citrobacter , Serratia y Enterobacter, en las que su expresión suele ser inducible ; también puede ocurrir en Escherichia coli pero no suele ser inducible, aunque puede estar hiperexpresado. Las β-lactamasas de tipo AmpC también pueden transportarse en plásmidos. [3]Las β-lactamasas AmpC, a diferencia de las BLEE, hidrolizan cefalosporinas de amplio y extendido espectro (cefamicinas y oxiimino-β-lactámicos) pero no son inhibidas por inhibidores de β-lactamasa como el ácido clavulánico . Los organismos β-lactamasa de tipo AmpC a menudo se agrupan clínicamente mediante el acrónimo "SPACE": Serratia , Pseudomonas o Proteus , Acinetobacter , Citrobacter y Enterobacter .
Carbapenemasas
Los carbapenémicos son famosos por su estabilidad frente a las β-lactamasas AmpC y las β-lactamasas de espectro extendido. Las carbapenemasas son un grupo diverso de β-lactamasas que son activas no solo contra las oxiiminocefalosporinas y cefamicinas, sino también contra los carbapenémicos. Aztreonam es estable a las metalo-β-lactamasas, pero muchos productores de IMP y VIM son resistentes debido a otros mecanismos. Anteriormente se creía que las carbapenemasas se derivaban solo de las clases A, B y D, pero se ha descrito una carbapenemasa de clase C.
Carbapenemasas tipo IMP (metalo-β-lactamasas) (clase B)
Las carbapenemasas de tipo IMP mediadas por plásmidos (IMP significa activo sobre imipenem), de las cuales se conocen actualmente 19 variedades, se establecieron en Japón en la década de 1990 tanto en organismos gramnegativos entéricos como en especies de Pseudomonas y Acinetobacter . Las enzimas IMP se propagan lentamente a otros países del Lejano Oriente, se informaron en Europa en 1997 y se han encontrado en Canadá y Brasil.
VIM (metalo-β-lactamasa codificada por integrones de Verona) (Clase B)
Una segunda familia creciente de carbapenemasas, la familia VIM, se informó en Italia en 1999 y ahora incluye 10 miembros, que tienen una amplia distribución geográfica en Europa, América del Sur y el Lejano Oriente y se han encontrado en los Estados Unidos. VIM-1 se descubrió en P. aeruginosa en Italia en 1996; desde entonces, VIM-2 - ahora la variante predominante - se encontró repetidamente en Europa y el Lejano Oriente; VIM-3 y -4 son variantes menores de VIM-2 y -1, respectivamente. Las enzimas VIM se encuentran principalmente en P. aeruginosa , también P. putida y, en menor grado, en Enterobacteriaceae. [14]
La diversidad de secuencias de aminoácidos es de hasta el 10% en la familia VIM, el 15% en la familia IMP y el 70% entre VIM e IMP. Sin embargo, las enzimas de ambas familias son similares. Ambos están asociados a integrones, a veces dentro de plásmidos. Ambos hidrolizan todos los betalactámicos excepto los monobactamas y evaden todos los inhibidores de betalactámicos. Las enzimas VIM se encuentran entre las MBL más ampliamente distribuidas, y se han informado> 40 variantes de VIM. Los estudios bioquímicos y biofísicos revelaron que las variantes de VIM tienen solo pequeñas variaciones en sus parámetros cinéticos, pero diferencias sustanciales en sus estabilidades térmicas y perfiles de inhibición. [15]
Grupo de β-lactamasas OXA (oxacilinasa) (clase D)
El grupo OXA de β-lactamasas se encuentra principalmente en las especies de Acinetobacter y se divide en dos grupos. Las carbapenemasas OXA hidrolizan los carbapenémicos in vitro muy lentamente , y las CIM altas observadas para algunos hospedadores Acinetobacter (> 64 mg / L) pueden reflejar mecanismos secundarios. En ocasiones, en los aislados clínicos se incrementan mediante mecanismos de resistencia adicionales, como la impermeabilidad o el flujo de salida. Las carbapenemasas OXA también tienden a tener una eficiencia hidrolítica reducida frente a las penicilinas y cefalosporinas. [dieciséis]
KPC ( K. pneumoniae carbapenemase) (clase A)
Algunas enzimas de clase A, la mayoría de las cuales son las enzimas KPC mediadas por plásmidos, también son carbapenemasas eficaces. Se conocen diez variantes, KPC-2 a KPC-11, y se distinguen por una o dos sustituciones de aminoácidos (KPC-1 se volvió a secuenciar en 2008 y se encontró que era 100% homóloga a las secuencias publicadas de KPC-2). KPC-1 se encontró en Carolina del Norte, KPC-2 en Baltimore y KPC-3 en Nueva York. Tienen solo un 45% de homología con las enzimas SME y NMC / IMI y, a diferencia de ellas, pueden ser codificadas por plásmidos autotransmisibles.
En febrero de 2009 [actualizar], la carbapenemasa de Klebsiella pneumoniae ( KPC ) de clase A a nivel mundial ha sido la carbapenemasa más común y se detectó por primera vez en 1996 en Carolina del Norte , EE. UU. [17] Una publicación de 2010 indicó que las enterobacterias productoras de KPC se estaban volviendo comunes en los Estados Unidos. [18]
CMY (clase C)
La carbapenemasa de primera clase C se describió en 2006 y se aisló de una cepa virulenta de Enterobacter aerogenes . [19] Se transporta en un plásmido, pYMG-1, y por lo tanto es transmisible a otras cepas bacterianas. [20]
SME (enzimas de Serratia marcescens), IMI (β-lactamasa hidrolizante de IMIpenem), NMC y CcrA
En general, estos tienen poca importancia clínica.
CcrA (CfiA). Su gen ocurre en ca. 1-3% de los aislados de B. fragilis , pero pocos producen la enzima, ya que la expresión exige una migración adecuada de una secuencia de inserción. El CcrA se conocía antes de que se introdujera el imipenem y los productores han mostrado poco aumento posterior.
NDM-1 (metalo-β-lactamasa de Nueva Delhi) (clase B)
Descrito originalmente en Nueva Delhi en 2009, este gen ahora está muy extendido en Escherichia coli y Klebsiella pneumoniae de India y Pakistán. A mediados de 2010, se introdujeron bacterias portadoras de NDM-1 en otros países (incluidos los Estados Unidos y el Reino Unido), probablemente debido a la gran cantidad de turistas que viajan por todo el mundo y que pueden haber adquirido la cepa del medio ambiente. ya que se han encontrado cepas que contienen el gen NDM-1 en muestras ambientales en la India. [21] Los NDM tienen varias variantes que comparten diferentes propiedades. [15]
Tratamiento de BLEE / AmpC / carbapenemasas
Visión general
En general, se sospecha que un aislado es productor de BLEE cuando muestra susceptibilidad in vitro a las cefalosporinas de segunda generación ( cefoxitina , cefotetan ) pero resistencia a las cefalosporinas de tercera generación y al aztreonam . Además, se deben sospechar estas cepas cuando falla el tratamiento con estos agentes para infecciones gramnegativas a pesar de la susceptibilidad in vitro informada . Una vez que se detecta una cepa productora de BLEE, el laboratorio debe informarla como "resistente" a todas las penicilinas, cefalosporinas y aztreonam, incluso si se analiza (in vitro) como susceptible. [ cita requerida ] Resistencia asociada alos aminoglucósidos y trimetoprim - sulfametoxazol , así como la alta frecuencia de coexistencia de resistencia a las fluoroquinolonas , crean problemas. Los inhibidores de betalactamasas como clavulanato , sulbactam y tazobactam inhiben in vitro la mayoría de las BLEE, pero no se puede confiar en la eficacia clínica de las combinaciones de betalactámicos / inhibidores de betalactamasas para la terapia. Cefamicinas ( cefoxitina y cefotetan) no son hidrolizados por la mayoría de las BLEE, sino que son hidrolizados por la β-lactamasa de tipo AmpC asociada. Además, las combinaciones de β-lactámicos / inhibidores de β-lactamasa pueden no ser efectivas contra organismos que producen β-lactamasa de tipo AmpC. A veces, estas cepas disminuyen la expresión de proteínas de la membrana externa, haciéndolas resistentes a las cefamicinas. Los estudios in vivo han arrojado resultados mixtos contra K. pneumoniae productora de BLEE . (La cefepima , una cefalosporina de cuarta generación, ha demostrado estabilidad in vitro en presencia de muchas cepas de BLEE / AmpC). Actualmente, los carbapenémicosson, en general, considerados como el agente preferido para el tratamiento de infecciones debidas a organismos productores de BLEE. Los carbapenémicos son resistentes a la hidrólisis mediada por BLEE y exhiben una excelente actividad in vitro contra cepas de Enterobacteriaceae que expresan BLEE. [ cita requerida ]
Según genes
BLEE
Las cepas que sólo producen BLEE son susceptibles a cefamicinas y carbapenémicos in vitro y muestran poco o ningún efecto de inóculo con estos agentes.
Para los organismos que producen BLEE de tipo TEM y SHV , la sensibilidad in vitro aparente a la cefepima y a la piperacilina / tazobactam es común, pero ambos fármacos muestran un efecto de inóculo, con una susceptibilidad disminuida a medida que el tamaño del inóculo aumenta de 10 5 a 10 7 organismos.
Las cepas con algunas BLEE de tipo CTX-M y de tipo OXA son resistentes a la cefepima en las pruebas, a pesar del uso de un inóculo estándar.
Β-lactamasas resistentes a inhibidores
Aunque las variantes de TEM resistentes a inhibidores son resistentes a la inhibición por ácido clavulánico y sulbactam , por lo que muestran resistencia clínica a las combinaciones betalactámico-inhibidor de beta lactamasa de amoxicilina - clavulánico ( Co-amoxiclav ), ticarcilina - clavulánico y ampicilina / sulbactam , siguen siendo susceptibles a la inhibición por tazobactam y posteriormente a la combinación de piperacilina / tazobactam .
AmpC
Las cepas productoras de AmpC son típicamente resistentes a las oxiimino-beta-lactamas ya las cefamicinas y son susceptibles a los carbapenémicos ; sin embargo, la expresión disminuida de la porina también puede hacer que una cepa de este tipo sea resistente a los carbapenémicos.
Carbapenemasas
Las cepas con carbapenemasas de tipo IMP, VIM y OXA suelen seguir siendo susceptibles. La resistencia a los antibióticos no betalactámicos es común en las cepas que producen cualquiera de estas enzimas, por lo que las opciones alternativas para la terapia sin betalactámicos deben determinarse mediante pruebas de sensibilidad directa. La resistencia a las fluoroquinolonas y aminoglucósidos es especialmente alta.
Según especie
Escherichia coli o Klebsiella
Para las infecciones causadas por especies de Escherichia coli o Klebsiella productoras de BLEE , el tratamiento con imipenem o meropenem se ha asociado con los mejores resultados en términos de supervivencia y eliminación bacteriológica. La cefepima y la piperacilina / tazobactam han tenido menos éxito. La ceftriaxona , la cefotaxima y la ceftazidima han fallado incluso con más frecuencia, a pesar de la susceptibilidad del organismo al antibiótico in vitro . Varios informes han documentado el fracaso de la terapia con cefamicina como resultado de la resistencia debido a la pérdida de porina. Algunos pacientes han respondido aaminoglucósido o quinolona terapia, pero, en una reciente comparación de ciprofloxacina y imipenem de bacteriemia que implica un productoras de ESBL K. pneumoniae , imipenem produjeron el mejor resultado
Pseudomonas aeruginosa
Se han realizado pocos estudios clínicos para definir la terapia óptima para las infecciones causadas por cepas de Pseudomonas aeruginosa productoras de BLEE .
Utilizar como farmacéutico
En 1957, en medio de la preocupación por las reacciones alérgicas a los antibióticos que contienen penicilina, se vendió una beta-lactamasa como antídoto bajo la marca neutrapen. [22] Se teorizó que la descomposición de la penicilina por la enzima trataría la reacción alérgica. [23] Si bien no fue útil en el shock anafiláctico agudo, mostró resultados positivos en casos de urticaria y dolor articular que se sospechaba que era causado por alergia a la penicilina. [24] [25] Se propuso su uso en casos pediátricos en los que se descubrió alergia a la penicilina tras la administración de la vacuna contra la polio, que utilizaba penicilina como conservante. [26] Sin embargo, algunos pacientes desarrollaron alergias al neutrapen. [27] [28] El Hospital de Albany lo eliminó de su formulario en 1960, solo dos años después de agregarlo, citando su falta de uso. [29] Algunos investigadores continuaron usándolo en experimentos sobre la resistencia a la penicilina hasta 1972. [30] Fue retirado voluntariamente del mercado estadounidense por 3M Pharmaceuticals en 1997. [31]
Detección
La actividad enzimática de la beta-lactamasa se puede detectar usando nitrocefina , un sustrato de cefalosporina cromogénico que cambia de color de amarillo a rojo tras la hidrólisis mediada por beta-lactamasa. [32]
Evolución
Las betalactamasas son enzimas bacterianas antiguas. Las metalo β-lactamasas ("clase B") son todas estructuralmente similares a la RNasa Z y pueden haber evolucionado a partir de ella. De las tres subclases B1, B2 y B3, se teoriza que B1 y B2 evolucionaron hace unos mil millones de años , mientras que B3 parece haber surgido de forma independiente, posiblemente antes de la divergencia de las eubacterias Gram-positivas y Gram-negativas alrededor de dos mil millones. hace años que. [33] PNGM-1 (Metalo-β-lactamasa-1 de Papua Nueva Guinea) tiene actividades de metalo-β-lactamasa (MBL) y tRNasa Z, lo que sugiere que se cree que PNGM-1 ha evolucionado a partir de una tRNasa Z, y que la actividad MBL B3 de PNGM-1 es una actividad promiscua y se cree que las MBL de la subclase B3 han evolucionado a través de la actividad PNGM-1. [34]Las subclases B1 y B3 se han subdividido aún más. [35]
Las betalactamasas de serina (clases A, C y D) parecen haber evolucionado a partir de DD-transpeptidasas , que son proteínas de unión a penicilina que participan en la biosíntesis de la pared celular y, como tales, son uno de los principales objetivos de los antibióticos betalactámicos. [36] Estas tres clases muestran una similitud de secuencia indetectable entre sí, pero aún pueden compararse usando homología estructural. Los grupos A y D son taxones hermanos y el grupo C divergió antes que A y D. [37] Estas enzimas basadas en serina, como las betalactamasas del grupo B, son de origen antiguo y se teoriza que evolucionaron hace unos dos mil millones de años. [38]
Se teoriza que el grupo OXA (en la clase D) en particular ha evolucionado en los cromosomas y se ha trasladado a los plásmidos en al menos dos ocasiones distintas. [39]
Etimología
El "β" ( beta ) se refiere a la posición del nitrógeno en el segundo carbono del anillo. La lactama es una mezcla de lactona (del latín lactis , leche , ya que el ácido láctico se aisló de la leche agria) y amida . El sufijo -asa , que indica una enzima, se deriva de diastasa (del griego diastasis , "separación"), la primera enzima descubierta en 1833 por Payen y Persoz. [40]
Ver también
- inhibidor de β-lactamasa
- E. coli productora de BLEE
- Nitrocefina
Referencias
- ^ Neu HC (junio de 1969). "Efecto de la ubicación de la beta-lactamasa en Escherichia coli sobre la sinergia de la penicilina" . Microbiología aplicada . 17 (6): 783–6. doi : 10.1128 / AEM.17.6.783-786.1969 . PMC 377810 . PMID 4894721 .
- ^ Abraham EP, Cadena E (1940). "Una enzima de una bacteria capaz de destruir la penicilina". Naturaleza . 46 (3713): 837. Bibcode : 1940Natur.146..837A . doi : 10.1038 / 146837a0 . S2CID 4070796 .
- ↑ a b Philippon A, Arlet G, Jacoby GA (enero de 2002). "Beta-lactamasas de tipo AmpC determinadas por plásmido" . Agentes antimicrobianos y quimioterapia . 46 (1): 1–11. doi : 10.1128 / AAC.46.1.1-11.2002 . PMC 126993 . PMID 11751104 .
- ^ Sanders CC, Sanders WE (junio de 1979). "Aparición de resistencia a cefamandol: posible papel de las betalactamasas inducibles por cefoxitina" . Agentes antimicrobianos y quimioterapia . 15 (6): 792–7. doi : 10.1128 / AAC.15.6.792 . PMC 352760 . PMID 314270 .
- ^ Spadafino JT, Cohen B, Liu J, Larson E (2014). "Tendencias temporales y factores de riesgo para Escherichia coli productora de betalactamasa de espectro extendido en adultos con infecciones del tracto urinario asociadas a catéter" . Resistencia a los antimicrobianos y control de infecciones . 3 (1): 39. doi : 10.1186 / s13756-014-0039-y . PMC 4306238 . PMID 25625011 .
- ^ Emery CL, Weymouth LA (agosto de 1997). "Detección e importancia clínica de betalactamasas de espectro extendido en un centro médico de tercer nivel" . Revista de microbiología clínica . 35 (8): 2061–7. doi : 10.1128 / JCM.35.8.2061-2067.1997 . PMC 229903 . PMID 9230382 .
- ^ Cooksey R, Swenson J, Clark N, Gay E, Thornsberry C (mayo de 1990). "Patrones y mecanismos de resistencia a betalactámicos entre aislamientos de Escherichia coli de hospitales de Estados Unidos" . Agentes antimicrobianos y quimioterapia . 34 (5): 739–45. doi : 10.1128 / AAC.34.5.739 . PMC 171683 . PMID 2193616 .
- ^ a b Paterson DL, Hujer KM, Hujer AM, Yeiser B, Bonomo MD, Rice LB, Bonomo RA (noviembre de 2003). "Betalactamasas de espectro extendido en aislados del torrente sanguíneo de Klebsiella pneumoniae de siete países: predominio y prevalencia generalizada de betalactamasas de tipo SHV y CTX-M" . Agentes antimicrobianos y quimioterapia . 47 (11): 3554–60. doi : 10.1128 / AAC.47.11.3554-3560.2003 . PMC 253771 . PMID 14576117 .
- ^ a b Bradford PA (octubre de 2001). "Betalactamasas de espectro extendido en el siglo XXI: caracterización, epidemiología y detección de esta importante amenaza de resistencia" . Revisiones de microbiología clínica . 14 (4): 933–51, índice. doi : 10.1128 / CMR.14.4.933-951.2001 . PMC 89009 . PMID 11585791 .
- ^ Jacoby GA, Munoz-Price LS (enero de 2005). "Las nuevas betalactamasas". La Revista de Medicina de Nueva Inglaterra . 352 (4): 380–91. doi : 10.1056 / NEJMra041359 . PMID 15673804 .
- ↑ Ruiz, Joaquim (2018). "Etimología: TEM" . Enfermedades infecciosas emergentes . 24 (4): 709. doi : 10.3201 / eid2404.et2404 .
- ^ a b Woodford N, Ward E, Kaufmann ME, et al. "Caracterización molecular de aislamientos de Escherichia coli que producen β-lactamasa de espectro extendido (BLEE) CTX-M-15 en el Reino Unido" (PDF) . Agencia de Protección de la Salud. Archivado desde el original (PDF) el 15 de junio de 2007 . Consultado el 19 de noviembre de 2006 .
- ^ Hudson CM, Bent ZW, Meagher RJ, Williams KP (7 de junio de 2014). "Determinantes de resistencia y elementos genéticos móviles de una cepa de Klebsiella pneumoniae que codifica NDM-1" . PloS One . 9 (6): e99209. Código bibliográfico : 2014PLoSO ... 999209H . doi : 10.1371 / journal.pone.0099209 . PMC 4048246 . PMID 24905728 .
- ^ Ghaith DM, Mohamed ZK, Farahat MG, Aboulkasem Shahin W, Mohamed HO (marzo de 2019). "Colonización de la microbiota intestinal con enterobacterias productoras de carbapenemasas en unidades de cuidados intensivos pediátricos en El Cairo, Egipto". Revista árabe de gastroenterología . 20 (1): 19-22. doi : 10.1016 / j.ajg.2019.01.002 . PMID 30733176 .
- ^ a b Makena A, Düzgün AÖ, Brem J, McDonough MA, Rydzik AM, Abboud MI, et al. (Diciembre de 2015). "La comparación de variantes de Verona Integron-Borne Metalo-β-Lactamase (VIM) revela diferencias en los perfiles de estabilidad e inhibición" . Agentes antimicrobianos y quimioterapia . 60 (3): 1377–84. doi : 10.1128 / AAC.01768-15 . PMC 4775916 . PMID 26666919 .
- ^ Santillana E, Beceiro A, Bou G, Romero A (marzo de 2007). "La estructura cristalina de la carbapenemasa OXA-24 revela conocimientos sobre el mecanismo de hidrólisis de carbapenem" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (13): 5354–9. Código Bibliográfico : 2007PNAS..104.5354S . doi : 10.1073 / pnas.0607557104 . PMC 1838445 . PMID 17374723 .
- ^ Nordmann P, Cuzon G, Naas T (abril de 2009). "La amenaza real de las bacterias productoras de carbapenemasas de Klebsiella pneumoniae". La lanceta. Enfermedades infecciosas . 9 (4): 228–36. doi : 10.1016 / S1473-3099 (09) 70054-4 . PMID 19324295 .
- ^ Cuzon G, Naas T, Nordmann P (febrero de 2010). "[Carbapenemasas KPC: ¿qué está en juego en la microbiología clínica?]". Pathologie-Biologie (en francés). 58 (1): 39–45. doi : 10.1016 / j.patbio.2009.07.026 . PMID 19854586 .
- ^ Kim JY, Jung HI, An YJ, Lee JH, Kim SJ, Jeong SH, et al. (Mayo de 2006). "Base estructural para el espectro de sustrato extendido de CMY-10, una beta-lactamasa de clase C codificada por plásmido" . Microbiología molecular . 60 (4): 907–16. doi : 10.1111 / j.1365-2958.2006.05146.x . PMID 16677302 . S2CID 44982704 .
- ^ Lee JH, Jung HI, Jung JH, Park JS, Ahn JB, Jeong SH, et al. (2004). "Difusión de betalactamasa transferible tipo AmpC (CMY-10) en un hospital coreano". Resistencia a fármacos microbianos . 10 (3): 224-30. doi : 10.1089 / mdr.2004.10.224 . PMID 15383166 .
- ^ Walsh TR, Weeks J, Livermore DM, Toleman MA (mayo de 2011). "Diseminación de bacterias positivas NDM-1 en el medio ambiente de Nueva Delhi y sus implicaciones para la salud humana: un estudio de prevalencia de puntos ambientales". La lanceta. Enfermedades infecciosas . 11 (5): 355–62. doi : 10.1016 / S1473-3099 (11) 70059-7 . PMID 21478057 .
- ^ "NUEVA DROGA LUCHA CONTRA LAS ENFERMEDADES DE PENICILLINA; Experto en antibióticos encuentra Neutrapen eficaz en el efecto secundario de la inyección ALERGIA RISE ESTUDIADO US Aide Reporta aumento en la reacción a la penicilina y sustancias similares Reacciones severas Pocas algunas reacciones severas (publicado en 1957)" . The New York Times . 4 de octubre de 1957. ISSN 0362-4331 . Consultado el 24 de diciembre de 2020 .
- ^ Hyman AL (febrero de 1959). "Choque anafiláctico después de la terapia con penicilinasa". Revista de la Asociación Médica Estadounidense . 169 (6): 593–4. doi : 10.1001 / jama.1959.73000230003011a . PMID 13620512 .
- ^ Friedlaender S (abril de 1959). "Penicilinasa en el tratamiento de reacciones alérgicas a la penicilina". El diario de la alergia . 30 (2): 181–7. doi : 10.1016 / 0021-8707 (59) 90087-5 . PMID 13630649 .
- ^ Pediatría, Academia Americana de (1 de octubre de 1958). "Un nuevo concepto en el tratamiento de las reacciones a la penicilina: uso de penicilinasa" . Pediatría . 22 (4): 658. ISSN 0031-4005 .
- ^ Zimmerman MC (agosto de 1958). "Alergia probada por penicilinasa a la penicilina en la vacuna contra la poliomielitis". Revista de la Asociación Médica Estadounidense . 167 (15): 1807–9. doi : 10.1001 / jama.1958.02990320001001 . PMID 13563181 .
- ^ Weiss RC, Crepea SB (julio de 1959). "Desarrollo de la sensibilidad a la penicilinasa después de su uso en la reacción a la penicilina". El diario de la alergia . 30 (4): 337–41. doi : 10.1016 / 0021-8707 (59) 90041-3 . PMID 13664435 .
- ^ Poder judicial, Comité del Senado del Congreso de Estados Unidos sobre el (1961). Ley de Defensa de la Competencia de la Industria de las Drogas: Audiencias ante el Subcomité de Defensa de la Competencia y Monopolio del Comité de la Judicatura, Senado de los Estados Unidos, 87º Congreso, Primera [-segunda] sesión, de conformidad con S. Res. 52 sobre S. 1552, un proyecto de ley para enmendar y complementar las leyes antimonopolio, con respecto a la fabricación y distribución de medicamentos y para otros fines . Oficina de Imprenta del Gobierno de EE. UU.
- ^ Monopolio, Comité del Senado del Congreso de los Estados Unidos en el Subcomité Judicial de Defensa de la Competencia y (1957). Precios administrados: cont] pt.25. Precios administrados en la industria farmacéutica (Antibióticos-Apéndice A). 1961. págs. 14201-15329 . Oficina de Imprenta del Gobierno de EE. UU.
- ^ Lindström EB, Nordström K (febrero de 1972). "Método automatizado para la determinación de penicilinas, cefalosporinas y penicilinasas" . Agentes antimicrobianos y quimioterapia . 1 (2): 100–6. doi : 10.1128 / aac.1.2.100 . PMC 444176 . PMID 4618452 .
- ^ "Registro Federal, volumen 62 número 229 (viernes 28 de noviembre de 1997)" . www.govinfo.gov . Consultado el 24 de diciembre de 2020 .
- ^ O'Callaghan CH, Morris A, Kirby SM, Shingler AH (abril de 1972). "Nuevo método para la detección de betalactamasas mediante el uso de un sustrato de cefalosporina cromogénica" . Agentes antimicrobianos y quimioterapia . 1 (4): 283–8. doi : 10.1128 / AAC.1.4.283 . PMC 444209 . PMID 4208895 .
- ^ Hall BG, Salipante SJ, Barlow M (julio de 2004). "Orígenes independientes del subgrupo Bl + B2 y subgrupo B3 metalo-beta-lactamasas". Revista de evolución molecular . 59 (1): 133–41. doi : 10.1007 / s00239-003-2572-9 . PMID 15383916 . S2CID 30833168 .
- ^ Lee JH, Takahashi M, Jeon JH, Kang LW, Seki M, Park KS, et al. (2019). "La actividad dual de PNGM-1 señala el origen evolutivo de las metalo- β- lactamasas de la subclase B3 : un estudio molecular y evolutivo" . Microbios e infecciones emergentes . 8 (1): 1688-1700. doi : 10.1080 / 22221751.2019.1692638 . PMC 6882493 . PMID 31749408 .
- ^ Berglund F, Johnning A, Larsson DG, Kristiansson E (enero de 2021). "Una filogenia actualizada de las metalo-β-lactamasas". La revista de quimioterapia antimicrobiana . 76 (1): 117-123. doi : 10.1093 / jac / dkaa392 . PMID 33005957 .
- ^ InterPro : IPR012338
- ^ Hall BG, Barlow M (septiembre de 2003). "Filogenias basadas en la estructura de las betalactamasas de serina". Revista de evolución molecular . 57 (3): 255–60. doi : 10.1007 / s00239-003-2473-y . PMID 14629035 . S2CID 187389 .
- ^ Hall BG, Barlow M (abril de 2004). "Evolución de las serina betalactamasas: pasado, presente y futuro". Actualizaciones sobre resistencia a los medicamentos . 7 (2): 111–23. doi : 10.1016 / j.drup.2004.02.003 . PMID 15158767 .
- ^ Barlow M, Hall BG (septiembre de 2002). "El análisis filogenético muestra que los genes de la betalactamasa OXA han estado en plásmidos durante millones de años". Revista de evolución molecular . 55 (3): 314-21. Código Bibliográfico : 2002JMolE..55..314B . doi : 10.1007 / s00239-002-2328-y . PMID 12187384 . S2CID 8679468 .
- ^ "Etimología: β-lactamasa" . Enfermedades infecciosas emergentes . 22 (9): 1689-1631. 2016. doi : 10.3201 / eid2209.ET2209 .
Otras lecturas
- Sawa T, Kooguchi K, Moriyama K (diciembre de 2020). "Diversidad molecular de β-lactamasas y carbapenemasas de espectro extendido y resistencia a los antimicrobianos" . Revista de cuidados intensivos . 8 (1): 13. doi : 10.1186 / s40560-020-0429-6 . PMID 32015881 .
- Philippon A, Slama P, Dény P, Labia R (enero de 2016). "Una clasificación basada en la estructura de la clase A β-lactamasas, una familia de enzimas ampliamente diversa" . Revisiones de microbiología clínica . 29 (1): 29–57. doi : 10.1128 / CMR.00019-15 . PMID 26511485 .
- Yoon EJ, Jeong SH (marzo de 2021). "Β-lactamasas de clase D". La revista de quimioterapia antimicrobiana . 76 (4): 836–864. doi : 10.1093 / jac / dkaa513 . PMID 33382875 .
enlaces externos
- Herramienta de genotipado de BLEE en línea (EGT)
- Secuencias de aminoácidos en línea para enzimas BLEE
- beta-lactamasas en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .
- Antibióticos betalactámicos
- EC 3.5.2
- Enzimas de estructura conocida