Dominio de proteína quinasa
| Dominio de proteína quinasa | ||||||||
|---|---|---|---|---|---|---|---|---|
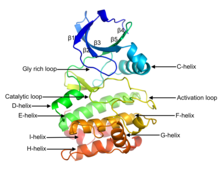
 Estructura de la subunidad catalítica de la proteína quinasa dependiente de cAMP. [1] | ||||||||
| Identificadores | ||||||||
| Símbolo | Pkinase | |||||||
| Pfam | PF00069 | |||||||
| InterPro | IPR000719 | |||||||
| INTELIGENTE | TyrKc | |||||||
| PROSITE | PDOC00100 | |||||||
| SCOP2 | 1apm / SCOPe / SUPFAM | |||||||
| Superfamilia OPM | 186 | |||||||
| CDD | cd00180 | |||||||
| Membranome | 3 | |||||||
| ||||||||
El dominio de proteína quinasa es un dominio de proteína conservado estructuralmente que contiene la función catalítica de proteína quinasas . [2] [3] [4] Las proteínas quinasas son un grupo de enzimasque mueven un grupo fosfato hacia las proteínas, en un proceso llamado fosforilación. Esto funciona como un interruptor de encendido / apagado para muchos procesos celulares, incluido el metabolismo, la transcripción, la progresión del ciclo celular, el reordenamiento citoesquelético y el movimiento celular, la apoptosis y la diferenciación. También funcionan en el desarrollo embrionario, las respuestas fisiológicas y en el sistema nervioso e inmunológico. La fosforilación anormal causa muchas enfermedades humanas, incluido el cáncer, y los medicamentos que afectan la fosforilación pueden tratar esas enfermedades. [5]
Las proteínas quinasas poseen una subunidad catalítica que transfiere el gamma fosfato de los nucleósidos trifosfatos (casi siempre ATP ) a la cadena lateral de un aminoácido en una proteína, lo que da como resultado cambios conformacionales y / o dinámicos que afectan la función de la proteína. Estas enzimas se clasifican en dos amplias clases, caracterizadas con respecto a la especificidad del sustrato: específicas de serina / treonina y específicas de tirosina . [6]
Función
La función de la proteína quinasa se ha conservado evolutivamente desde Escherichia coli hasta Homo sapiens . Las proteínas quinasas desempeñan un papel en una multitud de procesos celulares, incluida la división, proliferación, apoptosis y diferenciación. [7] La fosforilación generalmente da como resultado un cambio funcional de la proteína objetivo al cambiar la estructura, la dinámica, la actividad enzimática, la ubicación celular o la asociación con otras proteínas.
Estructura
Las subunidades catalíticas de las proteína quinasas están muy conservadas y se han determinado las estructuras de más de 270 de los aproximadamente 500 dominios de quinasas humanas, [8] lo que lleva a grandes pantallas para desarrollar inhibidores específicos de quinasas para el tratamiento de varias enfermedades. [9]
Las proteínas quinasas eucariotas [2] [3] [10] [11] son enzimas que pertenecen a una familia muy extensa de proteínas que comparten un núcleo catalítico conservado común con las proteínas quinasas serina / treonina y tirosina. El dominio consta de dos subdominios denominados dominios N- y C-terminales. El dominio N-terminal consta de cinco hebras de hoja beta y una hélice alfallamado C-hélice, y el dominio C-terminal generalmente consta de seis hélices alfa (etiquetadas como D, E, F, G, H e I). El dominio C-terminal contiene dos bucles largos, llamados bucle catalítico y bucle de activación, que son esenciales para la actividad catalítica. El bucle catalítico incluye el "motivo HRD" (para la secuencia de aminoácidos His-Arg-Asp), cuyo residuo de ácido aspártico interactúa directamente con el grupo hidroxilo del residuo de serina, treonina o tirosina diana que está fosforilado. [12]
El bucle de activación comienza con el motivo DFG (para la secuencia de aminoácidos Asp-Phe-Gly), que ayuda a unir ATP y magnesio en el sitio activo. En términos generales, el estado o conformación de la quinasa puede clasificarse como DFGin o DFGout , dependiendo de si el residuo Asp del motivo DFG está dentro o fuera del sitio activo. En la forma activa, los primeros residuos del bucle de activación adoptan una forma específica de la conformación DFGin. Algunas estructuras inactivas pueden adoptar una de varias otras conformaciones de DFGin, mientras que otras estructuras inactivas son DFGout. [13]
Ejemplos de
La siguiente es una lista de proteínas humanas que contienen el dominio de proteína quinasa: [14]
AAK1 ; AATK ; ABL1 ; ABL2 ; ACVR1 ; ACVR1B ; ACVR1C ; ACVR2A ; ACVR2B ; ACVRL1 ; AKT1 ; AKT2 ; AKT3 ; ALK ; AMHR2 ; ANKK1 ; ARAF ; AURKA ; AURKB ; AURKC ; AXL ; BLK ; BMP2K ; BMPR1A ; BMPR1B ;BMPR2 ; BMX ; BRAF ; BRSK1 ; BRSK2 ; BTK ; BUB1 ; BUB1B ; CAMK1 ; CAMK1D ; CAMK1G ; CAMK2A ; CAMK2B ; CAMK2D ; CAMK2G ; CAMK4 ; CAMKK1 ; CAMKK2 ; CAMKV ; CASK ; CDC42BPA ; CDC42BPB ; CDC42BPG ; CDC7 ; CDK1 ;CDK10 ; CDK11A ; CDK11B ; CDK12 ; CDK13 ; CDK14 ; CDK15 ; CDK16 ; CDK17 ; CDK18 ; CDK19 ; CDK2 ; CDK20 ; CDK3 ; CDK4 ; CDK5 ; CDK6 ; CDK7 ; CDK8 ; CDK9 ; CDKL1 ; CDKL2 ; CDKL3 ; CDKL4 ; CDKL5 ;CHEK1 ; CHEK2 ; CHUK ; CIT ; CLK1 ; CLK2 ; CLK3 ; CLK4 ; CSF1R ; CSK ; CSNK1A1 ; CSNK1A1L ; CSNK1D ; CSNK1E ; CSNK1G1 ; CSNK1G2 ; CSNK1G3 ; CSNK2A1 ; CSNK2A2 ; CSNK2A3 ; DAPK1 ; DAPK2 ; DAPK3 ; DCLK1 ; DCLK2 ;DCLK3 ; DDR1 ; DDR2 ; DMPK ; DSTYK ; DYRK1A ; DYRK1B ; DYRK2 ; DYRK3 ; DYRK4 ; EGFR ; EIF2AK1 ; EIF2AK2 ; EIF2AK3 ; EIF2AK4 ; EPHA1 ; EPHA10 ; EPHA2 ; EPHA3 ; EPHA4 ; EPHA5 ; EPHA6 ; EPHA7 ; EPHA8 ; EPHB1 ;EPHB2 ; EPHB3 ; EPHB4 ; EPHB6 ; ERBB2 ; ERBB3 ; ERBB4 ; ERN1 ; ERN2 ; FER ; FES ; FGFR1 ; FGFR2 ; FGFR3 ; FGFR4 ; FGR ; FLT1 ; FLT3 ; FLT4 ; FRK ; FYN ; GAK ; GRK1 ; GRK2 ; GRK3 ; GRK4 ; GRK5 ; GRK6 ; GRK7 ; GSG2 ; GSK3A ; GSK3B ; GUCY2C ; GUCY2D ; GUCY2F ; HCK ; HIPK1 ; HIPK2 ; HIPK3 ; HIPK4 ; HUNK; ICK ; IGF1R ; IKBKB ; IKBKE ; ILK ; INSR ; INSRR ; IRAK1 ; IRAK2 ; IRAK3 ; IRAK4 ; ITK ; JAK1 ; JAK2 ; JAK3 ; KALRN ; KDR ; KIT ; KSR1 ; KSR2 ; LATS1 ; LATS2 ; LCK ; LIMK1 ; LIMK2 ; LMTK2 ; LMTK3 ; LRRK1 ; LRRK2 ; LTK ; LYN ; MAK ; MAP2K1 ; MAP2K2 ; MAP2K3 ; MAP2K4 ;MAP2K5 ; MAP2K6 ; MAP2K7 ; MAP3K1 ; MAP3K10 ; MAP3K11 ; MAP3K12 ; MAP3K13 ; MAP3K14 ; MAP3K15 ; MAP3K19 ; MAP3K2 ; MAP3K20 ; MAP3K21 ; MAP3K3 ; MAP3K4 ; MAP3K5 ; MAP3K6 ; MAP3K7 ; MAP3K8 ; MAP3K9 ; MAP4K1 ; MAP4K2 ; MAP4K3 ;MAP4K4 ; MAP4K5 ; MAPK1 ; MAPK10 ; MAPK11 ; MAPK12 ; MAPK13 ; MAPK14 ; MAPK15 ; MAPK3 ; MAPK4 ; MAPK6 ; MAPK7 ; MAPK8 ; MAPK9 ; MAPKAPK2 ; MAPKAPK3 ; MAPKAPK5 ; MARK1 ; MARK2 ; MARK3 ; MARK4 ; MAST1 ; MAST2 ; MAST3 ; MAST4 ; MASTL ; MATK ; MELK ; MERTK ; MET ; MINK1 ; MKNK1 ; MKNK2 ; MLKL ; MOK ; MOS ; MST1R ; MUSK ; MYLK ; MYLK2 ; MYLK3 ; MYLK4 ; MYO3A ; MYO3B ; NEK1 ; NEK10 ; NEK11 ; NEK2 ; NEK3 ;NEK4 ; NEK5 ; NEK6 ; NEK7 ; NEK8 ; NEK9 ; NIM1K ; NLK ; NPR1 ; NPR2 ; NRBP1 ; NRBP2 ; NRK ; NTRK1 ; NTRK2 ; NTRK3 ; NUAK1 ; NUAK2 ; OBSCN ; OXSR1 ; PAK1 ; PAK2 ; PAK3 ; PAK4 ; PAK5 ; PAK6 ; PAN3 ; PASK ; PBK ; PDGFRA ; PDGFRB ; PDIK1L ; PDPK1 ; PDPK2P ; PEAK1 ; PEAK3 ; PHKG1 ; PHKG2 ; PIK3R4 ; PIM1 ; PIM2 ; PIM3 ; PINK1 ; PKDCC ; PKMYT1 ; PKN1 ; PKN2 ; PKN3 ; PLK1 ; PLK2 ; PLK3 ;PLK4 ; PLK5 ; PNCK ; POMK ; PRKAA1 ; PRKAA2 ; PRKACA ; PRKACB ; PRKACG ; PRKCA ; PRKCB ; PRKCD ; PRKCE ; PRKCG ; PRKCH ; PRKCI ; PRKCQ ; PRKCZ ; PRKD1 ; PRKD2 ; PRKD3 ; PRKG1 ; PRKG2 ; PRKX ; PRKY ;PRPF4B ; PSKH1 ; PSKH2 ; PTK2 ; PTK2B ; PTK6 ; PTK7 ; PXK ; RAF1 ; RET ; RIOK1 ; RIOK2 ; RIOK3 ; RIPK1 ; RIPK2 ; RIPK3 ; RIPK4 ; RNASEL ; ROCK1 ; ROCK2 ; ROR1 ; ROR2 ; ROS1 ; RPS6KA1 ; RPS6KA2 ;RPS6KA3 ; RPS6KA4 ; RPS6KA5 ; RPS6KA6 ; RPS6KB1 ; RPS6KB2 ; RPS6KC1 ; RPS6KL1 ; RSKR ; RYK ; SBK1 ; SBK2 ; SBK3 ; SCYL1 ; SCYL2 ; SCYL3 ; SGK1 ; SGK2 ; SGK223 ; SGK3 ; SIK1 ; SIK1B ; SIK2 ; SIK3 ; SLK ;SNRK ; SPEG ; SRC ; SRMS ; SRPK1 ; SRPK2 ; SRPK3 ; STK10 ; STK11 ; STK16 ; STK17A ; STK17B ; STK24 ; STK25 ; STK26 ; STK3 ; STK31 ; STK32A ; STK32B ; STK32C ; STK33 ; STK35 ; STK36 ; STK38 ; STK38L ;STK39 ; STK4 ; STK40 ; STKLD1 ; STRADA ; STRADB ; STYK1 ; SYK ; TAOK1 ; TAOK2 ; TAOK3 ; TBCK ; TBK1 ; TEC ; TEK ; TESK1 ; TESK2 ; TEX14 ; TGFBR1 ; TGFBR2 ; TIE1 ; TLK1 ; TLK2 ; TNIK ; TNK1 ; TNK2 ; TNNI3K ; TP53RK ; TRIB1 ; TRIB2 ; TRIB3 ; TRIO ; TSSK1B ; TSSK2 ; TSSK3 ; TSSK4 ; TSSK6 ; TTBK1 ; TTBK2 ; TTK ; TTN ; TXK ; TYK2 ; TYRO3 ; UHMK1 ; ULK1 ; ULK2 ; ULK3 ; ULK4 ; VRK1 ; VRK2 ;VRK3 ; WEE1 ; WEE2 ; WNK1 ; WNK2 ; WNK3 ; WNK4 ; SÍ1 ; ZAP70
Referencias
- ^ Knighton DR, Bell SM, Zheng J, et al. (Mayo de 1993). "2.0 Una estructura cristalina refinada de la subunidad catalítica de la proteína quinasa dependiente de AMPc complejada con un inhibidor de péptidos y detergente". Acta Crystallogr. D . 49 (Pt 3): 357–61. doi : 10.1107 / S0907444993000502 . PMID 15299526 .
- ↑ a b Hanks SK, Quinn AM (1991). "Base de datos de secuencia de dominio catalítico de proteína quinasa: identificación de características conservadas de estructura primaria y clasificación de miembros de la familia". Meth. Enzymol . Métodos en enzimología. 200 : 38–62. doi : 10.1016 / 0076-6879 (91) 00126-H . ISBN 978-0-12-182101-2. PMID 1956325 .
- ↑ a b Hanks SK, Hunter T (mayo de 1995). "Proteína quinasas 6. La superfamilia de proteína quinasa eucariota: estructura y clasificación del dominio de quinasa (catalítica)" . FASEB J . 9 (8): 576–96. doi : 10.1096 / fasebj.9.8.7768349 . PMID 7768349 . S2CID 21377422 .
- ^ Scheeff ED, Bourne PE (octubre de 2005). "Evolución estructural de la superfamilia de proteínas quinasas" . PLOS Comput. Biol . 1 (5): e49. doi : 10.1371 / journal.pcbi.0010049 . PMC 1261164 . PMID 16244704 .
- ^ Manning G, Whyte DB, Martinez R, Hunter T, Sudarsanam S (diciembre de 2002). "El complemento de proteína quinasa del genoma humano". Ciencia . 298 (5600): 1912-1934. doi : 10.1126 / science.1075762 . PMID 12471243 . S2CID 26554314 .
- ^ Hunter T, Hanks SK, Quinn AM (1988). "La familia de la proteína quinasa: características conservadas y filogenia deducida de los dominios catalíticos". Ciencia . 241 (4861): 42–51. doi : 10.1126 / science.3291115 . PMID 3291115 .
- ^ Manning G, Ploughman GD, Hunter T, Sudarsanam S (octubre de 2002). "Evolución de la señalización de la proteína quinasa de la levadura al hombre". Trends Biochem. Sci . 27 (10): 514-20. doi : 10.1016 / S0968-0004 (02) 02179-5 . PMID 12368087 .
- ^ Modi, V; Dunbrack, RL (24 de diciembre de 2019). "Una alineación de secuencia múltiple validada estructuralmente de 497 dominios de proteína quinasa humana" . Informes científicos . 9 (1): 19790. doi : 10.1038 / s41598-019-56499-4 . PMC 6930252 . PMID 31875044 .
- ^ Li B, Liu Y, Uno T, Gray N (agosto de 2004). "Creación de diversidad química para dirigirse a las proteínas quinasas" . Peine. Chem. Pantalla de alto rendimiento . 7 (5): 453–72. doi : 10.2174 / 1386207043328580 . PMID 15320712 . Archivado desde el original el 14 de abril de 2013.
- ^ Hanks SK (2003). "Análisis genómico de la superfamilia de proteína quinasa eucariota: una perspectiva" . Genome Biol . 4 (5): 111. doi : 10.1186 / gb-2003-4-5-111 . PMC 156577 . PMID 12734000 .
- ^ Hunter T (1991). "Clasificación de proteína quinasa". Meth. Enzymol . Métodos en enzimología. 200 : 3-37. doi : 10.1016 / 0076-6879 (91) 00125-G . ISBN 978-0-12-182101-2. PMID 1835513 .
- ^ Knighton DR, Zheng JH, Ten Eyck LF, Ashford VA, Xuong NH, Taylor SS, Sowadski JM (julio de 1991). "Estructura cristalina de la subunidad catalítica de la proteína quinasa dependiente de monofosfato de adenosina cíclico". Ciencia . 253 (5018): 407–14. doi : 10.1126 / science.1862342 . PMID 1862342 .
- ^ Modi, V; Dunbrack, RL (2 de abril de 2019). "Definición de una nueva nomenclatura para las estructuras de quinasas activas e inactivas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 116 (14): 6818–6827. doi : 10.1073 / pnas.1814279116 . PMC 6452665 . PMID 30867294 .
- ^ "Proteínas quinasas humanas y de ratón: clasificación e índice" . pkinfam.txt . Consorcio UniProt . Consultado el 10 de junio de 2019 .
- Dominios proteicos
- Proteínas de membrana periférica