Secuenciación de células individuales
La secuenciación de células individuales examina la información de la secuencia de células individuales con tecnologías optimizadas de secuenciación de próxima generación (NGS), lo que proporciona una mayor resolución de las diferencias celulares y una mejor comprensión de la función de una célula individual en el contexto de su microentorno. [1] Por ejemplo, en el cáncer, la secuenciación del ADN de células individuales puede brindar información sobre las mutaciones que tienen pequeñas poblaciones de células. En desarrollo, la secuenciación de los ARN expresados por células individuales puede dar una idea de la existencia y el comportamiento de diferentes tipos de células. [2]En los sistemas microbianos, una población de la misma especie puede parecer genéticamente clonal, pero la secuenciación de ARN de una sola célula o las modificaciones epigenéticas pueden revelar la variabilidad de célula a célula que puede ayudar a las poblaciones a adaptarse rápidamente para sobrevivir en entornos cambiantes. [3]
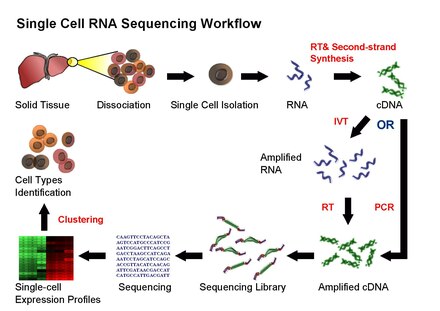
Una célula humana típica consta de aproximadamente 2 x 3300 millones de pares de bases de ADN y 600 millones de bases de ARNm. Por lo general, se utiliza una combinación de millones de células para secuenciar el ADN o el ARN utilizando métodos tradicionales como la secuenciación de Sanger o la secuenciación de Illumina . Mediante el uso de la secuenciación profunda del ADN y el ARN de una sola célula, las funciones celulares se pueden investigar de forma exhaustiva. [1] Al igual que los experimentos NGS típicos, los protocolos de secuenciación de una sola célula generalmente contienen los siguientes pasos: aislamiento de una sola célula, extracción y amplificación de ácidos nucleicos, preparación de bibliotecas de secuenciación, secuenciación y análisis bioinformático .análisis de los datos. Es más desafiante realizar la secuenciación de una sola célula en comparación con la secuenciación de células a granel. La cantidad mínima de materiales de partida de una sola célula hace que la degradación, la pérdida de muestras y la contaminación ejerzan efectos pronunciados en la calidad de los datos de secuenciación. Además, debido al nivel de picogramos de la cantidad de ácidos nucleicos utilizados, [4] a menudo se necesita una gran amplificación durante la preparación de muestras de secuenciación de una sola célula, lo que da como resultado una cobertura desigual, ruido y una cuantificación imprecisa de los datos de secuenciación.
Las mejoras técnicas recientes hacen que la secuenciación de células individuales sea una herramienta prometedora para abordar un conjunto de problemas aparentemente inaccesibles. Por ejemplo, las muestras heterogéneas, los tipos de células raros, las relaciones de linaje celular, el mosaicismo de los tejidos somáticos, los análisis de microbios que no se pueden cultivar y la evolución de la enfermedad pueden dilucidarse mediante la secuenciación de una sola célula. [5] La secuenciación de células individuales fue seleccionada como el método del año 2013 por Nature Publishing Group. [6]
La secuenciación del genoma del ADN de una sola célula implica aislar una sola célula, amplificar todo el genoma o la región de interés, construir bibliotecas de secuenciación y luego aplicar la secuenciación de ADN de próxima generación (por ejemplo , Illumina , Ion Torrent , MGI ). En los sistemas de los mamíferos, la secuenciación del ADN de una sola célula se ha aplicado ampliamente para estudiar la fisiología normal y la enfermedad. La resolución de una sola célula puede descubrir las funciones del mosaicismo genético o la heterogeneidad genética intratumoral en el desarrollo del cáncer o la respuesta al tratamiento. [7]En el contexto de los microbiomas, un genoma de un único organismo unicelular se denomina genoma amplificado único (SAG). Los avances en la secuenciación del ADN de una sola célula han permitido la recopilación de datos genómicos de especies procarióticas no cultivadas presentes en microbiomas complejos. [8] Aunque los SAG se caracterizan por una baja integridad y un sesgo significativo, los avances computacionales recientes han logrado el ensamblaje de genomas casi completos a partir de SAG compuestos. [9] Los datos obtenidos de microorganismos podrían establecer procesos para el cultivo en el futuro. [10] Algunas de las herramientas de ensamblaje del genoma que se pueden usar en la secuenciación del genoma de una sola célula incluyen: SPAdes , IDBA-UD, Cortex e HyDA. [11]