Teoría del gen dividido
La teoría del gen dividido es una teoría del origen de los intrones , secuencias largas no codificantes en genes eucariotas entre los exones . [1] [2] [3] La teoría sostiene que la aleatoriedad de las secuencias de ADN primordial solo permitiría marcos de lectura abiertos (ORF) pequeños (<600 pb ), y que las estructuras intrónicas importantes y las secuencias reguladoras se derivan de los codones de terminación . En este marco de intrones primero, la maquinaria espliceosomal y el núcleo evolucionaron debido a la necesidad de unir estos ORF (ahora "exones") en proteínas más grandes, y que los genes bacterianos sin intrones son menos ancestrales que los genes eucariotas divididos. La teoría se originó conPeriannan Senapathy .
La teoría proporciona soluciones a preguntas clave relacionadas con la arquitectura de genes divididos, incluidos genes eucariotas divididos, exones, intrones, uniones de empalme y puntos de ramificación, basándose en el origen de genes divididos a partir de secuencias genéticas aleatorias. También proporciona posibles soluciones al origen de la maquinaria espliceosomal, el límite nuclear y la célula eucariota.
Esta teoría condujo al algoritmo Shapiro-Senapathy , que proporciona la metodología para detectar los sitios de empalme, exones y genes divididos en el ADN eucariota, y que es el método principal para detectar mutaciones en el sitio de empalme en genes que causan cientos de enfermedades.
La teoría del gen dividido requiere un origen separado de todas las especies eucariotas. También requiere que los procariotas más simples evolucionen a partir de eucariotas. Esto contradice completamente el consenso científico sobre la formación de células eucariotas por endosimbiosis de bacterias. En 1994, Senapathy escribió un libro sobre este aspecto de su teoría: El nacimiento independiente de organismos.Propuso que todos los genomas eucariotas se formaran por separado en un grupo primordial. El biólogo holandés Gert Korthoff criticó la teoría planteando varios problemas que no pueden ser explicados por una teoría de los orígenes independientes. Señaló que varios eucariotas necesitan cuidados y llamó a esto el "problema de las botas", en el sentido de que incluso el eucariota inicial necesitaba el cuidado de sus padres. Korthoff señala que una gran fracción de eucariotas son parásitos. La teoría de Senapathy requeriría una coincidencia para explicar su existencia. [4] [5] La teoría de Senapathy no puede explicar la fuerte evidencia de descendencia común ( homología , código genético universal, embriología , registro fósil ). [6]
Los genes de todos los organismos, excepto las bacterias, consisten en regiones codificantes de proteínas cortas ( exones ) interrumpidas por secuencias largas ( intrones ). [1] [2] Cuando se expresa un gen, su secuencia de ADN se copia en una secuencia de "ARN primario" mediante la enzima ARN polimerasa . Luego, la maquinaria del "espliceosoma" elimina físicamente los intrones de la copia de ARN del gen mediante el proceso de empalme, dejando solo una serie de exones conectados contiguamente, que se convierte en ARN mensajero (ARNm). Este ARNm ahora es leído por el ribosoma , que produce la proteína codificada. Por tanto, aunque los intrones no se eliminan físicamente de un gen, la secuencia de un gen se lee como si los intrones no estuvieran presentes.
Los exones suelen ser cortos, con una longitud media de unas 120 bases (p. Ej., En genes humanos). Las longitudes de los intrones varían ampliamente de 10 a 500.000, pero las longitudes de los exones tienen un límite superior de aproximadamente 600 bases en la mayoría de los eucariotas. Dado que los exones codifican secuencias de proteínas, son importantes para la célula, pero constituyen sólo ~ 2% de las secuencias. Los intrones, por el contrario, constituyen el 98% de las secuencias, pero parecen tener pocas funciones cruciales, excepto las secuencias potenciadoras y reguladores del desarrollo en raras ocasiones. [7] [8]

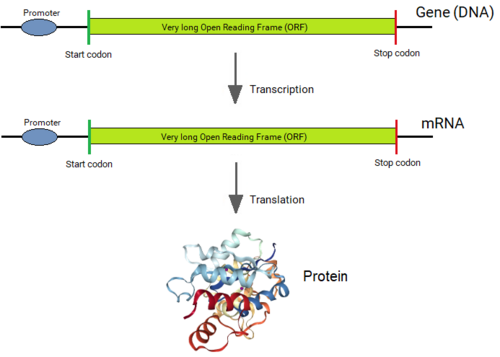
Transcripción, empalme y traducción de un gen eucariota.Un gen eucariota consta de un promotor, exones, intrones y un sitio de adición de poli-A. Se transcribe en una transcripción primaria de ARN (o pre-ARNm) por la enzima ARN polimerasa. Este ARN se somete al proceso de edición por parte del espliceosoma para la eliminación precisa de los intrones y la unión de los exones, lo que produce la molécula de ARNm. Este ARNm contiene la secuencia codificante completa sin ningún codón de parada de interrupción que es traducida por el ribosoma en la proteína codificada por el gen. En la figura, las longitudes de los intrones son cortas, pero en realidad son extremadamente largas, en promedio 20 veces más largas que los exones y, a menudo, incluso mucho más largas hasta alrededor de 500.000 bases. Los exones suelen ser cortos con un promedio de ~ 120 bases y un máximo superior de ~ 600 bases.
[1]También se muestra un ejemplo de estructura de proteína (
ID de PDB: 2VUX ) para la ribonucleótido reductasa humana, subunidad M2 B.

La agrupación de codones de terminación en una secuencia de ADN aleatoria conduce a ORF raros que son largos. La distribución de frecuencia exponencial negativa de las longitudes de ORF en una secuencia aleatoria indica que, en una secuencia lineal, cuanto más cortos son los ORF son más frecuentes y cuanto más largos son los ORF son cada vez menos frecuentes. Por tanto, existe una tendencia a que los codones de parada se agrupen en la mayoría de los lugares de una secuencia y, por tanto, los ORF más largos son más raros, incluso dentro de la longitud máxima superior de ~ 600 bases. Senapathy razonó que, los segmentos de secuencia codificante de los ORF largos disponibles podrían elegirse como exones, mientras que las secuencias intermedias con grupos de codones de terminación podrían marcarse como intrones para eliminarse de la transcripción del ARN primario, lo que conduciría a una estructura genética dividida.

La distribución exponencial negativa de longitudes de ORF en una secuencia de ADN aleatoria y en secuencias de ADN eucariotas. Senapathy descubrió que los codones de terminación ocurren con una frecuencia alta en una secuencia de ADN aleatoria, ya que existen 3 codones de terminación de 64 codones, lo que conduce a marcos de lectura abiertos cortos (ORF) con una longitud promedio de ~ 60 bases. También encontró que las longitudes de los ORF se distribuyen de manera exponencial negativa. Esta gráfica indica que la frecuencia de la longitud de ORF cero (codones de parada consecutivos que ocurren en tándem) es la más frecuente de todas las longitudes de ORF, la frecuencia de la longitud de ORF de un codón (3 bases) es la siguiente más frecuente, y así sucesivamente. La frecuencia de ORF más largos se reduce exponencialmente y alcanza una frecuencia cero alrededor de una longitud de ORF de ~ 600 bases, lo que significa que no se producen ORF de más de 600 bases.
[1] Sorprendentemente, el gráfico de las secuencias de ADN eucariotas era casi exactamente el mismo que el de las secuencias de ADN aleatorias.

Corroboración de la teoría del gen dividido mediante secuencias reales de ADN de genes humanos. La teoría del gen dividido predice que los tres codones de parada deberían estar presentes a una frecuencia alta en cada uno de los tres marcos de lectura (RF), lo que conduciría a marcos de lectura abiertos (ORF) cortos. También predice que: a) se producirían exones dentro de estos ORF cortos en los tres RF; b) los intrones serían largos, y que c) las longitudes de los exones se limitarían a las longitudes de ORF. Estas predicciones son precisamente ciertas en las secuencias de ADN de la mayoría de los genes eucariotas. Dos genes de ejemplo (
FLJ35894 y
ADCY1) del genoma humano. Todos los exones de cada gen son cortos y la mayoría de los intrones son largos. En cada gen, los exones (cuadros amarillos cortos) se limitan a ORF cortos que ocurrieron en la secuencia de ADN. Además, los codones de terminación se encuentran en los extremos de los exones, que en realidad son parte de las secuencias de unión de corte y empalme.

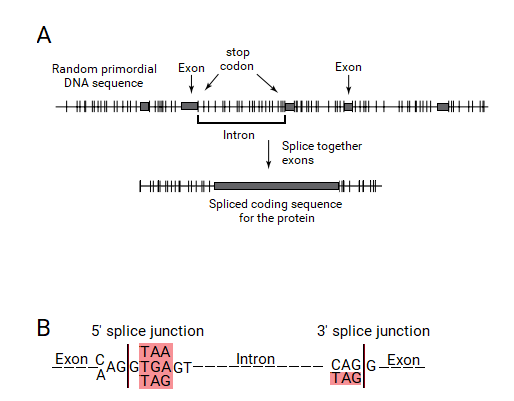
Origen de las secuencias de unión de corte y empalme de los codones de terminación. [1] [2] [3](A) La maquinaria molecular que eligió los exones de un gen dividido de una secuencia de ADN primordial aleatoria debería ser capaz de buscar codones de terminación (marcas de verificación) para identificar regiones sin codones de terminación (en la copia de ARN primaria, no mostrada), que son los ORF. Al hacerlo, el primer codón de terminación encontrado se marcará como el inicio del intrón. Este proceso conducirá a la presencia de un codón de parada al comienzo de los intrones. A veces, se elige todo un marco de lectura abierto para que sea un exón, por lo que el final del intrón anterior tendrá un codón de parada. (B) El inicio y el final del intrón son partes de las "secuencias de unión de empalme", que señalan el punto exacto de empalme a la maquinaria de empalme. Los codones de parada se muestran con fondo rojo.

Los codones de parada se presentan como partes clave de todos los elementos genéticos importantes en los genes eucariotas. Los elementos genéticos clave de los genes eucariotas son los promotores, las señales de unión de empalme del donante y el aceptor, las señales de lariat (punto de ramificación) y los sitios de adición de poli-A. El componente central de cada uno de estos elementos genéticos es un codón de terminación.

Origen de genes bacterianos a partir de genes divididos. Los genes divididos de eucariotas modernos con exones cortos (longitud promedio de 120 bases y un máximo de ~ 600 bases) interrumpidos por intrones largos son extremadamente probables en secuencias de ADN aleatorias debido a las razones descritas en la sección
Origen de los intrones y la estructura del gen dividido. , sobre. En contraste, los genes bacterianos de codificación larga contigua (que pueden ser tan largos y 10,000 bases, y más largos hasta 90,000 bases) sin intrones son prácticamente imposibles de producir en secuencias aleatorias. Por lo tanto, la única forma en que los genes bacterianos podían originarse era eliminar los intrones de los genes divididos que ocurrían en secuencias de ADN aleatorias y producir genes codificantes contiguos. La proteína de ejemplo que se muestra con su estructura 3D es de la base de datos PDB (
ID: 1UNF).