Dispersión biológica de ángulo pequeño
La dispersión biológica de ángulo pequeño es un método de dispersión de ángulo pequeño para el análisis estructural de materiales biológicos. La dispersión de ángulo pequeño se utiliza para estudiar la estructura de una variedad de objetos, como soluciones de macromoléculas biológicas, nanocompuestos, aleaciones y polímeros sintéticos. [1] La dispersión de rayos X de ángulo pequeño ( SAXS ) y la dispersión de neutrones de ángulo pequeño ( SANS ) son las dos técnicas complementarias conocidas conjuntamente como dispersión de ángulo pequeño (SAS). SAS es un método análogo a la difracción de rayos X y neutrones , la dispersión de rayos X de gran angular , así como a la dispersión de luz estática .. A diferencia de otros métodos de dispersión de neutrones y rayos X, SAS proporciona información sobre los tamaños y formas de las partículas cristalinas y no cristalinas. Cuando se utiliza para estudiar materiales biológicos, que muy a menudo se encuentran en solución acuosa, el patrón de dispersión se promedia por orientación. [2] [3]
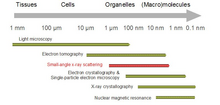
Los patrones SAS se recopilan en ángulos pequeños de unos pocos grados. SAS es capaz de entregar información estructural en el rango de resolución entre 1 y 25 nm , y de distancias repetidas en sistemas parcialmente ordenados de hasta 150 nm de tamaño. La dispersión de ángulo ultrapequeño (USAS) puede resolver dimensiones incluso mayores. La dispersión de ángulo pequeño de incidencia rasante (GISAS) es una técnica poderosa para el estudio de capas de moléculas biológicas en superficies.
En aplicaciones biológicas, SAS se utiliza para determinar la estructura de una partícula en términos de tamaño y forma de partícula promedio. También se puede obtener información sobre la relación superficie - volumen . Normalmente, las macromoléculas biológicas se dispersan en un líquido. El método es preciso, en su mayoría no destructivo y generalmente requiere solo un mínimo de preparación de la muestra. Sin embargo, las moléculas biológicas siempre son susceptibles al daño por radiación .
En comparación con otros métodos de determinación de estructuras, como la RMN en solución o la cristalografía de rayos X , SAS permite superar algunas restricciones. Por ejemplo, la RMN en solución se limita al tamaño de la proteína, mientras que la SAS se puede utilizar para moléculas pequeñas, así como para grandes ensamblajes multimoleculares. La RMN de estado sólido sigue siendo una herramienta indispensable para determinar la información del nivel atómico de macromoléculas de más de 40 kDa o muestras no cristalinas como las fibrillas de amiloide. La determinación de la estructura por cristalografía de rayos X puede tardar varias semanas o incluso años, mientras que las mediciones SAS tardan días. SAS también se puede acoplar a otras técnicas analíticas como la cromatografía de exclusión por tamaño para estudiar muestras heterogéneas. [4]Sin embargo, con SAS no es posible medir las posiciones de los átomos dentro de la molécula.
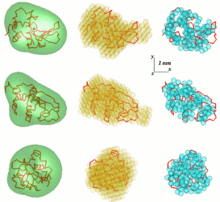
Conceptualmente, los experimentos de dispersión de ángulo pequeño son simples: la muestra se expone a rayos X o neutrones y un detector registra la radiación dispersa. Como las mediciones de SAS se realizan muy cerca del haz primario ("ángulos pequeños"), la técnica necesita un haz de rayos X o de neutrones altamente colimado o enfocado . La dispersión biológica de rayos X de ángulo pequeño a menudo se realiza en fuentes de radiación de sincrotrón , porque las moléculas biológicas normalmente se dispersan débilmente y las soluciones medidas están diluidas . El método biológico SAXS se beneficia de la alta intensidad de los haces de fotones de rayos X proporcionados por los anillos de almacenamiento de sincrotrón. La curva de dispersión de rayos X o de neutrones ( intensidad versus ángulo de dispersión ) se utiliza para crear un modelo de baja resolución de una proteína, que se muestra aquí en la imagen de la derecha. Se pueden utilizar además los datos de dispersión de rayos X o de neutrones y ajustar dominios separados (estructuras de rayos X o RMN ) en el "envoltorio SAXS".