En filogenética , apomorfia y sinapomorfia [2] se refieren a caracteres derivados de un clado : caracteres o rasgos que se derivan de caracteres ancestrales a lo largo de la historia evolutiva. [3] Una apomorfia es un carácter que es diferente de la forma encontrada en un antepasado, es decir, una innovación, que distingue al clado de otros clados. Una sinapomorfia es una apomorfia compartida que distingue a un clado de otros organismos. [1] [4] En otras palabras, es una apomorfia compartida por miembros de un monofiléticogrupo, y por lo tanto se supone que está presente en su antepasado común más reciente .
Descripción [ editar ]
Una apomorfia es un carácter que es diferente de la forma encontrada en un antepasado, es decir, una innovación, que distingue al clado (" apo– ") de otros clados. Una sinapomorfia es una apomorfia compartida (" syn ") que distingue a un clado de otros organismos. [1] [4] En otras palabras, es una apomorfia compartida por miembros de un grupo monofilético y, por lo tanto, se asume que está presente en su ancestro común más reciente .
Ejemplo [ editar ]
En la mayoría de los grupos de mamíferos, la columna vertebral está muy conservada, con el mismo número de vértebras que se encuentran en el cuello de una jirafa, por ejemplo, que en los mamíferos con cuellos más cortos. Sin embargo, en el clado Afrotheria , que incluye musarañas elefante , topos dorados y elefantes , hay un aumento en el número de vértebras toracolumbares. Se trata de una sinapomorfia del clado: una característica compartida que se considera derivada de un ancestro común. [5]
Etimología [ editar ]
La palabra sinapomorfia, acuñada por el entomólogo alemán Willi Hennig, se deriva de las palabras griegas σύν , syn = compartido; ἀπό , apo = lejos de; y μορφή , morphe = forma.
Similitudes filogenéticas [ editar ]
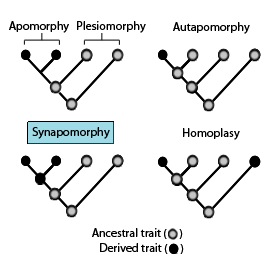
Estos términos filogenéticos se utilizan para describir diferentes patrones de estados de carácter o rasgo ancestrales y derivados como se indica en el diagrama anterior en asociación con sinapomorfias. [6] [7]
- Symplesiomorphy : un rasgo ancestral compartido por dos o más taxones.
- Plesiomorfia - una simmplesiomorfia discutida en referencia a un estado más derivado.
- Pseudoplesiomorfia: es un rasgo que no puede identificarse ni como una plesiomorfia ni como una apomorfia que sea una reversión. [8]
- Reversión: es la pérdida de un rasgo derivado presente en el antepasado y el restablecimiento de un rasgo plesiomórfico.
- Convergencia: evolución independiente de un rasgo similar en dos o más taxones.
- Apomorfia : un rasgo derivado. La apomorfia compartida por dos o más taxones y heredada de un ancestro común es la sinapomorfia. La apomorfia exclusiva de un taxón determinado es la autapomorfia. [9] [10] [11] [12]
- Sinapomorfia / Homología : un rasgo derivado que se encuentra en algunos o todos los grupos terminales de un clado y se hereda de un ancestro común, para el cual era una autapomorfia (es decir, no está presente en su ancestro inmediato).
- Sinapomorfia subyacente : una sinapomorfia que se ha vuelto a perder en muchos miembros del clado. Si se pierde en todos menos uno, puede ser difícil distinguirlo de una autapomorfia.
- Autapomorphy : un rasgo derivado distintivo que es exclusivo de un taxón o grupo determinado. [13]
- La homoplasia en la sistemática biológica es cuando un rasgo se ha ganado o perdido de forma independiente en linajes separados durante la evolución. Esta evolución convergente lleva a que las especies compartan de forma independiente un rasgo que es diferente del rasgo que se infiere que estuvo presente en su ancestro común. [14] [15] [16]
- Rasgo derivado de la homoplasia paralela presente en dos grupos o especies sin un ancestro común debido a la evolución convergente . [17]
- Homoplasia inversa : rasgo presente en un antepasado pero no en descendientes directos que reaparece en descendientes posteriores. [18]
- La hemiplasia es el caso en el que un carácter que parece homoplástico dado el árbol de la especie en realidad tiene un origen único en el árbol genético asociado. [19] [20] La hemiplasia refleja la discordancia genética entre árboles y especies debido a la coalescencia de múltiples especies .
Análisis [ editar ]
Un nuevo método para medir las características filogenéticas es el uso del análisis de sinapomorfia aparente relativa ( RASA ). El objetivo del análisis es determinar si una característica dada es común entre taxones como resultado de ancestros compartidos o del proceso de convergencia . [21] Este método permite varias ventajas como la eficiencia computacional y también administra una medida imparcial y confiable de la señal filogenética. [22]
El concepto de sinapomorfia es relativo a un clado dado en el árbol de la vida. Lo que cuenta como sinapomorfia para un clado bien puede ser un carácter primitivo o plesiomorfia en un clado menos inclusivo o anidado. Por ejemplo, la presencia de glándulas mamarias es una sinapomorfia para los mamíferos en relación con los tetrápodos, pero es una sinapomorfia para los mamíferos entre sí, por ejemplo, roedores y primates. Por tanto, el concepto también puede entenderse en términos de "un carácter más nuevo que" ( autapomorfía ) y "un carácter más antiguo que" ( plesiomorfia) la apomorfia: las glándulas mamarias son evolutivamente más nuevas que la columna vertebral, por lo que las glándulas mamarias son una autapomorfia si la columna vertebral es una apomorfia, pero si las glándulas mamarias son la apomorfia que se considera, la columna vertebral es una plesiomorfia.
Comprensión de cladograma [ editar ]
Los cladogramas son diagramas que representan relaciones evolutivas dentro de grupos de taxones. Estas ilustraciones son un dispositivo predictivo preciso en la genética moderna. Por lo general, se representan en forma de árbol o escalera. Las sinapomorfias luego crean evidencia de relaciones históricas y su estructura jerárquica asociada. Evolutivamente, una sinapomorfia es el marcador del ancestro común más reciente del grupo monofilético que consiste en un conjunto de taxones en un cladograma. [23]
Referencias [ editar ]
- ^ a b c Página de Roderick DM; Edward C. Holmes (14 de julio de 2009). Evolución molecular: un enfoque filogenético . John Wiley e hijos. ISBN 978-1-4443-1336-9.
- ^ Currie PJ, Padia K (1997). Enciclopedia de dinosaurios . Elsevier. pag. 543. ISBN 978-0-08-049474-6.
- ^ Biología de la enciclopedia concisa . Tubinga, DEU: Walter de Gruyter. 1996. p. 366 .
- ↑ a b Barton N, Briggs D, Eisen J, Goldstein D, Patel N (2007). "Reconstrucción filogenética" . Evolución . Prensa de laboratorio Cold Spring Harbor.
- ↑ Sánchez ‐ Villagra, Marcelo R .; Narita, Yuichi; Kuratani, Shigeru (1 de marzo de 2007). "Número vertebral toracolumbar: la primera sinapomorfia esquelética para mamíferos afrotherianos". Sistemática y Biodiversidad . 5 (1): 1–7. doi : 10.1017 / s1477200006002258 . ISSN 1477-2000 . S2CID 85675984 .
- ^ Página de Roderick DM; Edward C. Holmes (14 de julio de 2009). Evolución molecular: un enfoque filogenético . John Wiley e hijos. ISBN 978-1-4443-1336-9 .
- ^ Calow PP (2009). Enciclopedia de Ecología y Gestión Ambiental . John Wiley e hijos. ISBN 978-1-4443-1324-6. OCLC 1039167559 .
- ^ Williams D, Schmitt M, Wheeler Q (julio de 2016). El futuro de la sistemática filogenética: el legado de Willi Hennig . Prensa de la Universidad de Cambridge. ISBN 978-1-107-11764-8.
- ^ Simpson MG (9 de agosto de 2011). Sistemática de plantas . Elsevier . Amsterdam: Elsevier. ISBN 9780080514048.
- ^ Russell PJ, Hertz PE, McMillan B (2013). Biología: la ciencia dinámica . Aprendizaje Cengage. ISBN 978-1-285-41534-5.
- ^ Lipscomb D (1998). "Conceptos básicos del análisis cladístico" (PDF) . Washington DC: Universidad George Washington.
- ↑ Choudhuri S (9 de mayo de 2014). Bioinformática para principiantes: genes, genomas, evolución molecular, bases de datos y herramientas analíticas (1ª ed.). Prensa académica. pag. 51. ISBN 978-0-12-410471-6. OCLC 950546876 .
- ^ Appel, Ron D .; Feytmans, Ernest. Bioinformática: una perspectiva suiza. "Capítulo 3: Introducción a la filogenética y sus aspectos moleculares". World Scientific Publishing Company, 1ª edición. 2009.
- ^ Gauger A (17 de abril de 2012). "¡La similitud ocurre! El problema de la homoplasia" . Evolution Today y Science News .
- ^ Sanderson MJ, Hufford L (21 de octubre de 1996). Homoplasia: la recurrencia de la similitud en la evolución . Elsevier. ISBN 978-0-08-053411-4. OCLC 173520205 .
- ^ Brandley MC, Warren DL, Leaché AD, McGuire JA (abril de 2009). "Soporte de homoplastia y clado" . Biología sistemática . 58 (2): 184–98. doi : 10.1093 / sysbio / syp019 . PMID 20525577 .
- ^ Archie JW (septiembre de 1989). "Razones de exceso de homoplasia: nuevos índices para medir los niveles de homoplasia en la sistemática filogenética y una crítica del índice de coherencia". Biología sistemática . 38 (1): 253–269. doi : 10.2307 / 2992286 . JSTOR 2992286 .
- ^ Wake DB, Wake MH, Specht CD (febrero de 2011). "Homoplasia: de la detección del patrón a la determinación del proceso y mecanismo de evolución". Ciencia . 331 (6020): 1032–5. doi : 10.1126 / science.1188545 . PMID 21350170 . S2CID 26845473 . Resumen de Lay - Science Daily .
- ^ Avise JC, Robinson TJ (junio de 2008). "Hemiplasia: un nuevo término en el léxico de la filogenia" . Biología sistemática . 57 (3): 503–7. doi : 10.1080 / 10635150802164587 . PMID 18570042 .
- ^ Copetti D, Búrquez A, Bustamante E, Charboneau JL, Childs KL, Eguiarte LE, Lee S, Liu TL, McMahon MM, Whiteman NK, Wing RA, Wojciechowski MF, Sanderson MJ (noviembre de 2017). "Amplia discordancia del árbol de genes y hemiplasia dieron forma a los genomas de cactus columnares de América del Norte" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 114 (45): 12003–12008. doi : 10.1073 / pnas.1706367114 . PMC 5692538 . PMID 29078296 .
- ^ Lyons-Weiler J, Hoelzer GA, Tausch RJ (julio de 1996). "Análisis de sinapomorfia aparente relativa (RASA). I: La medición estadística de la señal filogenética" . Biología Molecular y Evolución . 13 (6): 749–57. doi : 10.1093 / oxfordjournals.molbev.a025635 . PMID 8754211 .
- ^ Simmons MP, Randle CP, Freudenstein JV, Wenzel JW (enero de 2002). "Limitaciones del análisis de sinapomorfia aparente relativa (RASA) para medir la señal filogenética" . Biología Molecular y Evolución . 19 (1): 14-23. doi : 10.1093 / oxfordjournals.molbev.a003978 . PMID 11752186 .
- ^ Novick LR, Catley KM. Comprensión de las filogenias en biología: la influencia de un principio perceptual Gestalt. J Exp Psychol Appl. 2007; 13: 197–223.
Enlaces externos [ editar ]
- Cladística , Berkeley