Estudios genéticos en cingaleses
Los estudios genéticos sobre los cingaleses son parte de la genética de poblaciones que investiga los orígenes de la población cingalesa .
Todos los estudios coinciden en que existe una relación significativa entre los cingaleses, los odia y los bengalíes y los tamiles del sur de la India y que existe una relación genética significativa entre los tamiles de Sri Lanka y los cingaleses, ya que están más cerca entre sí que otras poblaciones del sur de Asia. Esto también está respaldado por un estudio de distancia genética, que mostró bajas diferencias en la distancia genética entre los voluntarios cingaleses y bengalíes , odia , tamil y keralita . [1]
Según un estudio publicado en 2021 utilizando 16 marcadores de repetición en tándem cortos (STR) del cromosoma X, existe una historia demográfica sesgada por el sexo entre las etnias de Sri Lanka, donde los cingaleses y los moros están estrechamente relacionados entre sí. [2] No se detectó una subdivisión genética entre los tamiles cingaleses, moros y de Sri Lanka, mientras que los tamiles indios (del interior del país) tenían una diferencia sutil pero estadísticamente significativa. La estrecha relación observada entre moros y cingaleses puede explicarse por los lazos matrimoniales que establecieron los varones moros con las mujeres cingalesas durante su asentamiento original en Sri Lanka. Además, el filograma generado para los cuatro grupos étnicos principales de Sri Lanka sugería un origen indio para los moros en comparación con el origen árabe especulado por algunos.
Un estudio de 2017 realizado por Fumihiko Takeuchi, Tomohiro Katsuya, Ryosuke Kimura y Norihiro Kato mostró que la principal población ancestral que contribuyó a los cingaleses fue el componente "del sur de Asia" con una contribución significativa del componente de "sur de Asia y Asia central" y más pequeños. contribuciones de los componentes "Asia central", "Asia sudoriental", "Asia sudoriental y Asia oriental", "Asia oriental" y "japonés". [3]
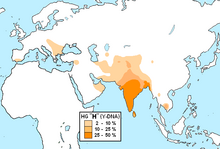
El pueblo cingalés y otros grupos étnicos del sur de Asia son de ascendencia principalmente indígena del sur de Asia (AASI). Los indígenas del sur de Asia (AASI) forman su propio linaje genético, no estrechamente relacionado con las poblaciones fuera del sur de Asia. [4]
Las AASI se originaron en el sur de Asia y fueron aisladas genéticamente de otras poblaciones más de 45.000 años antes de Cristo . La ascendencia indígena del sur de Asia (AASI) forma la ascendencia principal de los asiáticos del sur modernos (entre el 50% y el 70%), junto a los componentes recientes de Eurasia occidental y Eurasia oriental. También se detectó un flujo genético similar al AASI hacia los aborígenes australianos (hasta un 30%) y respalda aún más las olas de migración del sur de Asia a Oceanía. El pueblo Paniya es, junto a los Irula y Soliga, el mejor representante de la ascendencia indígena del sur de Asia. [4]
Relación con los bengalíes
Un análisis del polimorfismo Alu de cingaleses de Colombo realizado por el Dr. Sarabjit Mastanain en 2007 utilizando tamil, bengalí, gujarati ( Patel ) y punjabi como poblaciones parentales encontró diferentes proporciones de contribución genética: [5]
| Método estadístico | bengalí | Tamil | Noroeste |
|---|---|---|---|
| Punto estimado | 57,49% | 42,5% | - |
| Método de máxima verosimilitud | 88,07% | - | - |
| Utilizando tamil, bengalí y noroeste como población parental | 50-66% | 11-30% | 20-23% |
Un análisis de distancia genética realizado por el Dr. Robet Kirk también concluyó que los cingaleses modernos están más estrechamente relacionados con los bengalíes. [1]
Esto se ve corroborado por un estudio VNTR , que encontró que el 70-82% de los genes cingaleses se originan a partir de una mezcla bengalí: [6]
| Población parental | bengalí | Tamil | Gujarati | punjabi |
|---|---|---|---|---|
| Utilizando tamil y bengalí como población parental | 70,03% | 29,97% | - | |
| Utilizando tamil, bengalí y gujarati como población parental | 71,82% | 16,38% | 11,82% | |
| Utilizando bengalí, gujarati y punjabi como población parental | 82,09% | - | 15,39% | 2,52% |
La frecuencia del alelo D1S80 (un alelo popular para la toma de huellas genéticas) también es similar entre los cingaleses y los bengalíes, lo que sugiere que los dos grupos están estrechamente relacionados. [7] Los cingaleses también tienen frecuencias similares del alelo MTHFR 677T (13%) a las de Bengalis Occidental (17%). [8] [9]
Una prueba para haplogrupos de ADN del cromosoma Y realizada por el Dr. Toomas Kivisild en Sinhalese de Sri Lanka ha demostrado que el 23% de los sujetos eran positivos para R1a1a (R-SRY1532). [10] También en la misma prueba, el 24,1% de los sujetos fueron R2 positivos como subclades del haplogrupo P (92R7). [10] El haplogrupo R2 también se encuentra en un porcentaje considerable entre los bengalíes de la India. El tamaño de la muestra utilizada fue de 87 sujetos.
Un estudio en 2007 encontró frecuencias similares del alelo HLA-A * 02 en cingaleses (7,4%) y sujetos del norte de India (6,7%). HLA-A * 02 es un alelo raro que tiene una frecuencia relativamente alta en las poblaciones del norte de la India y se considera un alelo nuevo entre la población del norte de la India. Esto sugiere un posible origen de los cingaleses en el norte de la India. [11]
Relación con los tamiles
Otro estudio de GK Kshatriya realizado en 1995 para evaluar las 'afinidades genéticas de las poblaciones de Sri Lanka' encontró una gran contribución genética de los tamiles del sur de la India, así como de las poblaciones bengalí y vedda. El estudio se llevó a cabo utilizando cingaleses de regiones donde las interacciones cingaleses tamil eran métodos más antiguos y más altos en comparación con otros estudios modernos y precisos. [12]
| Población parental | Tamil | bengalí | Vedda |
|---|---|---|---|
| Utilizando Tamil, Bengali y Vedda como población parental | 69,86% | 25,41% | 4,73% |
El hallazgo del Dr. Sarabjit Mastanain afirma que la correlación cofenética fue de 0.8956 e indica a los cingaleses y tamil como población nativa. Además, se refleja en la distancia genética entre las cinco poblaciones de Sri Lanka según se indica a continuación vector propio terreno de la matriz-R . [13]
| Distancia genética |
| |||||||||||||||||||||||||||
| ( 5 poblaciones de Sri Lanka ) |
Relación con otros grupos étnicos en Sri Lanka
Un estudio que analizó la variación genética del gen FUT2 en la población cingalesa y tamil de Sri Lanka, encontró antecedentes genéticos similares para ambos grupos étnicos, con poco flujo genético de otros grupos poblacionales asiáticos vecinos. [14] Los estudios tampoco han encontrado diferencias significativas con respecto al grupo sanguíneo , los marcadores genéticos sanguíneos y el polimorfismo de un solo nucleótido entre los cingaleses y otros grupos étnicos en Sri Lanka. [15] [16] [17] Otro estudio también ha encontrado "ninguna variación genética significativa entre los principales grupos étnicos de Sri Lanka". [18] Esto está respaldado por un estudio que encontró frecuencias de alelos muy similaresMTHFR 677T , F2 20210A y F5 1691A en las poblaciones tamil del sur de la India, cingaleses, tamil de Sri Lanka y moros. [9]
Un estudio genético realizado en 2015 por Lian dang et. sobre el origen de los malayos y otras poblaciones de Sri Lanka que involucran a 200 personas cingalesas , 103 personas tamiles de origen Sri Lanka, 200 personas tamiles de origen indio y 35 personas burguesas calcularon la composición genética promedio de los individuos de cada población, [19] que muestra cantidad sustancialmente mayor de ascendencia de Asia Central y baja ascendencia de Asia del Sur entre los cingaleses en comparación con ambos grupos tamiles.
Relación con los asiáticos orientales y sudorientales
Los estudios genéticos muestran que los cingaleses han recibido cierto flujo genético de poblaciones vecinas en el este de Asia y el sudeste asiático , como los pueblos tibeto-birmanos y austro-asiáticos étnicamente diversos y dispares , [20] lo cual se debe a sus estrechos vínculos genéticos al noreste de la India. [21] [22] [23] Un estudio de 1985 realizado por Roychoudhury AK y Nei M, indicó que los valores de la distancia genética mostraron que los cingaleses estaban un poco más cerca de las poblaciones mongoloides debido al intercambio de genes en el pasado. [24] [25]En lo que respecta a las comparaciones de la morfología de la raíz y el conducto de los molares mandibulares de Sri Lanka, se demostró que estaban más lejos de las poblaciones mongoloides. [26] Entre los haplogrupos encuentran en Asia Oriental poblaciones, una menor frecuencia de Asia oriental ADNmt haplogrupo , G se ha encontrado entre las poblaciones de Sri Lanka de forma paralela haplogrupo D en conjunto con el principal haplogrupo ADN mitocondrial de los grupos étnicos de Sri Lanka, el haplogrupo M . [27] En lo que respecta al ADN-Y , el haplogrupo C-M130 se encuentra en frecuencias bajas a moderadas en Sri Lanka. [28]
Los marcadores genéticos de inmunoglobulina entre los cingaleses muestran altas frecuencias de afb1b3 que tiene su origen en las provincias de Yunnan y Guangxi en el sur de China. [29] También se encuentra en las frecuencias altas entre Odias, determinada de Nepal y el noreste de la India, el sur de china Han, el sudeste de Asia y ciertos austronesios poblaciones de las islas del Pacífico . [29] Con una frecuencia más baja, ab3st también se encuentra entre los cingaleses y generalmente se encuentra con frecuencias más altas entre las poblaciones de chinos Han del norte, tibetanos, mongoles, coreanos y japoneses. [29] La transferrinaEl alelo TF * Dchi, que es común entre las poblaciones de Asia oriental y los nativos americanos, también se encuentra entre los cingaleses. [24] HumDN1 * 4 y HumDN1 * 5 son los genes DNasa I predominantes entre los cingaleses y también son los genes predominantes entre los grupos étnicos del sur de China y el pueblo Tamang de Nepal. [30] Un estudio de 1988 realizado por N. Saha mostró que las frecuencias altas de GC * 1F y bajas de GC * 1S entre los cingaleses son comparables a las de los chinos, japoneses, coreanos, tailandeses, malayos, vietnamitas, laosianos y tibetanos. [31] Un estudio de 1998 realizado por DE Hawkey mostró que la morfología dental de los cingaleses está estrechamente relacionada con la de las poblaciones austroasiáticas del este y noreste de la India. [20] La hemoglobina E, una variante de la hemoglobina normal, que se originó y prevalece entre las poblaciones del sudeste asiático , también es común entre los cingaleses y puede alcanzar hasta el 40% en Sri Lanka. [32]
Relación con las poblaciones mundiales
En un estudio [2]que comparó las cuatro principales etnias de Sri Lanka (cingaleses, SL Tamil, indios tamil, moros) con otras 14 poblaciones mundiales (Bhil India, Bangladesh, Malasia, Tailandia, China, Japón, Taiwán, Alemania, Italia, Suecia, Dinamarca, Norte de Portugal , Somalia y Costa de Marfil) con ocho marcadores STR basados en el cromosoma X utilizando un diagrama de escala multidimensional (diagrama MDS), los habitantes de Sri Lanka se agruparon no solo con los del sudeste asiático como los indios y bangladesíes, sino también con los europeos. Los tamiles indios se colocaron hacia la periferia de este grupo principal, mientras que los asiáticos del sudeste, el este de Asia y los africanos se ubicaron en un lugar distante, fuera del grupo principal. Se cree que esta presentación se alinea bien con las afirmaciones históricas de los movimientos de población en Eurasia, donde se cree que los cingaleses descienden de los indo-arios.que partió de las fronteras del Mar Caspio y el Mar Negro hacia Europa y el sur de Asia, a principios del tercer milenio antes de Cristo.
Pigmentación de la piel
En 2008, un estudio analizó el polimorfismo SLC24A5, que representa el 25-40% de la diferencia en el color de la piel entre europeos y africanos y hasta un 30% de la variación del color de la piel en los sudasiáticos. [21] El estudio encontró que el rs1426654 SNP de SLC24A5 , que se fija en las poblaciones europeas [33] y se encuentra más comúnmente en personas de piel clara que en personas de piel oscura (49% en comparación con 10%), tiene una frecuencia de alrededor del 55% en los cingaleses y alrededor del 25% en los tamiles de Sri Lanka. [21] Este alelo podría haber surgido en los cingaleses debido a una mayor migración desde el norte de la India o fuertes factores de selección.
Referencias
- ↑ a b Kirk RL (julio de 1976). "La leyenda del príncipe Vijaya - un estudio de los orígenes cingaleses". Revista Estadounidense de Antropología Física . 45 (1): 91–99. doi : 10.1002 / ajpa.1330450112 .
- ↑ a b Perera N, Galhena G, Ranawaka G (junio de 2021). "Polimorfismos genéticos basados en STR del cromosoma X e historia demográfica de las etnias de Sri Lanka y su relación con las poblaciones globales" . Informes científicos . 11 (1): 12748. doi : 10.1038 / s41598-021-92314-9 . PMC 8211843 . PMID 34140598 .
- ^ Takeuchi F, Katsuya T, Kimura R, Nabika T, Isomura M, Ohkubo T, et al. (1 de noviembre de 2017). "La estructura genética a escala fina y la evolución de la población japonesa" . PLOS One . 12 (11): e0185487. Código bibliográfico : 2017PLoSO..1285487T . doi : 10.1371 / journal.pone.0185487 . PMC 5665431 . PMID 29091727 .
- ^ a b Yelmen B, Mondal M, Marnetto D, Pathak AK, Montinaro F, Gallego Romero I, et al. (Agosto de 2019). "Los análisis específicos de ascendencia revelan historias demográficas diferenciales y presiones selectivas opuestas en las poblaciones modernas del sur de Asia" . Biología Molecular y Evolución . 36 (8): 1628–1642. doi : 10.1093 / molbev / msz037 . PMC 6657728 . PMID 30952160 .
- ^ Mastana S (2007). "Antropología molecular: población y aplicaciones genéticas forenses" (PDF) . Especial Antropólogo . 3 : 373–383.
- ^ Papiha SS, Mastana SS, Purandare CA, Jayasekara R, Chakraborty R (octubre de 1996). "Estudio genético de la población de tres loci VNTR (D2S44, D7S22 y D12S11) en cinco poblaciones definidas étnicamente del subcontinente indio". Biología humana . 68 (5): 819–35. JSTOR 41465520 . PMID 8908803 .
- ^ Surinder Singh Papiha (1999). Diversidad genómica: aplicaciones en genética de poblaciones humanas. Londres: Springer. 7.
- ^ Mukhopadhyay K, Dutta S, Das Bhomik A (enero de 2007). "Polimorfismos del gen MTHFR analizados en población de Kolkata, Bengala Occidental" . Revista India de Genética Humana . 13 (1): 38. doi : 10.4103 / 0971-6866.32035 . PMC 3168154 . PMID 21957342 .
- ↑ a b Dissanayake VH, Weerasekera LY, Gammulla CG, Jayasekara RW (octubre de 2009). "Prevalencia de polimorfismos trombofílicos genéticos en la población de Sri Lanka: implicaciones para el diseño de estudios de asociación y los servicios de pruebas genéticas clínicas". Patología experimental y molecular . 87 (2): 159–62. doi : 10.1016 / j.yexmp.2009.07.002 . PMID 19591822 .
- ^ a b Kivisild T, Rootsi S, Metspalu M, Metspalu E, Parik J, Kaldma K, et al. (2003). "La genética del lenguaje y la agricultura difundida en la India" (PDF) . En Bellwood P, Renfrew C (eds.). Examinar la hipótesis de la dispersión agrícola / lingüística . Cambridge, Reino Unido: McDonald Institute for Archaeological Research. págs. 215-222.
- ^ Malavige GN, Rostron T, Seneviratne SL, Fernando S, Sivayogan S, Wijewickrama A, Ogg GS (octubre de 2007). "El análisis de HLA del cingalés de Sri Lanka predice el origen del norte de la India". Revista Internacional de Inmunogenética . 34 (5): 313–5. doi : 10.1111 / j.1744-313X.2007.00698.x . PMID 17845299 . S2CID 13210660 .
- ^ Kshatriya GK (diciembre de 1995). "Afinidades genéticas de las poblaciones de Sri Lanka". Biología humana . Asociación Americana de Genética Antropológica. 67 (6): 843–66. PMID 8543296 .
- ^ Mastana, Sarabjit (noviembre de 1996). "Variación genética en Sri Lanka" (PDF) . Informes científicos : 26-27.
- ^ Soejima M, Koda Y (diciembre de 2005). "Desnaturalización de la variación genética y genotipado basado en cromatografía líquida de alto rendimiento de FUT2 en Sri Lanka". Transfusión . 45 (12): 1934–9. doi : 10.1111 / j.1537-2995.2005.00651.x . PMID 16371047 . S2CID 10401001 .
- ^ Saha N (junio de 1988). "Marcadores genéticos de sangre en poblaciones de Sri Lanka - reevaluación de la leyenda del Príncipe Vijaya". Revista Estadounidense de Antropología Física . 76 (2): 217-25. doi : 10.1002 / ajpa.1330760210 . PMID 3166342 .
- ^ Roberts DF, Creen CK, Abeyaratne KP (1972). "Grupos sanguíneos de los cingaleses". Hombre . 7 (1): 122-127. doi : 10.2307 / 2799860 . JSTOR 2799860 .
- ^ Dissanayake VH, Giles V, Jayasekara RW, Seneviratne HR, Kalsheker N, Broughton Pipkin F, Morgan L (abril de 2009). "Un estudio de tres genes candidatos para la preeclampsia en una población cingalesa de Sri Lanka". Revista de investigación en obstetricia y ginecología . 35 (2): 234–42. doi : 10.1111 / j.1447-0756.2008.00926.x . PMID 19708171 . S2CID 24958292 .
- ^ Illeperuma RJ, Mohotti SN, De Silva TM, Fernandopulle ND, Ratnasooriya WD (junio de 2009). "Perfil genético de 11 loci STR autosómicos entre los cuatro grupos étnicos principales en Sri Lanka". Internacional de Ciencias Forenses. Genética . 3 (3): e105-6. doi : 10.1016 / j.fsigen.2008.10.002 . PMID 19414153 .
- ^ Deng L, Hoh BP, Lu D, Saw WY, Twee-Hee Ong R, Kasturiratne A, et al. (Septiembre de 2015). "Disección de la estructura genética y la mezcla de cuatro poblaciones malayas geográficas" . Informes científicos . 5 (1): 14375. Bibcode : 2015NatSR ... 514375D . doi : 10.1038 / srep14375 . PMC 4585825 . PMID 26395220 .
- ↑ a b Petraglia MD, Allchin B (2007). La evolución y la historia de las poblaciones humanas en el sur de Asia: estudios interdisciplinarios en arqueología, antropología biológica, lingüística y genética . Springer Science & Business Media. ISBN 978-1-4020-5562-1.[ página necesaria ]
- ↑ a b c Soejima M, Koda Y (enero de 2007). "Diferencias de población de dos SNP codificantes en los genes relacionados con la pigmentación SLC24A5 y SLC45A2". Revista Internacional de Medicina Legal . 121 (1): 36–9. doi : 10.1007 / s00414-006-0112-z . PMID 16847698 . S2CID 11192076 .
- ^ Kivisild T, Rootsi S, Metspalu M, Mastana S, Kaldma K, Parik J, et al. (Febrero de 2003). "La herencia genética de los primeros colonos persiste tanto en las poblaciones tribales indias como en las castas" . Revista Estadounidense de Genética Humana . 72 (2): 313–32. doi : 10.1086 / 346068 . PMC 379225 . PMID 12536373 .
- ^ Sengupta S, Zhivotovsky LA, King R, Mehdi SQ, Edmonds CA, Chow CE, et al. (Febrero de 2006). "La polaridad y la temporalidad de las distribuciones del cromosoma y de alta resolución en la India identifican expansiones tanto indígenas como exógenas y revelan una influencia genética menor de los pastores de Asia Central" . Revista Estadounidense de Genética Humana . 78 (2): 202-21. doi : 10.1086 / 499411 . PMC 1380230 . PMID 16400607 .
- ↑ a b Roychoudhury AK, Nei M (1985). "Relaciones genéticas entre indígenas y sus poblaciones vecinas". Herencia humana . 35 (4): 201–6. doi : 10.1159 / 000153545 . PMID 4029959 .
- ^ Bhasin MK (4 de septiembre de 2017). "Morfología a la antropología molecular: castas y tribus de la India". Revista Internacional de Genética Humana . 9 (3–4): 145–230. doi : 10.1080 / 09723757.2009.11886070 . S2CID 53353581 .
- ^ Peiris R, Takahashi M, Sasaki K, Kanazawa E (julio de 2007). "Morfología de la raíz y el conducto de los molares mandibulares permanentes en una población de Sri Lanka". Odontología . 95 (1): 16–23. doi : 10.1007 / s10266-007-0074-8 . PMID 17660977 . S2CID 8504778 .
- ^ Ranaweera L, Kaewsutthi S, Win Tun A, Boonyarit H, Poolsuwan S, Lertrit P (enero de 2014). "Historia del ADN mitocondrial de los pueblos étnicos de Sri Lanka: sus relaciones dentro de la isla y con las poblaciones subcontinentales de la India" . Revista de Genética Humana . 59 (1): 28–36. doi : 10.1038 / jhg.2013.112 . PMID 24196378 .
- ^ "Haplogrupo C de Y-DNA y sus subclades - 2017" . Sociedad Internacional de Genealogía Genética . Consultado el 31 de mayo de 2017 .
- ↑ a b c Matsumoto H (2009). "El origen de la raza japonesa basado en marcadores genéticos de inmunoglobulina G" . Actas de la Academia de Japón. Serie B, Ciencias Físicas y Biológicas . 85 (2): 69–82. Código bibliográfico : 2009PJAB ... 85 ... 69M . doi : 10.2183 / pjab.85.69 . PMC 3524296 . PMID 19212099 .
- ^ Fujihara J, Yasuda T, Iida R, Ueki M, Sano R, Kominato Y, et al. (Julio de 2015). "Análisis global de variaciones genéticas en un número variable de 56 pb de polimorfismos de repetición en tándem dentro del gen de la desoxirribonucleasa I humana". Medicina legal . 17 (4): 283–6. doi : 10.1016 / j.legalmed.2015.01.005 . PMID 25771153 .
- ^ Malhotra R (1992). Antropología del desarrollo: volumen conmemorativo en honor del profesor IP Singh . Publicaciones Mittal. ISBN 978-81-7099-328-5.[ página necesaria ]
- ^ Kumar D (2012). Trastornos genéticos del subcontinente indio . Springer Science & Business Media. ISBN 978-1-4020-2231-9.[ página necesaria ]
- ^ "rs1426654 Cromosoma chr15: 46213776" . Universidad Stanford. 2009.
enlaces externos
- Genealogía genética
- Genética de poblaciones humanas
- Genética por etnia
- Pueblos indo-arios
- Pueblo cingalés