Retrovirus endógeno
Los retrovirus endógenos ( ERV ) son elementos virales endógenos en el genoma que se parecen mucho a los retrovirus y pueden derivarse de ellos. Son abundantes en los genomas de los vertebrados con mandíbula y comprenden hasta el 5-8% del genoma humano (estimaciones más bajas de ~ 1%). [1] [2] Los ERV son unasecuencia proviral heredada verticalmentey una subclase de un tipo de gen llamado transposón , que normalmente se puede empaquetar y mover dentro del genoma para desempeñar un papel vital en la expresión génica y en la regulación . [3] [4]Sin embargo, los ERV carecen de la mayor parte de la función de los transposones, por lo general no son infecciosos y, a menudo, son restos genómicos defectuosos del ciclo de replicación retroviral. [5] [6] Se distinguen como retroelementos de provirus de línea germinal debido a su integración y transcripción inversa en el genoma nuclear de la célula huésped. Los investigadores han sugerido que los retrovirus evolucionaron a partir de un tipo de transposón llamado retrotransposón, un elemento de Clase I; [7] estos genes pueden mutar y, en lugar de trasladarse a otra ubicación del genoma, pueden volverse exógenos o patógenos. Esto significa que es posible que no todos los ERV se hayan originado como una inserción de un retrovirus, pero que algunos pueden haber sido la fuente de información genética en los retrovirus a los que se asemejan. [8] Cuando la integración del ADN viral ocurre en la línea germinal, puede dar lugar a un ERV, que más tarde se puede fijar en el acervo genético de la población huésped. [1] [9]
Formación
El ciclo de replicación de un retrovirus implica la inserción ("integración") de una copia de ADN del genoma viral en el genoma nuclear de la célula huésped . La mayoría de los retrovirus infectan las células somáticas , pero también puede ocurrir una infección ocasional de las células de la línea germinal (células que producen óvulos y espermatozoides). En raras ocasiones, la integración retroviral puede ocurrir en una célula de la línea germinal que se convierte en un organismo viable. Este organismo llevará el genoma retroviral insertado como parte integral de su propio genoma, un retrovirus "endógeno" (ERV) que puede ser heredado por su descendencia como un alelo nuevo.. Muchos ERV han persistido en el genoma de sus huéspedes durante millones de años. Sin embargo, la mayoría de estos han adquirido mutaciones inactivadoras durante la replicación del ADN del hospedador y ya no son capaces de producir el virus. Los ERV también pueden escindirse parcialmente del genoma mediante un proceso conocido como deleción recombinacional, en el que la recombinación entre las secuencias idénticas que flanquean los retrovirus recién integrados da como resultado la deleción de las regiones codificadoras de proteínas internas del genoma viral.
El genoma general del retrovirus consta de tres genes vitales para la invasión, replicación, escape y propagación de su genoma viral. Estos tres genes son gag (codifica las proteínas estructurales del núcleo viral), pol (codifica la transcriptasa inversa , la integrasa y la proteasa ) y env (codifica las proteínas de la cubierta del exterior del virus). Estas proteínas virales están codificadas como poliproteínas.. Para llevar a cabo su ciclo de vida, el retrovirus depende en gran medida de la maquinaria de la célula huésped. La proteasa degrada los enlaces peptídicos de las poliproteínas virales, haciendo que las proteínas separadas sean funcionales. La transcriptasa inversa funciona para sintetizar el ADN viral a partir del ARN viral en el citoplasma de la célula huésped antes de que ingrese al núcleo. La integrasa guía la integración del ADN viral en el genoma del hospedador. [9] [10]
Con el tiempo, el genoma de los ERV no solo adquiere mutaciones puntuales, sino que también se mezcla y se recombina con otros ERV. [11] Es más probable que los ERV con una secuencia decaída para el env se propaguen. [12]
Papel en la evolución genómica
Los retrovirus endógenos pueden desempeñar un papel activo en la formación de genomas. La mayoría de los estudios en esta área se han centrado en los genomas de humanos y primates superiores, pero también se han estudiado en profundidad otros vertebrados, como ratones y ovejas. [13] [14] [15] [16] Las secuencias de repetición terminal larga ( LTR ) que flanquean los genomas del ERV con frecuencia actúan como promotores y potenciadores alternativos, y a menudo contribuyen al transcriptoma mediante la producción de variantes específicas de tejido. Además, las proteínas retroviralesellos mismos han sido cooptados para cumplir funciones novedosas del huésped, particularmente en reproducción y desarrollo. La recombinación entre secuencias retrovirales homólogas también ha contribuido a la mezcla de genes y la generación de variación genética. Además, en el caso de los efectos potencialmente antagonistas de las secuencias retrovirales, los genes represores han evolucionado conjuntamente para combatirlos.
Aproximadamente el 90% de los retrovirus endógenos son LTR individuales, que carecen de todos los marcos de lectura abiertos (ORF). [17] Se ha demostrado que las LTR y LTR solo asociadas con secuencias retrovirales completas actúan como elementos transcripcionales en los genes del huésped. Su rango de acción es principalmente por inserción en las 5 ' UTR de genes codificantes de proteínas; sin embargo, se sabe que actúan sobre genes de hasta 70-100 kb de distancia. [13] [18] [19] [20] La mayoría de estos elementos se insertan en la dirección del sentido de sus genes correspondientes, pero ha habido evidencia de que las LTR actúan en la dirección antisentido y como promotores bidireccionales para genes vecinos. [21] [22]En algunos casos, el LTR funciona como el principal promotor del gen. Por ejemplo, en humanos, AMY1C tiene una secuencia ERV completa en su región promotora; la LTR asociada confiere expresión salival específica de la enzima digestiva amilasa . [23] Además, el promotor principal del ácido biliar-CoA: aminoácido N-aciltransferasa (BAAT), que codifica una enzima que es integral en el metabolismo de la bilis, es de origen LTR. [19] [24]
La inserción de un ERV-9 LTR solo puede haber producido un marco de lectura abierto funcional, provocando el renacimiento del gen GTPasa relacionado con la inmunidad humana (IRGM). [25] También se ha demostrado que las inserciones de ERV generan sitios de empalme alternativos ya sea por integración directa en el gen, como con el receptor de la hormona leptina humana, o impulsadas por la expresión de un LTR corriente arriba, como con la proteína similar a la fosfolipasa A-2. [26]
Sin embargo, la mayoría de las veces, el LTR funciona como uno de los muchos promotores alternativos, lo que a menudo confiere una expresión específica de tejido relacionada con la reproducción y el desarrollo. De hecho, el 64% de las variantes de transcripción promovidas por LTR conocidas se expresan en tejidos reproductivos. [27] Por ejemplo, el gen CYP19 codifica la aromatasa P450, una enzima importante para la síntesis de estrógenos, que normalmente se expresa en el cerebro y los órganos reproductores de la mayoría de los mamíferos. [19] Sin embargo, en primates, una variante transcripcional promovida por LTR confiere expresión a la placenta y es responsable de controlar los niveles de estrógeno durante el embarazo. [19] Además, la proteína inhibidora de la apoptosis neuronal(NAIP), normalmente extendido, tiene una LTR de la familia HERV-P que actúa como un promotor que confiere expresión a los testículos y la próstata. [28] Otras proteínas, como la óxido nítrico sintasa 3 (NOS3), el receptor B de interleucina-2 (IL2RB) y otro mediador de la síntesis de estrógenos, HSD17B1, también están reguladas alternativamente por LTR que confieren expresión placentaria, pero sus funciones específicas son aún no conocido. [24] [29] Se cree que el alto grado de expresión reproductiva es un efecto secundario del método mediante el cual fueron endogenizados; sin embargo, esto también puede deberse a la falta de metilación del ADN en los tejidos de la línea germinal. [24]
La instancia mejor caracterizada de expresión de la proteína placentaria no proviene de un gen huésped promovido alternativamente, sino de una cooptación completa de una proteína retroviral. Las proteínas env fusogénicas retrovirales, que desempeñan un papel en la entrada del virión en la célula huésped, han tenido un impacto importante en el desarrollo de la placenta de los mamíferos . En los mamíferos, las proteínas env intactas llamadas sincitinas son responsables de la formación y función de los sincitiotrofoblastos . [15] Estas células multinucleadas son principalmente responsables de mantener el intercambio de nutrientes y separar al feto del sistema inmunológico de la madre. [15]Se ha sugerido que la selección y fijación de estas proteínas para esta función han jugado un papel crítico en la evolución de la viviparidad . [30]
Además, la inserción de ERV y sus respectivas LTR tienen el potencial de inducir un reordenamiento cromosómico debido a la recombinación entre secuencias virales en loci intercromosómicos. Se ha demostrado que estos reordenamientos inducen duplicaciones y deleciones de genes que contribuyen en gran medida a la plasticidad del genoma y cambian drásticamente la dinámica de la función de los genes. [31] Además, los retroelementos en general prevalecen en gran medida en familias de genes específicos de mamíferos que evolucionan rápidamente, cuya función está relacionada en gran medida con la respuesta al estrés y los estímulos externos. [19] En particular, tanto los genes humanos del MHC de clase I como de clase II tienen una alta densidad de elementos HERV en comparación con otras familias de genes de múltiples locus. [26]Se ha demostrado que los HERV han contribuido a la formación de bloques de duplicones ampliamente duplicados que componen la familia de genes HLA de clase 1. [32] Más específicamente, los HERV ocupan principalmente regiones dentro y entre los puntos de ruptura entre estos bloques, lo que sugiere que los eventos de duplicación y deleciones considerables, típicamente asociados con cruces desiguales, facilitaron su formación. [33] La generación de estos bloques, heredados como inmunohaplotipos, actúa como un polimorfismo protector contra una amplia gama de antígenos que pueden haber imbuido a los humanos con una ventaja sobre otros primates. [32]
Se ha sugerido que la característica de que las placentas son órganos evolutivos distintos entre diferentes especies es el resultado de la cooptación de potenciadores de ERV. Las mutaciones reguladoras, en lugar de mutaciones en genes que codifican hormonas y factores de crecimiento , apoyan la evolución conocida de la morfología placentaria, especialmente porque la mayoría de los genes de hormonas y factores de crecimiento se expresan en respuesta al embarazo, no durante el desarrollo placentario. Los investigadores estudiaron el panorama regulador del desarrollo placentario entre la rata y el ratón, dos especies estrechamente relacionadas. Esto se hizo mapeando todos los elementos reguladores de las células madre del trofoblasto de rata (TSC) y comparándolos con susortólogos en TSC de ratón. Se observaron TSC porque reflejan las células iniciales que se desarrollan en la placenta fetal. Independientemente de sus similitudes tangibles, las regiones potenciadoras y reprimidas eran en su mayoría específicas de especies. Sin embargo, la mayoría de las secuencias promotoras se conservaron entre el ratón y la rata. En conclusión de su estudio, los investigadores propusieron que los ERV influyeron en la evolución placentaria específica de la especie a través de la mediación del crecimiento placentario, la inmunosupresión y la fusión celular . [34]
Otro ejemplo de ERV que explota los mecanismos celulares es p53 , un gen supresor de tumores (TSG). El daño del ADN y el estrés celular inducen la vía p53, que da como resultado la apoptosis celular . Usando inmunoprecipitación de cromatina con secuenciación, el treinta por ciento de todos los sitios de unión a p53 se ubicaron dentro de copias de algunas familias de ERV específicas de primates. Un estudio sugirió que esto beneficia a los retrovirus porque el mecanismo de p53 proporciona una rápida inducción de la transcripción, lo que conduce a la salida del ARN viral de la célula huésped. [7]
Por último, la inserción de ERV o elementos de ERV en regiones génicas del ADN del huésped, o la sobreexpresión de sus variantes transcripcionales, tiene un potencial mucho mayor de producir efectos deletéreos que los positivos. Su aparición en el genoma ha creado una dinámica coevolutiva de huésped-parásito que proliferó la duplicación y expansión de genes represores. El ejemplo más claro de esto implica la rápida duplicación y proliferación de genes de dedos de zinc en tándem en genomas de mamíferos. Los genes de dedos de zinc, particularmente aquellos que incluyen un dominio KRAB, existen en un alto número de copias en los genomas de vertebrados, y su rango de funciones se limita a roles transcripcionales. [35]Sin embargo, se ha demostrado en mamíferos que la diversificación de estos genes se debe a múltiples eventos de duplicación y fijación en respuesta a nuevas secuencias retrovirales o sus copias endógenas para reprimir su transcripción. [20]
Papel en la enfermedad
La mayoría de los ERV que se encuentran en los genomas de los vertebrados son antiguos, inactivados por mutación y han alcanzado la fijación genética en su especie huésped. Por estas razones, es muy poco probable que tengan efectos negativos en sus anfitriones, excepto en circunstancias inusuales. No obstante, está claro a partir de estudios en aves y especies de mamíferos no humanos, incluidos ratones, gatos y koalas , que los ERV más jóvenes (es decir, más recientemente integrados) pueden asociarse con enfermedades. [36] El número de ERV activos en el genoma de los mamíferos está relacionado negativamente con el tamaño de su cuerpo, lo que sugiere una contribución a la paradoja de Peto a través de la patogénesis del cáncer. [37] Esto ha llevado a los investigadores a proponer un papel para los ERV en varias formas de cáncer humano y enfermedades autoinmunes , aunque faltan pruebas concluyentes. [38] [39] [40] [41]
Desórdenes neurológicos
En los seres humanos, se ha propuesto que los ERV están implicados en la esclerosis múltiple (EM). Se ha informado de una asociación específica entre la EM y el gen ERVWE1 , o "sincitina", que se deriva de una inserción de ERV, junto con la presencia de un "retrovirus asociado a EM" (MSRV), en pacientes con la enfermedad. [42] [43] Los ERV humanos (HERV) también se han relacionado con la ELA [44] y la adicción. [45] [46] [47]
En 2004 se informó que los anticuerpos contra HERV se encontraron con mayor frecuencia en el suero de personas con esquizofrenia . Además, el líquido cefalorraquídeo de personas con esquizofrenia de inicio reciente contenía niveles de un marcador retroviral, la transcriptasa inversa , cuatro veces más altos que los sujetos de control. [48] Los investigadores continúan buscando un posible vínculo entre los HERV y la esquizofrenia, con la posibilidad adicional de que una infección desencadenante induzca esquizofrenia . [49]
Inmunidad
Se ha descubierto que los ERV están asociados a enfermedades no solo a través de las relaciones que las causan, sino también a través de la inmunidad. La frecuencia de ERV en repeticiones terminales largas (LTR) probablemente se correlaciona con adaptaciones virales para aprovechar las vías de señalización de inmunidad que promueven la transcripción y replicación viral. Un estudio realizado en 2016 investigó el beneficio del ADN viral antiguo integrado en un huésped a través de redes de regulación genética inducidas por interferones , una rama de la inmunidad innata. [50] Estas citocinas son las primeras en responder a la infección viral y también son importantes en la vigilancia inmunológica de células malignas. [51]Se prevé que los ERV actúen como elementos reguladores en cis, pero aún se desconocen muchas de las consecuencias adaptativas de esto para ciertas funciones fisiológicas. Hay datos que apoyan el papel general de los ERV en la regulación de la respuesta al interferón humano, específicamente al interferón-gamma (IFNG). Por ejemplo, se encontró que los genes estimulados por interferón estaban muy enriquecidos con ERV unidos por el transductor de señal y el activador de la transcripción (STAT1) y / o el factor regulador de interferón (IRF1) en macrófagos CD14 + . [1]
Los HERV también desempeñan varias funciones en la configuración de la respuesta de la inmunidad innata humana , con algunas secuencias que activan el sistema y otras lo suprimen. También pueden proteger de infecciones retrovirales exógenas: las transcripciones similares a virus pueden activar receptores de reconocimiento de patrones y las proteínas pueden interferir con retrovirus activos. Se ha demostrado que una proteína gag de HERV-K (HML2) se mezcla con VIH Gag, lo que dificulta la formación de la cápside del VIH como resultado. [52]
Regulación genética
Otra idea propuesta fue que los ERV de la misma familia desempeñaban un papel en el reclutamiento de múltiples genes en la misma red de regulación. Se encontró que los elementos de MER41 proporcionaban además una mejora reguladora redundante a los genes ubicados cerca de los sitios de unión de STAT1. [1]
Papel en la medicina
Retrovirus endógeno porcino
Para los seres humanos, los retrovirus endógenos porcinos (PERV) plantean una preocupación cuando se utilizan tejidos y órganos porcinos en xenotrasplantes, el trasplante de células, tejidos y órganos vivos de un organismo de una especie a un organismo de diferentes especies. Aunque los cerdos son generalmente los donantes más adecuados para tratar enfermedades de órganos humanos debido a razones prácticas, económicas, de seguridad y éticas, [50] anteriormente los PERV no podían eliminarse de los cerdos debido a su naturaleza viral de integrarse en el genoma del huésped y ser transmitidos. en la descendencia hasta el año 2017, cuando el laboratorio del Dr. George Church eliminó los 62 retrovirus del genoma de los cerdos. [53] Las consecuencias de la transmisión entre especies siguen sin explorarse y tienen un potencial muy peligroso. [54]
Los investigadores indicaron que la infección de tejidos humanos por PERV es muy posible, especialmente en individuos inmunosuprimidos. Una condición inmunodeprimida podría permitir una replicación más rápida y tenaz del ADN viral y, más adelante, tendría menos dificultades para adaptarse a la transmisión de persona a persona. Aunque los patógenos infecciosos conocidos presentes en el órgano / tejido del donante pueden eliminarse criando rebaños libres de patógenos, el donante puede contener retrovirus desconocidos. Estos retrovirus suelen estar latentes y asintomáticos en el donante, pero pueden volverse activos en el receptor. Algunos ejemplos de virus endógenos que pueden infectar y multiplicarse en células humanas son los de babuinos (BaEV), gatos (RD114) y ratones. [50]
Hay tres clases diferentes de PERV, PERV-A, PERV-B y PERV-C. PERV-A y PERV-B son politrópicos y pueden infectar células humanas in vitro, mientras que PERV-C es ecotrópico y no se replica en células humanas. Las principales diferencias entre las clases se encuentran en el dominio de unión al receptor de la proteína env y las repeticiones terminales largas (LTR) que influyen en la replicación de cada clase. PERV-A y PERV-B muestran LTR que tienen repeticiones en el U3región. Sin embargo, PERV-A y PERV-C muestran LTR sin repetición. Los investigadores encontraron que los PERV en cultivo se adaptaban activamente a la estructura repetida de su LTR para igualar el mejor rendimiento de replicación que podría realizar una célula huésped. Al final de su estudio, los investigadores concluyeron que la LTR PERV repetida evolucionó a partir de la LTR portadora repetida. Es probable que esto haya ocurrido a partir de una mutación de inserción y se demostró mediante el uso de datos sobre LTR y env / Env. Se cree que la generación de LTR sin repetición podría reflejar un proceso de adaptación del virus, cambiando de un estilo de vida exógeno a uno endógeno. [55]
En un ensayo clínico realizado en 1999, se tomaron muestras de 160 pacientes que fueron tratados con diferentes tejidos de cerdo vivo y no se observó evidencia de una infección persistente por PERV en el 97% de los pacientes para los que se disponía de una cantidad suficiente de ADN para la PCR para la amplificación de las secuencias de PERV. Sin embargo, este estudio indicó que los estudios retrospectivos se limitan a encontrar la verdadera incidencia de infección o síntomas clínicos asociados. Sugirió el uso de ensayos prospectivos monitoreados de cerca, lo que proporcionaría una evaluación más completa y detallada de la posible transmisión de PERV entre especies y una comparación de PERV. [56]
Retrovirus endógenos humanos
Los retrovirus endógenos humanos (HERV) comprenden una parte significativa del genoma humano , con aproximadamente 98 000 elementos y fragmentos de ERV que constituyen el 5–8%. [1] Según un estudio publicado en 2005, no se había identificado ningún HERV capaz de replicarse; todos parecían ser defectuosos, con deleciones importantes o mutaciones sin sentido. Esto se debe a que la mayoría de los HERV son simplemente rastros de virus originales, que se integraron por primera vez hace millones de años. Se está realizando un análisis de las integraciones de HERV como parte del Proyecto 100,000 Genomes . [57]
Los retrovirus endógenos humanos se descubrieron por accidente utilizando un par de experimentos diferentes. Se seleccionaron bibliotecas genómicas humanas en condiciones de baja rigurosidad utilizando sondas de retrovirus animales, lo que permitió el aislamiento y caracterización de provirus múltiples, aunque defectuosos, que representaban varias familias. Otro experimento dependió de oligonucleótidos con homología con los sitios de unión del cebador viral. [1]
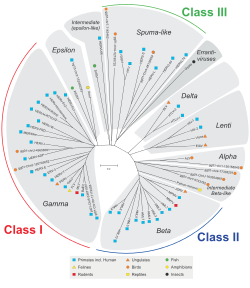
Los HERV se clasifican en función de sus homologías con los retrovirus animales. Las familias que pertenecen a la Clase I son similares en secuencia a los Gammaretrovirus (tipo C) y Epsilonretrovirus (Tipo E) de mamíferos . Las familias que pertenecen a la clase II muestran homología con los betaretrovirus de mamíferos (tipo B) y los deltaretrovirus (tipo D). Las familias que pertenecen a la Clase III son similares a los virus espumosos . Para todas las clases, si las homologías aparecen bien conservadas en los genes gag , pol y env , se agrupan en una superfamilia . Se sabe que existen más familias de Clase I. [1] [11]Las familias mismas se nombran de una manera menos uniforme, con una mezcla de nombres basada en un retrovirus exógeno, el ARNt de cebado (HERV-W, K) o algún gen vecino (HERV-ADP), número de clon (HERV-S71) , o algún motivo de aminoácidos (HERV-FRD). Una nomenclatura propuesta tiene como objetivo limpiar los estándares a veces parafiléticos. [58]
Hay dos propuestas sobre cómo los HERV se fijaron en el genoma humano. El primero asume que en algún momento durante la evolución humana, los progenitores exógenos de HERV se insertaron en las células de la línea germinal y luego se replicaron junto con los genes del huésped utilizando y explotando los mecanismos celulares del huésped. Debido a su estructura genómica distinta, los HERV se sometieron a muchas rondas de amplificación y transposición, lo que condujo a una distribución generalizada de ADN retroviral. La segunda hipótesis afirma la evolución continua de retroelementos a partir de antepasados estructurados más simples. [1]
Sin embargo, una familia de virus ha estado activa desde la divergencia entre humanos y chimpancés . Esta familia, denominada HERV-K (HML2), constituye menos del 1% de los elementos HERV, pero es una de las más estudiadas. Hay indicios de que incluso ha estado activo en los últimos cientos de miles de años, por ejemplo, algunos individuos humanos llevan más copias de HML2 que otros. [59] Tradicionalmente, las estimaciones de edad de los HERV se realizan comparando el LTR de 5 'y 3' de un HERV; sin embargo, este método solo es relevante para los HERV completos. Un método reciente, llamado datación transversal, [60]utiliza variaciones dentro de una sola LTR para estimar las edades de las inserciones de HERV. Este método es más preciso para estimar las edades de HERV y se puede utilizar para cualquier inserción de HERV. La datación transversal se ha utilizado para sugerir que dos miembros de HERV-K (HML2), HERV-K106 y HERV-K116, estuvieron activos en los últimos 800.000 años y que HERV-K106 pudo haber infectado a los humanos modernos hace 150.000 años. [61] Sin embargo, la ausencia de miembros infecciosos conocidos de la familia HERV-K (HML2) y la falta de elementos con un potencial completo de codificación dentro de la secuencia del genoma humano publicada, sugiere para algunos que es menos probable que la familia sea activa. Actualmente. En 2006 y 2007, investigadores que trabajaban de forma independiente en Francia y Estados Unidos recrearon versiones funcionales de HERV-K (HML2). [62] [63]
MER41.AIM2 es un HERV que regula la transcripción de AIM2 (ausente en el melanoma 2) que codifica un sensor de ADN citosólico extraño. Esto actúa como un sitio de unión para AIM2, lo que significa que es necesario para la transcripción de AIM2. Los investigadores habían demostrado esto al eliminar MER41.AIM2 en células HeLa usando CRISPR / Cas9, lo que condujo a un nivel de transcripción indetectable de AIM2 en células HeLa modificadas. Las células de control, que todavía contenían el ERV MER41.AIM2, se observaron con cantidades normales de transcripción AIM2. En términos de inmunidad, los investigadores concluyeron que MER41.AIM2 es necesario para una respuesta inflamatoria a la infección. [64]
Los estudios inmunológicos han mostrado alguna evidencia de las respuestas inmunitarias de las células T contra los HERV en personas infectadas por el VIH. [65] La hipótesis de que el VIH induce la expresión de HERV en las células infectadas por el VIH llevó a la propuesta de que una vacuna dirigida a los antígenos de HERV podría eliminar específicamente las células infectadas por el VIH. La ventaja potencial de este nuevo enfoque es que, mediante el uso de antígenos HERV como marcadores sustitutos de células infectadas por el VIH, podría sortear la dificultad inherente de dirigirse directamente a antígenos del VIH notoriamente diversos y de rápida mutación. [sesenta y cinco]
Hay algunas clases de retrovirus endógenos humanos que todavía tienen marcos de lectura abiertos intactos. Por ejemplo, la expresión de HERV-K, una familia biológicamente activa de HERV, produce proteínas que se encuentran en la placenta. Además, la expresión de los genes de la envoltura de HERV-W ( ERVW-1 ) y HERV-FRD ( ERVFRD-1 ) produce sincitinas que son importantes para la generación de la capa de células sincitiotrofoblasto durante la placentogénesis al inducir la fusión célula-célula. [66] El Comité de Nomenclatura Genética de HUGO (HGNC) aprueba los símbolos genéticos para los ERV humanos transcritos. [67]
Técnicas para caracterizar ERV
Secuenciación del genoma completo
Ejemplo: Se secuenció completamente y junto con diferentes razas y líneas celulares un aislado de minipig nacido en China de ERV porcino (PERV) , PERV-A-BM, para comprender su variación genética y evolución. El número observado de sustituciones de nucleótidos y entre las diferentes secuencias del genoma ayudó a los investigadores a determinar una edad estimada en la que PERV-A-BM se integró en su genoma huésped, que se encontró que era de una edad evolutiva anterior a los aislados de cerdos nacidos en Europa. [54]
Inmunoprecipitación de cromatina con secuenciación (ChIP-seq)
Esta técnica se utiliza para encontrar marcas de histonas indicativas de promotores y potenciadores, que son sitios de unión para proteínas de ADN y regiones reprimidas y trimetilación. [34] Se ha demostrado que la metilación del ADN es vital para mantener el silenciamiento de los ERV en las células somáticas de ratón, mientras que las marcas de histonas son vitales para el mismo propósito en las células madre embrionarias (ESC) y en la embriogénesis temprana. [7]
Aplicaciones
Construyendo filogenias
Debido a que la mayoría de los HERV no tienen función, son selectivamente neutrales y son muy abundantes en los genomas de primates, sirven fácilmente como marcadores filogenéticos para el análisis de ligamiento. Pueden explotarse comparando los polimorfismos del sitio de integración o las secuencias de nucleótidos provirales en evolución de los ortólogos. Para estimar cuándo ocurrió la integración, los investigadores utilizaron distancias de cada árbol filogenético para encontrar la tasa de evolución molecular en cada locus en particular. También es útil que los ERV sean ricos en genomas de muchas especies (es decir, plantas, insectos, moluscos, peces, roedores, mascotas domésticas y ganado) porque su aplicación puede usarse para responder una variedad de preguntas filogenéticas. [9]
Designación de la edad del provirus y los puntos temporales de los eventos de separación de especies
Esto se logra comparando los diferentes HERV de diferentes períodos evolutivos. Por ejemplo, este estudio se realizó para diferentes hominoides, que iban desde humanos hasta simios y monos. Esto es difícil de hacer con PERV debido a la gran diversidad presente. [55]
Más investigación
Variabilidad epigenética
Los investigadores podrían analizar epigenomas y transcriptomas individuales para estudiar la reactivación de elementos transponibles latentes a través de la liberación epigenética y sus posibles asociaciones con enfermedades humanas y explorar los detalles de las redes reguladoras de genes. [7]
Problemas inmunológicos del xenotrasplante
Se sabe poco sobre una forma eficaz de superar el rechazo hiperagudo (HAR), que sigue a la activación del complemento iniciada por anticuerpos xenorreactivos que reconocen antígenos de galactosil-alfa1-3galatosil (alfa-Gal) en el epitelio del donante. [50]
Factores de riesgo de HERV en terapia génica
Debido a que los retrovirus pueden recombinarse entre sí y con otras secuencias de ADN endógenas, sería beneficioso para la terapia génica explorar los riesgos potenciales que pueden causar los HERV, si los hay. Además, esta capacidad de los HERV para recombinarse puede manipularse para la integración dirigida al sitio mediante la inclusión de secuencias de HERV en vectores retrovirales. [1]
Expresión del gen HERV
Los investigadores creen que el ARN y las proteínas codificadas por los genes HERV deben seguir siendo explorados para determinar su función putativa en la fisiología celular y en condiciones patológicas. Tendría sentido examinar esto para definir más profundamente el significado biológico de las proteínas sintetizadas. [1]
Ver también
- Virus de la leucosis del sarcoma aviar (ASLV)
- Elemento viral endógeno
- ERV3
- HERV-FRD
- Transferencia horizontal de genes
- Retrovirus de la oveja Jaagsiekte (JSRV)
- Retrovirus koala (KoRV)
- Virus del tumor mamario del ratón (MMTV)
- Virus de la leucemia murina (MLV) y virus xenotrópico relacionado con el virus de la leucemia murina (XMRV)
- Paleovirología
Referencias
- ↑ a b c d e f g h i j Belshaw R, Pereira V, Katzourakis A, Talbot G, Paces J, Burt A, Tristem M (abril de 2004). "Reinfección a largo plazo del genoma humano por retrovirus endógenos" . Proc Natl Acad Sci USA . 101 (14): 4894–99. Código bibliográfico : 2004PNAS..101.4894B . doi : 10.1073 / pnas.0307800101 . PMC 387345 . PMID 15044706 .
- ^ Nelson PN, Hooley P, Roden D, Davari Ejtehadi H, Rylance P, Warren P, et al. (Octubre de 2004). "Retrovirus endógenos humanos: ¿elementos transponibles con potencial?" . Inmunología clínica y experimental . 138 (1): 1–9. doi : 10.1111 / j.1365-2249.2004.02592.x . PMC 1809191 . PMID 15373898 .
- ^ Khodosevich K, Lebedev Y, Sverdlov E (octubre de 2002). "Retrovirus endógenos y evolución humana" . Genómica comparativa y funcional . 3 (6): 494–98. doi : 10.1002 / cfg.216 . PMC 2448423 . PMID 18629260 .
- ^ Kim FJ, Battini JL, Manel N, Sitbon M (enero de 2004). "Aparición de retrovirus vertebrados y captura de envoltura" . Virología . 318 (1): 183–91. doi : 10.1016 / j.virol.2003.09.026 . PMID 14972546 .
- ^ Stoye JP, Boeke JD (1997). "Retrotransposones, retrovirus endógenos y la evolución de retroelementos" . Retrovirus . Cold Spring Harbor (Nueva York): Cold Spring Harbor Laboratory Press. pag. 343. PMID 21433351 . NBK19468 . Consultado el 22 de febrero de 2021 .
- ↑ Gifford RJ, Blomberg J, Coffin JM, Fan H, Heidmann T, Mayer J, Stoye J, Tristem M, Johnson WE (28 de septiembre de 2018). "Nomenclatura para loci de retrovirus endógenos (ERV)" . Retrovirología . 15 (1): 59. doi : 10.1186 / s12977-018-0442-1 . PMC 6114882 . PMID 30153831 .
- ↑ a b c d Rebollo R, Romanish MT, Mager DL (1 de enero de 2012). "Elementos transponibles: una fuente abundante y natural de secuencias reguladoras de genes del huésped" . Revisión anual de genética . 46 (1): 21–42. doi : 10.1146 / annurev-genet-110711-155621 . PMID 22905872 .
- ^ Algodón, J. (2001). "Retrovirus de retrotransposones" . Biología del genoma . 2 (2): 6. doi : 10.1186 / gb-2001-2-2-reports0006 .
Parece que la transición del retrotransposón no viral al retrovirus se ha producido de forma independiente al menos ocho veces, y la fuente del gen de la envoltura responsable de la capacidad infecciosa ahora se puede rastrear hasta un virus en al menos cuatro de estos casos. Esto sugiere que
, potencialmente, cualquier retrotransposón LTR puede convertirse en un virus
mediante la adquisición de genes virales existentes.
- ↑ a b c Johnson WE, Coffin JM (agosto de 1999). "Construcción de filogenias de primates a partir de secuencias de retrovirus antiguas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 96 (18): 10254–60. Código bibliográfico : 1999PNAS ... 9610254J . doi : 10.1073 / pnas.96.18.10254 . PMC 17875 . PMID 10468595 .
- ^ Fujiwara T, Mizuuchi K (agosto de 1988). "Integración del ADN retroviral: estructura de un intermedio de integración". Celular . 54 (4): 497–504. doi : 10.1016 / 0092-8674 (88) 90071-2 . PMID 3401925 . S2CID 37801365 .
- ^ a b Vargiu, L; Rodríguez-Tomé, P; Sperber, GO; Cadeddu, M; Grandi, N; Blikstad, V; Tramontano, E; Blomberg, J (22 de enero de 2016). "Clasificación y caracterización de retrovirus endógenos humanos; las formas de mosaico son comunes" . Retrovirología . 13 : 7. doi : 10.1186 / s12977-015-0232-y . PMC 4724089 . PMID 26800882 .
- ^ Magiorkinis, G; Gifford, RJ; Katzourakis, A; De Ranter, J; Belshaw, R (8 de mayo de 2012). "Los retrovirus endógenos env-menos son superpropagadores genómicos" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (19): 7385–90. doi : 10.1073 / pnas.1200913109 . PMC 3358877 . PMID 22529376 .
- ^ a b Li J, Akagi K, Hu Y, Trivett AL, Hlynialuk CJ, Swing DA, Volfovsky N, Morgan TC, Golubeva Y, Stephens RM, Smith DE, Symer DE (marzo de 2012). "Los retrovirus endógenos de ratón pueden desencadenar la terminación transcripcional prematura a distancia" . Genome Res . 22 (5): 870–84. doi : 10.1101 / gr.130740.111 . PMC 3337433 . PMID 22367191 .
- ^ Spencer TE, Palmarini M (2012). "Retrovirus endógenos de ovejas: un sistema modelo para comprender la adaptación fisiológica a un genoma de rumiantes en evolución" . J Reprod Dev . 58 (1): 33–37. doi : 10.1262 / jrd.2011-026 . PMID 22450282 .
- ↑ a b c Black SG, Arnaud F, Palmarini M, Spencer TE (2010). "Retrovirus endógenos en la diferenciación de trofoblastos y el desarrollo placentario" . Revista estadounidense de inmunología reproductiva . 64 (4): 255–64. doi : 10.1111 / j.1600-0897.2010.00860.x . PMC 4198168 . PMID 20528833 .
- ^ Ryan FP (diciembre de 2004). "Retrovirus endógenos humanos en salud y enfermedad: una perspectiva simbiótica" . Revista de la Real Sociedad de Medicina . 97 (12): 560–65. doi : 10.1258 / jrsm.97.12.560 . PMC 1079666 . PMID 15574851 .
- ^ Ishak, Charles A .; De Carvalho, Daniel D. (2020). "Reactivación de retroelementos endógenos en el desarrollo y la terapia del cáncer" . Revisión anual de biología del cáncer . 4 : 159-176. doi : 10.1146 / annurev-cancerbio-030419-033525 .
- ^ Pi W, Zhu X, Wu M, Wang Y, Fulzele S, Eroglu A, Ling J, Tuan D (julio de 2010). "Función de largo alcance de un retrotransposón intergénico" . PNAS . 107 (29): 12992–97. Código bibliográfico : 2010PNAS..10712992P . doi : 10.1073 / pnas.1004139107 . PMC 2919959 . PMID 20615953 .
- ↑ a b c d e van de Lagemaat LN, Landry JR, Mager DL, Medstrand P (octubre de 2003). "Los elementos transponibles en mamíferos promueven la variación reguladora y la diversificación de genes con funciones especializadas". Trends Genet . 19 (10): 530–36. doi : 10.1016 / j.tig.2003.08.004 . PMID 14550626 .
- ↑ a b Kovalskaya E, Buzdin A, Gogvadze E, Vinogradova T, Sverdlov E (2006). "Los sitios de inicio de la transcripción de LTR retrovirales endógenos humanos funcionales están ubicados entre las regiones R y U5" . Virología . 346 (2): 373–78. doi : 10.1016 / j.virol.2005.11.007 . PMID 16337666 .
- ^ Dunn CA, Romanish MT, Gutierrez LE, van de Lagemaat LN, Mager DL (2006). "Transcripción de dos genes humanos a partir de un promotor de retrovirus endógeno bidireccional". Gene . 366 (2): 335–42. doi : 10.1016 / j.gene.2005.09.003 . PMID 16288839 .
- ^ Gogvadze E, Stukacheva E, Buzdin A, Sverdlov E (2009). "Modulación humana específica de la actividad transcripcional proporcionada por inserciones retrovirales endógenas" . J Virol . 83 (12): 6098-105. doi : 10.1128 / JVI.00123-09 . PMC 2687385 . PMID 19339349 .
- ↑ Ting CN, Rosenberg MP, Snow CM, Samuelson LC, Meisler MH (1992). "Se requieren secuencias retrovirales endógenas para la expresión específica de tejido de un gen de amilasa salival humana" . Genes Dev . 6 (8): 1457–65. doi : 10.1101 / gad.6.8.1457 . PMID 1379564 .
- ↑ a b c Cohen CJ, Lock WM, Mager DL (2009). "LTR retrovirales endógenos como promotores de genes humanos: una evaluación crítica". Gene . 448 (2): 105-14. doi : 10.1016 / j.gene.2009.06.020 . PMID 19577618 .
- ^ Bekpen C, Marques-Bonet T, Alkan C, Antonacci F, Leogrande MB, Ventura M, Kidd JM, Siswara P, Howard JC, Eichler EE (2009). "Muerte y resurrección del gen IRGM humano" . PLOS Genet . 5 (3): e1000403. doi : 10.1371 / journal.pgen.1000403 . PMC 2644816 . PMID 19266026 .
- ↑ a b Jern P, Coffin JM (2008). "Efectos de los retrovirus sobre la función del genoma del huésped". Annu Rev Genet . 42 : 709–32. doi : 10.1146 / annurev.genet.42.110807.091501 . PMID 18694346 .
- ^ Oliver KR, Greene WK (2011). "ADN móvil y la hipótesis de TE-Thrust: evidencia de apoyo de los primates" . ADN de la mafia . 2 (1): 8. doi : 10.1186 / 1759-8753-2-8 . PMC 3123540 . PMID 21627776 .
- ^ Romanish MT, Lock WM, van de Lagemaat LN, Dunn CA, Mager DL (2007). "Reclutamiento repetido de retrotransposones LTR como promotores por el locus antiapoptótico NAIP durante la evolución de los mamíferos" . PLOS Genet . 3 (1): e10. doi : 10.1371 / journal.pgen.0030010 . PMC 1781489 . PMID 17222062 .
- ^ Huh JW, Ha HS, Kim DS, Kim HS (2008). "Expresión restringida por placenta de NOS3 derivada de LTR". Placenta . 29 (7): 602–08. doi : 10.1016 / j.placenta.2008.04.002 . PMID 18474398 .
- ^ Villarreal LP, Villareal LP (febrero de 1997). "Sobre virus, sexo y maternidad" . J. Virol . 71 (2): 859–65. doi : 10.1128 / JVI.71.2.859-865.1997 . PMC 191132 . PMID 8995601 .
- ^ Hughes JF, Coffin JM (diciembre de 2001). "Evidencia de reordenamientos genómicos mediados por retrovirus endógenos humanos durante la evolución de los primates". Genética de la naturaleza . 29 (4): 487–89. doi : 10.1038 / ng775 . PMID 11704760 . S2CID 20262212 .
- ^ a b Dawkins R, Leelayuwat C, Gaudieri S, Tay G, Hui J, Cattley S, et al. (Febrero de 1999). "Genómica del complejo mayor de histocompatibilidad: haplotipos, duplicación, retrovirus y enfermedad". Revisiones inmunológicas . 167 : 275-304. doi : 10.1111 / j.1600-065X.1999.tb01399.x . PMID 10319268 . S2CID 9924684 .
- ^ Doxiadis GG, de Groot N, Bontrop RE (julio de 2008). "Impacto de los retrovirus intrónicos endógenos en la diversidad y estabilidad de clase II del complejo principal de histocompatibilidad" . Revista de Virología . 82 (13): 6667–77. doi : 10.1128 / JVI.00097-08 . PMC 2447082 . PMID 18448532 .
- ↑ a b Chuong EB, Rumi MA, Soares MJ, Baker JC (marzo de 2013). "Los retrovirus endógenos funcionan como elementos potenciadores específicos de la especie en la placenta" . Genética de la naturaleza . 45 (3): 325–29. doi : 10.1038 / ng.2553 . PMC 3789077 . PMID 23396136 .
- ^ Thomas JH, Schneider S (2011). "Coevolución de retroelementos y genes de dedos de zinc en tándem" . Genome Res . 21 (11): 1800–12. doi : 10.1101 / gr.121749.111 . PMC 3205565 . PMID 21784874 .
- ^ McEwen, Gayle K .; Alquezar-Planas, David E .; Dayaram, Anisha; Gillett, ámbar; Tarlinton, Rachael; Mongano, Nigel; Chappell, Keith J .; Henning, Joerg; Tan, Milton; Timms, Peter; Young, Paul R. (26 de febrero de 2021). "Las integraciones retrovirales contribuyen a tasas elevadas de cáncer del huésped durante la invasión de la línea germinal" . Comunicaciones de la naturaleza . 12 (1): 1316. doi : 10.1038 / s41467-021-21612-7 . ISSN 2041-1723 . PMC 7910482 . PMID 33637755 .
- ^ Katzourakis A, Magiorkinis G, Lim AG, Gupta S, Belshaw R, Gifford R (julio de 2014). "El tamaño del cuerpo de un mamífero más grande conduce a una menor actividad retroviral" . PLOS Patógenos . 10 (7): e1004214. doi : 10.1371 / journal.ppat.1004214 . PMC 4102558 . PMID 25033295 .
- ^ Bannert N, Kurth R (octubre de 2004). "Retroelementos y el genoma humano: nuevas perspectivas sobre una vieja relación" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 101 Suppl 2 (Suppl 2): 14572–79. Código bibliográfico : 2004PNAS..10114572B . doi : 10.1073 / pnas.0404838101 . PMC 521986 . PMID 15310846 .
- ^ Nelson PN, Carnegie PR, Martin J, Davari Ejtehadi H, Hooley P, Roden D, et al. (Febrero de 2003). "Desmitificado. Retrovirus endógenos humanos" . Patología molecular . 56 (1): 11–18. doi : 10.1136 / mp.56.1.11 . PMC 1187282 . PMID 12560456 .
- ^ Singh SK (junio de 2007). "Retrovirus endógenos: sospechosos en el mundo de las enfermedades". Microbiología del futuro . 2 (3): 269–75. doi : 10.2217 / 17460913.2.3.269 . PMID 17661701 .
- ^ Magiorkinis G, Belshaw R, Katzourakis A (septiembre de 2013). " 'De ida y vuelta': revisando las funciones fisiopatológicas de los retrovirus endógenos humanos en la era post-genómica" . Transacciones filosóficas de la Royal Society de Londres. Serie B, Ciencias Biológicas . 368 (1626): 20120504. doi : 10.1098 / rstb.2012.0504 . PMC 3758188 . PMID 23938753 .
- ^ Mameli G, Astone V, Arru G, Marconi S, Lovato L, Serra C, et al. (Enero de 2007). "Cerebros y células mononucleares de sangre periférica de pacientes con esclerosis múltiple (EM) hiperexpresan retrovirus asociado a EM / retrovirus endógeno HERV-W, pero no herpesvirus humano 6" . La Revista de Virología General . 88 (Parte 1): 264–74. doi : 10.1099 / vir.0.81890-0 . PMID 17170460 .
- ^ Serra C, Mameli G, Arru G, Sotgiu S, Rosati G, Dolei A (diciembre de 2003). "Modulación in vitro del retrovirus asociado a la esclerosis múltiple (EM) por citocinas: implicaciones para la patogénesis de la EM". Revista de neurovirología . 9 (6): 637–43. doi : 10.1080 / 714044485 . PMID 14602576 .
- ^ "El virus reactivado puede contribuir a la ELA" . 2016-01-23.
- ^ Karamitros T, Hurst T, Marchi E, Karamichali E, Georgopoulou U, Mentis A, et al. (Octubre de 2018). "RASGRF2 está asociado con el abuso de drogas intravenosas y modula la transcripción en un modelo de línea celular" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 115 (41): 10434–39. doi : 10.1073 / pnas.1811940115 . PMC 6187174 . PMID 30249655 .
- ^ Rob Picheta. "La adicción puede provenir de un retrovirus antiguo, dice un estudio" . CNN . Consultado el 13 de octubre de 2019 .
- ^ "Un virus antiguo puede promover la adicción en la gente moderna" . The Economist . 2018-10-02. ISSN 0013-0613 . Consultado el 13 de octubre de 2019 .
- ^ Yolken R (junio de 2004). "Virus y esquizofrenia: un enfoque en el virus del herpes simple" . Herpes . 11 (Supl. 2): 83A – 88A. PMID 15319094 .
- ^ Fox D (2010). "El virus de la locura" . Descubrir . Consultado el 17 de febrero de 2011 .
- ^ a b c d Paciencia C, Takeuchi Y, Weiss RA (marzo de 1997). "Infección de células humanas por un retrovirus endógeno de cerdos". Medicina de la naturaleza . 3 (3): 282–86. doi : 10,1038 / nm0397-282 . PMID 9055854 . S2CID 33977939 .
- ^ Platanias LC (mayo de 2005). "Mecanismos de señalización mediada por interferón tipo I y tipo II" . Reseñas de la naturaleza. Inmunologia . 5 (5): 375–86. doi : 10.1038 / nri1604 . PMID 15864272 .
- ↑ Grandi, N; Tramontano, E (2018). "Los retrovirus endógenos humanos son elementos adquiridos antiguos que todavía dan forma a las respuestas inmunes innatas" . Fronteras en inmunología . 9 : 2039. doi : 10.3389 / fimmu.2018.02039 . PMC 6139349 . PMID 30250470 .
- ^ Niu D, Wei HJ, Lin L, George H, Wang T, Lee IH, Zhao HY, Wang Y, Kan Y, Shrock E, Lesha E, Wang G, Luo Y, Qing Y, Jiao D, Zhao H, Zhou X, Wang S, Wei H, Güell M, Church GM, Yang L (septiembre de 2017). "Inactivación de retrovirus endógeno porcino en cerdos mediante CRISPR-Cas9" . Ciencia . 357 (6357): 1303–07. Código Bibliográfico : 2017Sci ... 357.1303N . doi : 10.1126 / science.aan4187 . PMC 5813284 . PMID 28798043 .
- ^ a b Tang HB, Ouyang K, Rao GB, Ma L, Zhong H, Bai A, et al. (01/01/2016). "La caracterización de las secuencias del genoma completo de un retrovirus endógeno porcino aislado de China Bama Minipig revela un tiempo evolutivo antes que el de los aislados de minipigs europeos". Procedimientos de trasplante . 48 (1): 222-28. doi : 10.1016 / j.transproceed.2015.12.005 . PMID 26915872 .
- ↑ a b Tönjes RR, Niebert M (noviembre de 2003). "Edad relativa de secuencias de retrovirus endógeno porcino proviral en Sus scrofa basado en la hipótesis del reloj molecular" . Revista de Virología . 77 (22): 12363–68. doi : 10.1128 / JVI.77.22.12363-12368.2003 . PMC 254287 . PMID 14581574 .
- ^ Paradis K, Langford G, Long Z, Heneine W, Sandstrom P, Switzer WM, et al. (Agosto de 1999). "Búsqueda de transmisión entre especies de retrovirus endógeno porcino en pacientes tratados con tejido de cerdo vivo. El Grupo de Estudio XEN 111". Ciencia . 285 (5431): 1236–41. doi : 10.1126 / science.285.5431.1236 . PMID 10455044 .
- ^ "Genómica Inglaterra› Patógenos integrados y dominio de elementos móviles GeCIP " . Consultado el 13 de octubre de 2019 .
- ^ Gifford, RJ; Blomberg, J; Ataúd, JM; Fan, H; Heidmann, T; Mayer, J; Stoye, J; Tristem, M; Johnson, WE (28 de agosto de 2018). "Nomenclatura para loci de retrovirus endógenos (ERV)" . Retrovirología . 15 (1): 59. doi : 10.1186 / s12977-018-0442-1 . PMC 6114882 . PMID 30153831 .
- ^ Belshaw R, Dawson AL, Woolven-Allen J, Redding J, Burt A, Tristem M (octubre de 2005). "El cribado del genoma revela altos niveles de polimorfismo de inserción en la familia de retrovirus endógenos humanos HERV-K (HML2): implicaciones para la actividad actual" . J. Virol . 79 (19): 12507–14. doi : 10.1128 / JVI.79.19.12507-12514.2005 . PMC 1211540 . PMID 16160178 .
- ^ Jha AR, Pillai SK, York VA, Sharp ER, Storm EC, Wachter DJ, Martin JN, Deeks SG, Rosenberg MG, Nixon DF, Garrison KE (agosto de 2009). "La datación transversal de nuevos haplotipos de HERV-K 113 y HERV-K 115 indican que estos provirus se originaron en África antes del Homo sapiens" . Mol Biol Evol . 26 (11): 2617–26. doi : 10.1093 / molbev / msp180 . PMC 2760466 . PMID 19666991 .
- ^ Jha AR, Nixon DF, Rosenberg MG, Martin JN, Deeks SG, Hudson RR, Garrison KE, Pillai SK (mayo de 2011). "El retrovirus endógeno humano K106 (HERV-K106) fue infeccioso después de la aparición de humanos anatómicamente modernos" . PLOS ONE . 6 (5): e20234. Código bibliográfico : 2011PLoSO ... 620234J . doi : 10.1371 / journal.pone.0020234 . PMC 3102101 . PMID 21633511 .
- ^ Bieniasz, Paul ; Lee, Young Nam (enero de 2007). "Reconstitución de un retrovirus endógeno humano infeccioso" . PLOS Pathog . 3 (1): e10. doi : 10.1371 / journal.ppat.0030010 . PMC 1781480 . PMID 17257061 .
- ^ Dewannieux M, Harper F, Richaud A, Letzelter C, Ribet D, Pierron G, Heidmann T (diciembre de 2006). "Identificación de un progenitor infeccioso para los retroelementos endógenos humanos HERV-K de copias múltiples" . Genome Res . 16 (12): 1548–56. doi : 10.1101 / gr.5565706 . PMC 1665638 . PMID 17077319 .
- ^ Chuong EB, Elde NC, Feschotte C (marzo de 2016). "Evolución reguladora de la inmunidad innata a través de la cooptación de retrovirus endógenos" . Ciencia . 351 (6277): 1083–87. Código Bibliográfico : 2016Sci ... 351.1083C . doi : 10.1126 / science.aad5497 . PMC 4887275 . PMID 26941318 .
- ^ a b Garrison KE, Jones RB, Meiklejohn DA, Anwar N, Ndhlovu LC, Chapman JM, Erickson AL, Agrawal A, Spotts G, Hecht FM, Rakoff-Nahoum S, Lenz J, Ostrowski MA, Nixon DF (noviembre de 2007) . "Respuestas de células T a retrovirus endógenos humanos en la infección por VIH-1" . PLOS Pathog . 3 (11): e165. doi : 10.1371 / journal.ppat.0030165 . PMC 2065876 . PMID 17997601 .
- ^ Morozov VA, Dao Thi VL, Denner J (2013). "La proteína transmembrana del retrovirus endógeno humano - K (HERV-K) modula la liberación de citocinas y la expresión génica" . PLOS ONE . 8 (8): e70399. Código bibliográfico : 2013PLoSO ... 870399M . doi : 10.1371 / journal.pone.0070399 . PMC 3737193 . PMID 23950929 .
- ^ Mayer J, Blomberg J, Seal RL (mayo de 2011). "Una nomenclatura revisada para loci retrovirales endógenos humanos transcritos" . ADN móvil . 2 (1): 7. doi : 10.1186 / 1759-8753-2-7 . PMC 3113919 . PMID 21542922 .
Otras lecturas
- Löwer R, Löwer J, Kurth R (mayo de 1996). "Los virus en todos nosotros: características y significado biológico de las secuencias de retrovirus endógenos humanos" . Proc Natl Acad Sci USA . 93 (11): 5177–84. Código Bibliográfico : 1996PNAS ... 93.5177L . doi : 10.1073 / pnas.93.11.5177 . PMC 39218 . PMID 8643549 .
- Molès JP, Tesniere A, Guilhou JJ (julio de 2005). “Se expresa una nueva secuencia retroviral endógena en piel de pacientes con psoriasis”. Br J Dermatol . 153 (1): 83–89. doi : 10.1111 / j.1365-2133.2005.06555.x . PMID 16029331 . S2CID 6642536 .
- Seifarth W, Frank O, Zeilfelder U, Spiess B, Greenwood AD, Hehlmann R, Leib-Mösch C (enero de 2005). "Análisis completo de la actividad transcripcional de retrovirus endógenos humanos en tejidos humanos con una micromatriz específica de retrovirus" . J. Virol . 79 (1): 341–52. doi : 10.1128 / JVI.79.1.341-352.2005 . PMC 538696 . PMID 15596828 .
- Knerr I, Beinder E, Rascher W (febrero de 2002). "Sincitina, un gen retroviral endógeno humano novedoso en la placenta humana: evidencia de su desregulación en la preeclampsia y el síndrome HELLP". Soy J Obstet Gynecol . 186 (2): 210-13. doi : 10.1067 / mob.2002.119636 . PMID 11854637 .
- Gifford R, Tristem M (mayo de 2003). "La evolución, distribución y diversidad de retrovirus endógenos" (PDF) . Genes de virus . 26 (3): 291–315. doi : 10.1023 / A: 1024455415443 . PMID 12876457 . S2CID 34639116 . Archivado desde el original (PDF) el 15 de febrero de 2013 . Consultado el 16 de enero de 2008 .
enlaces externos
- Endógenos + retrovirus en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .
- HERVd - base de datos de retrovirus endógenos humanos
- Biología Molecular
- Retrovirus endógenos
- Enfermedades porcinas