Este artículo incluye una lista de referencias generales , pero permanece en gran parte sin verificar porque carece de suficientes citas en línea correspondientes . ( Mayo de 2018 ) ( Obtenga información sobre cómo y cuándo eliminar este mensaje de plantilla ) |

La reparación de desajustes de ADN ( MMR ) es un sistema para reconocer y reparar la inserción, deleción e incorporación errónea de bases que pueden surgir durante la replicación y recombinación del ADN , así como para reparar algunas formas de daño del ADN . [1] [2]
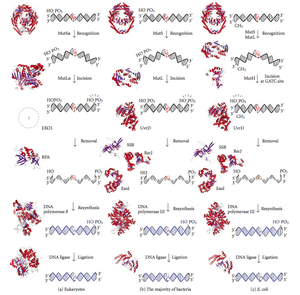
La reparación de desajustes es específica de cada hebra. Durante la síntesis de ADN, la hebra recién sintetizada (hija) comúnmente incluirá errores. Para comenzar la reparación, la maquinaria de reparación de desajustes distingue la hebra recién sintetizada de la plantilla (parental). En las bacterias gramnegativas, la hemimetilación transitoria distingue las hebras (la parental está metilada y la hija no). Sin embargo, en otros procariotas y eucariotas, el mecanismo exacto no está claro. Se sospecha que, en eucariotas, el ADN de hebra rezagada recién sintetizado contiene transitoriamente mellas(antes de ser sellado por ADN ligasa) y proporciona una señal que dirige los sistemas de corrección de errores de emparejamiento a la hebra apropiada. Esto implica que estas mellas deben estar presentes en la cadena principal, y recientemente se ha encontrado evidencia de esto. [3] Un trabajo reciente [4] ha demostrado que las muescas son sitios para la carga dependiente de RFC de la pinza deslizante de replicación PCNA, de una manera específica de orientación, de modo que una cara de la proteína en forma de rosquilla se yuxtapone hacia el 3'- OH termina en el nick. A continuación, el PCNA cargado dirige la acción de la endonucleasa MutLalpha [5] a la cadena hija en presencia de un desajuste y MutSalpha o MutSbeta.
Cualquier evento mutacional que interrumpa la estructura superhelical del ADN conlleva el potencial de comprometer la estabilidad genética de una célula. El hecho de que los sistemas de detección y reparación de daños sean tan complejos como la propia maquinaria de replicación destaca la importancia que la evolución ha atribuido a la fidelidad del ADN.
Los ejemplos de bases no emparejadas incluyen un emparejamiento G / T o A / C (ver reparación del ADN ). Los desajustes se deben comúnmente a la tautomerización de bases durante la replicación del ADN. El daño se repara reconociendo la deformidad causada por el desajuste, determinando la hebra plantilla y sin plantilla, y escindiendo la base incorporada incorrectamente y reemplazándola con el nucleótido correcto . El proceso de eliminación implica algo más que el propio nucleótido no emparejado. Se pueden eliminar unos pocos o hasta miles de pares de bases de la cadena de ADN recién sintetizada.
Proteínas de reparación no coincidentes [ editar ]
| Proteína reparadora de desajustes de ADN, dominio C-terminal | ||||||||
|---|---|---|---|---|---|---|---|---|
hpms2-atpgs | ||||||||
| Identificadores | ||||||||
| Símbolo | DNA_mis_repair | |||||||
| Pfam | PF01119 | |||||||
| Clan pfam | CL0329 | |||||||
| InterPro | IPR013507 | |||||||
| PROSITE | PDOC00057 | |||||||
| SCOP2 | 1bkn / SCOPe / SUPFAM | |||||||
| ||||||||
La reparación de desajustes es un proceso altamente conservado de procariotas a eucariotas . La primera evidencia de reparación de desajustes se obtuvo de S. pneumoniae (los genes hexA y hexB ). El trabajo posterior sobre E. coli ha identificado una serie de genes que, cuando se inactivan mutacionalmente , causan cepas hipermutables. Por lo tanto, los productos génicos se denominan proteínas "Mut" y son los principales componentes activos del sistema de reparación de errores de apareamiento. Tres de estas proteínas son esenciales para detectar el desajuste y dirigir la maquinaria de reparación a él: MutS , MutH y MutL (MutS es un homólogo de HexA y MutL de HexB).
MutS forma un dímero (MutS 2 ) que reconoce la base no emparejada en la cadena hija y se une al ADN mutado. MutH se une en sitios hemimetilados a lo largo del ADN hijo, pero su acción es latente, y se activa solo al contacto con un dímero MutL (MutL 2 ), que se une al complejo MutS-ADN y actúa como mediador entre MutS 2 y MutH, activando el último. El ADN se coloca en bucle para buscar el sitio de metilación d (GATC) más cercano al desajuste, que podría estar a una distancia de hasta 1 kb. Tras la activación por el complejo MutS-DNA, MutH corta la hebra hija cerca del sitio hemimetilado. MutL recluta helicasa UvrD (ADN Helicasa II) para separar las dos hebras con una polaridad específica de 3 'a 5'. El complejo MutSHL completo luego se desliza a lo largo del ADN en la dirección del desajuste, liberando la hebra para que sea escindida a medida que avanza. Una exonucleasa rastrea el complejo y digiere la cola del ss-DNA. La exonucleasa reclutada depende de qué lado del emparejamiento incorrecto MutH incide en la hebra: 5 'o 3'. Si la muesca hecha por MutH está en el extremo 5 'del emparejamiento incorrecto, se usa RecJ o ExoVII (ambas exonucleasas 5' a 3 '). Sin embargo, si la muesca está en el extremo 3 'del desajuste, se usa ExoI (una enzima de 3' a 5 ').
Todo el proceso termina más allá del sitio de desajuste, es decir, tanto el sitio en sí como los nucleótidos circundantes se extirpan por completo. La brecha de una sola hebra creada por la exonucleasa puede luego ser reparada por la ADN polimerasa III (asistida por la proteína de unión de una sola hebra), que usa la otra hebra como plantilla y finalmente sellada por la ADN ligasa. La ADN metilasa luego metila rápidamente la hebra hija.
Homólogos de MutS [ editar ]
Cuando se une, el dímero MutS 2 dobla la hélice de ADN y protege aproximadamente 20 pares de bases. Tiene una actividad de ATPasa débil y la unión de ATP conduce a la formación de estructuras terciarias en la superficie de la molécula. La estructura cristalina de MutS revela que es excepcionalmente asimétrica y, aunque su conformación activa es un dímero, solo una de las dos mitades interactúa con el sitio de desajuste.
En eucariotas, M ut S h omologs forman dos heterodímeros principales: MSH2 / MSH6 (MutSα) y MSH2 / Msh3 (MutSβ). La vía MutSα participa principalmente en la sustitución de bases y la reparación de errores de apareamiento de bucles pequeños. La vía MutSβ también participa en la reparación de bucle pequeño, además de la reparación de bucle grande (~ 10 bucles de nucleótidos). Sin embargo, MutSβ no repara las sustituciones de bases.
Homólogos MutL [ editar ]
MutL también tiene una actividad de ATPasa débil (utiliza ATP para fines de movimiento). Forma un complejo con MutS y MutH, aumentando la huella de MutS en el ADN.
Sin embargo, la procesividad (la distancia que la enzima puede moverse a lo largo del ADN antes de disociarse) de UvrD es sólo de ~ 40 a 50 pb. Debido a que la distancia entre la muesca creada por MutH y el desajuste puede promediar ~ 600 bp, si no hay otro UvrD cargado, la sección desenrollada puede volver a recocerse con su hebra complementaria, lo que obliga al proceso a comenzar de nuevo. Sin embargo, cuando está asistido por MutL, la tasa de carga de UvrD aumenta considerablemente. Si bien la procesividad (y la utilización de ATP) de las moléculas de UvrD individuales sigue siendo la misma, el efecto total sobre el ADN aumenta considerablemente; el ADN no tiene posibilidad de reasociarse, ya que cada UvrD desenrolla 40-50 pb de ADN, se disocia y luego es reemplazado inmediatamente por otro UvrD, repitiendo el proceso. Esto expone grandes secciones de ADN a la exonucleasa.digestión, lo que permite la escisión rápida (y posterior reemplazo) del ADN incorrecto.
Los eucariotas tienen cinco M ut L h omologs designados como MLH1, MLH2, MLH3, PMS1 y PMS2. Forman heterodímeros que imitan MutL en E. coli . Los homólogos humanos de MutL procariótico forman tres complejos denominados MutLα, MutLβ y MutLγ. El complejo MutLα está formado por subunidades MLH1 y PMS2, el heterodímero MutLβ está formado por MLH1 y PMS1, mientras que MutLγ está formado por MLH1 y MLH3. MutLα actúa como una endonucleasa que introduce roturas de hebra en la hebra hija tras la activación por desajuste y otras proteínas necesarias, MutSα y PCNA. Estas interrupciones de la cadena sirven como puntos de entrada para una actividad exonucleasa que elimina el ADN no emparejado. Los roles que desempeñan MutLβ y MutLγ en la reparación de errores de apareamiento se comprenden menos.
MutH: una endonucleasa presente en E. coli y Salmonella [ editar ]
MutH es una endonucleasa muy débil que se activa una vez que se une a MutL (que a su vez está unida a MutS). Se mellas no metilada de ADN y la hebra no metilada de ADN hemimethylated pero no nick metilado totalmente ADN. Los experimentos han demostrado que la reparación de los desajustes es aleatoria si ninguna de las hebras está metilada. [ cita requerida ] Estos comportamientos llevaron a la propuesta de que MutH determina qué hebra contiene el desajuste. MutH no tiene homólogo eucariota. Su función de endonucleasa es asumida por homólogos de MutL, que tienen alguna actividad de exonucleasa 5'-3 'especializada. El sesgo de la hebra para eliminar los desajustes de la hebra hija recién sintetizada en eucariotas puede ser proporcionada por los extremos libres 3 'de los fragmentos de Okazaki. en la nueva hebra creada durante la replicación.
Abrazadera deslizante PCNA β [ editar ]
PCNA y la pinza deslizante β se asocian con MutSα / β y MutS, respectivamente. Aunque los informes iniciales sugirieron que el complejo PCNA-MutSα puede mejorar el reconocimiento de desajustes, [6] se ha demostrado recientemente [7] que no hay un cambio aparente en la afinidad de MutSα por un desajuste en presencia o ausencia de PCNA. Además, los mutantes de MutSα que no pueden interactuar con PCNA in vitro exhiben la capacidad de llevar a cabo el reconocimiento de errores de apareamiento y la escisión de los mismos hasta niveles cercanos al tipo salvaje. Dichos mutantes son defectuosos en la reacción de reparación dirigida por una rotura de la hebra 5 ', lo que sugiere por primera vez la función MutSα en una etapa posterior a la escisión de la reacción.
Importancia clínica [ editar ]
Defectos heredados en la reparación de discrepancias [ editar ]
Las mutaciones en los homólogos humanos de las proteínas Mut afectan la estabilidad genómica, lo que puede resultar en inestabilidad de microsatélites (MSI), implicada en algunos cánceres humanos. En concreto, los cánceres colorrectales hereditarios sin poliposis ( HNPCC o síndrome de Lynch) se atribuyen a variantes dañinas de la línea germinal en los genes que codifican los homólogos de MutS y MutL MSH2 y MLH1 respectivamente, que por tanto se clasifican como genes supresores de tumores. Un subtipo de HNPCC, el síndrome de Muir-Torre (MTS), está asociado con tumores de piel. Si ambas copias heredadas (alelos) de un gen MMR tienen variantes genéticas dañinas, esto da como resultado una afección muy rara y grave: el síndrome del cáncer de reparación de desajustes.(o deficiencia constitucional de reparación de desajustes, CMMR-D), que se manifiesta como múltiples apariciones de tumores a una edad temprana, a menudo tumores de colon y cerebro . [8]
Silenciamiento epigenético de genes de reparación de desajustes [ editar ]
Los cánceres esporádicos con una deficiencia en la reparación del ADN rara vez tienen una mutación en un gen de reparación del ADN, pero en cambio tienden a tener alteraciones epigenéticas como la metilación del promotor que inhibe la expresión del gen de reparación del ADN. [9] Aproximadamente el 13% de los cánceres colorrectales son deficientes en la reparación de errores de emparejamiento del ADN, comúnmente debido a la pérdida de MLH1 (9,8%), o algunas veces MSH2, MSH6 o PMS2 (todos ≤ 1,5%). [10] Para la mayoría de los cánceres colorrectales esporádicos deficientes en MLH1, la deficiencia se debió a la metilación del promotor MLH1. [10] Otros tipos de cáncer tienen frecuencias más altas de pérdida de MLH1 (ver tabla a continuación), que nuevamente son en gran parte resultado de la metilación del promotor de MLH1. gene. A different epigenetic mechanism underlying MMR deficiencies might involve over-expression of a microRNA, for example miR-155 levels inversely correlate with expression of MLH1 or MSH2 in colorectal cancer.[11]
| Cancer type | Frequency of deficiency in cancer | Frequency of deficiency in adjacent field defect |
|---|---|---|
| Stomach | 32%[12][13] | 24%-28% |
| Stomach (foveolar type tumors) | 74%[14] | 71% |
| Stomach in high-incidence Kashmir Valley | 73%[15] | 20% |
| Esophageal | 73%[16] | 27% |
| Head and neck squamous cell carcinoma (HNSCC) | 31%-33%[17][18] | 20%-25% |
| Non-small cell lung cancer (NSCLC) | 69%[19] | 72% |
| Colorectal | 10%[10] |
MMR failures in field defects[edit]
A field defect (field cancerization) is an area of epithelium that has been preconditioned by epigenetic or genetic changes, predisposing it towards development of cancer. As pointed out by Rubin " ...there is evidence that more than 80% of the somatic mutations found in mutator phenotype human colorectal tumors occur before the onset of terminal clonal expansion."[20][21] Similarly, Vogelstein et al.[22] point out that more than half of somatic mutations identified in tumors occurred in a pre-neoplastic phase (in a field defect), during growth of apparently normal cells.
MLH1 deficiencies were common in the field defects (histologically normal tissues) surrounding tumors; see Table above. Epigenetically silenced or mutated MLH1 would likely not confer a selective advantage upon a stem cell, however, it would cause increased mutation rates, and one or more of the mutated genes may provide the cell with a selective advantage. The deficientMLH1 gene could then be carried along as a selectively near-neutral passenger (hitch-hiker) gene when the mutated stem cell generates an expanded clone. The continued presence of a clone with an epigenetically repressed MLH1 would continue to generate further mutations, some of which could produce a tumor.
MMR components in humans[edit]
In humans, seven DNA mismatch repair (MMR) proteins (MLH1, MLH3, MSH2, MSH3, MSH6, PMS1 and PMS2) work coordinately in sequential steps to initiate repair of DNA mismatches.[23] In addition, there are Exo1-dependent and Exo1-independent MMR subpathways.[24]
Other gene products involved in mismatch repair (subsequent to initiation by MMR genes) in humans include DNA polymerase delta, PCNA, RPA, HMGB1, RFC and DNA ligase I, plus histone and chromatin modifying factors.[25][26]
In certain circumstances, the MMR pathway may recruit an error-prone DNA polymerase eta (POLH). This happens in B-lymphocytes during somatic hypermutation, where POLH is used to introduce genetic variation into antibody genes.[27] However, this error-prone MMR pathway may be triggered in other types of human cells upon exposure to genotoxins [28] and indeed it is broadly active in various human cancers, causing mutations that bear a signature of POLH activity.[29]
MMR and mutation frequency[edit]
Recognizing and repairing mismatches and indels is important for cells because failure to do so results in microsatellite instability (MSI) and an elevated spontaneous mutation rate (mutator phenotype). In comparison to other cancer types, MMR-deficient (MSI) cancer has a very high frequency of mutations, close to melanoma and lung cancer,[30] cancer types caused by much exposure to UV radiation and mutagenic chemicals.
In addition to a very high mutation burden, MMR deficiencies result in an unusual distribution of somatic mutations across the human genome: this suggests that MMR preferentially protects the gene-rich, early-replicating euchromatic regions.[31] In contrast, the gene-poor, late-replicating heterochromatic genome regions exhibit high mutation rates in many human tumors.[32]
The histone modification H3K36me3, an epigenetic mark of active chromatin, has the ability to recruit the MSH2-MSH6 (hMutSα) complex.[33] Consistently, regions of the human genome with high levels of H3K36me3 accumulate less mutations due to MMR activity.[29]
Loss of multiple DNA repair pathways in tumors[edit]
Lack of MMR often occurs in coordination with loss of other DNA repair genes.[9] For example, MMR genes MLH1 and MLH3 as well as 11 other DNA repair genes (such as MGMT and many NER pathway genes) were significantly down-regulated in lower grade as well as in higher grade astrocytomas, in contrast to normal brain tissue.[34] Moreover, MLH1 and MGMT expression was closely correlated in 135 specimens of gastric cancer and loss of MLH1 and MGMT appeared to be synchronously accelerated during tumor progression.[35]
Deficient expression of multiple DNA repair genes is often found in cancers,[9] and may contribute to the thousands of mutations usually found in cancers (see Mutation frequencies in cancers).
See also[edit]
- Base excision repair
- Nucleotide excision repair
References[edit]
- ^ Iyer RR, Pluciennik A, Burdett V, Modrich PL (February 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- ^ Larrea AA, Lujan SA, Kunkel TA (May 2010). "SnapShot: DNA mismatch repair". Cell. 141 (4): 730–730.e1. doi:10.1016/j.cell.2010.05.002. PMID 20478261. S2CID 26969788.
- ^ Heller RC, Marians KJ (December 2006). "Replisome assembly and the direct restart of stalled replication forks". Nature Reviews. Molecular Cell Biology. 7 (12): 932–43. doi:10.1038/nrm2058. PMID 17139333. S2CID 27666329.
- ^ Pluciennik A, Dzantiev L, Iyer RR, Constantin N, Kadyrov FA, Modrich P (September 2010). "PCNA function in the activation and strand direction of MutLα endonuclease in mismatch repair". Proceedings of the National Academy of Sciences of the United States of America. 107 (37): 16066–71. doi:10.1073/pnas.1010662107. PMC 2941292. PMID 20713735.
- ^ Kadyrov FA, Dzantiev L, Constantin N, Modrich P (July 2006). "Endonucleolytic function of MutLalpha in human mismatch repair". Cell. 126 (2): 297–308. doi:10.1016/j.cell.2006.05.039. PMID 16873062. S2CID 15643051.
- ^ Flores-Rozas H, Clark D, Kolodner RD (November 2000). "Proliferating cell nuclear antigen and Msh2p-Msh6p interact to form an active mispair recognition complex". Nature Genetics. 26 (3): 375–8. doi:10.1038/81708. PMID 11062484. S2CID 20861705.
- ^ Iyer RR, Pohlhaus TJ, Chen S, Hura GL, Dzantiev L, Beese LS, Modrich P (May 2008). "The MutSalpha-proliferating cell nuclear antigen interaction in human DNA mismatch repair". The Journal of Biological Chemistry. 283 (19): 13310–9. doi:10.1074/jbc.M800606200. PMC 2423938. PMID 18326858.
- ^ Online Mendelian Inheritance in Man (OMIM): 276300
- ^ a b c Bernstein C, Bernstein H (May 2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ a b c Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, et al. (May 2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, et al. (April 2010). "Modulation of mismatch repair and genomic stability by miR-155". Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–7. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ Kupčinskaitė-Noreikienė R, Skiecevičienė J, Jonaitis L, Ugenskienė R, Kupčinskas J, Markelis R, et al. (2013). "CpG island methylation of the MLH1, MGMT, DAPK, and CASP8 genes in cancerous and adjacent noncancerous stomach tissues". Medicina. 49 (8): 361–6. doi:10.3390/medicina49080056. PMID 24509146.
- ^ Waki T, Tamura G, Tsuchiya T, Sato K, Nishizuka S, Motoyama T (August 2002). "Promoter methylation status of E-cadherin, hMLH1, and p16 genes in nonneoplastic gastric epithelia". The American Journal of Pathology. 161 (2): 399–403. doi:10.1016/S0002-9440(10)64195-8. PMC 1850716. PMID 12163364.
- ^ Endoh Y, Tamura G, Ajioka Y, Watanabe H, Motoyama T (September 2000). "Frequent hypermethylation of the hMLH1 gene promoter in differentiated-type tumors of the stomach with the gastric foveolar phenotype". The American Journal of Pathology. 157 (3): 717–22. doi:10.1016/S0002-9440(10)64584-1. PMC 1949419. PMID 10980110.
- ^ Wani M, Afroze D, Makhdoomi M, Hamid I, Wani B, Bhat G, et al. (2012). "Promoter methylation status of DNA repair gene (hMLH1) in gastric carcinoma patients of the Kashmir valley" (PDF). Asian Pacific Journal of Cancer Prevention. 13 (8): 4177–81. doi:10.7314/apjcp.2012.13.8.4177. PMID 23098428.
- ^ Chang Z, Zhang W, Chang Z, Song M, Qin Y, Chang F, et al. (January 2015). "Expression characteristics of FHIT, p53, BRCA2 and MLH1 in families with a history of oesophageal cancer in a region with a high incidence of oesophageal cancer". Oncology Letters. 9 (1): 430–436. doi:10.3892/ol.2014.2682. PMC 4246613. PMID 25436004.
- ^ Tawfik HM, El-Maqsoud NM, Hak BH, El-Sherbiny YM (2011). "Head and neck squamous cell carcinoma: mismatch repair immunohistochemistry and promoter hypermethylation of hMLH1 gene". American Journal of Otolaryngology. 32 (6): 528–36. doi:10.1016/j.amjoto.2010.11.005. PMID 21353335.
- ^ Zuo C, Zhang H, Spencer HJ, Vural E, Suen JY, Schichman SA, et al. (October 2009). "Increased microsatellite instability and epigenetic inactivation of the hMLH1 gene in head and neck squamous cell carcinoma". Otolaryngology–Head and Neck Surgery. 141 (4): 484–90. doi:10.1016/j.otohns.2009.07.007. PMID 19786217. S2CID 8357370.
- ^ Safar AM, Spencer H, Su X, Coffey M, Cooney CA, Ratnasinghe LD, et al. (June 2005). "Methylation profiling of archived non-small cell lung cancer: a promising prognostic system". Clinical Cancer Research. 11 (12): 4400–5. doi:10.1158/1078-0432.CCR-04-2378. PMID 15958624.
- ^ Rubin H (March 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays. 33 (3): 224–31. doi:10.1002/bies.201000067. PMID 21254148. S2CID 44981539.
- ^ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, et al. (February 2000). "Genetic reconstruction of individual colorectal tumor histories". Proceedings of the National Academy of Sciences of the United States of America. 97 (3): 1236–41. Bibcode:2000PNAS...97.1236T. doi:10.1073/pnas.97.3.1236. PMC 15581. PMID 10655514.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ Pal T, Permuth-Wey J, Sellers TA (August 2008). "A review of the clinical relevance of mismatch-repair deficiency in ovarian cancer". Cancer. 113 (4): 733–42. doi:10.1002/cncr.23601. PMC 2644411. PMID 18543306.
- ^ Goellner EM, Putnam CD, Kolodner RD (August 2015). "Exonuclease 1-dependent and independent mismatch repair". DNA Repair. 32: 24–32. doi:10.1016/j.dnarep.2015.04.010. PMC 4522362. PMID 25956862.
- ^ Li GM (January 2008). "Mechanisms and functions of DNA mismatch repair". Cell Research. 18 (1): 85–98. doi:10.1038/cr.2007.115. PMID 18157157.
- ^ Li GM (July 2014). "New insights and challenges in mismatch repair: getting over the chromatin hurdle". DNA Repair. 19: 48–54. doi:10.1016/j.dnarep.2014.03.027. PMC 4127414. PMID 24767944.
- ^ Chahwan R, Edelmann W, Scharff MD, Roa S (August 2012). "AIDing antibody diversity by error-prone mismatch repair". Seminars in Immunology. 24 (4): 293–300. doi:10.1016/j.smim.2012.05.005. PMC 3422444. PMID 22703640.
- ^ Hsieh P (September 2012). "DNA mismatch repair: Dr. Jekyll and Mr. Hyde?". Molecular Cell. 47 (5): 665–6. doi:10.1016/j.molcel.2012.08.020. PMC 3457060. PMID 22980456.
- ^ a b Supek F, Lehner B (July 2017). "Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes". Cell. 170 (3): 534–547.e23. doi:10.1016/j.cell.2017.07.003. hdl:10230/35343. PMID 28753428.
- ^ Tuna M, Amos CI (November 2013). "Genomic sequencing in cancer". Cancer Letters. 340 (2): 161–70. doi:10.1016/j.canlet.2012.11.004. PMC 3622788. PMID 23178448.
- ^ Supek F, Lehner B (May 2015). "Differential DNA mismatch repair underlies mutation rate variation across the human genome". Nature. 521 (7550): 81–4. Bibcode:2015Natur.521...81S. doi:10.1038/nature14173. PMC 4425546. PMID 25707793.
- ^ Schuster-Böckler B, Lehner B (August 2012). "Chromatin organization is a major influence on regional mutation rates in human cancer cells". Nature. 488 (7412): 504–7. Bibcode:2012Natur.488..504S. doi:10.1038/nature11273. PMID 22820252. S2CID 205229634.
- ^ Li F, Mao G, Tong D, Huang J, Gu L, Yang W, Li GM (April 2013). "The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα". Cell. 153 (3): 590–600. doi:10.1016/j.cell.2013.03.025. PMC 3641580. PMID 23622243.
- ^ Jiang Z, Hu J, Li X, Jiang Y, Zhou W, Lu D (December 2006). "Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array". Neuroscience Letters. 409 (2): 112–7. doi:10.1016/j.neulet.2006.09.038. PMID 17034947.
- ^ Kitajima Y, Miyazaki K, Matsukura S, Tanaka M, Sekiguchi M (2003). "Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer". Gastric Cancer. 6 (2): 86–95. doi:10.1007/s10120-003-0213-z. PMID 12861399.
Further reading[edit]
- Hsieh P, Yamane K (2008). "DNA mismatch repair: molecular mechanism, cancer, and ageing". Mechanisms of Ageing and Development. 129 (7–8): 391–407. doi:10.1016/j.mad.2008.02.012. PMC 2574955. PMID 18406444.
- Iyer RR, Pluciennik A, Burdett V, Modrich PL (February 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- Joseph N, Duppatla V, Rao DN (2006). Prokaryotic DNA mismatch repair. Progress in Nucleic Acid Research and Molecular Biology. 81. pp. 1–49. doi:10.1016/S0079-6603(06)81001-9. ISBN 9780125400817. PMID 16891168.
- Yang W (August 2000). "Structure and function of mismatch repair proteins". Mutation Research. 460 (3–4): 245–56. doi:10.1016/s0921-8777(00)00030-6. PMID 10946232.
- Griffiths JF, Gilbert WM, Lewontin RC, Wessler SR, Suzuki DT, Miller JH (2004). An introduction to genetic analysis (8th ed.). New York, NY: Freeman. ISBN 978-0-7167-4939-4.
- Kunkel TA, Erie DA (2005). "DNA mismatch repair". Annual Review of Biochemistry. 74: 681–710. doi:10.1146/annurev.biochem.74.082803.133243. PMID 15952900.
- Friedberg EC, Walker GC, Siede W, Wood RD, Schultz RA, Ellenberger (2005). DNA repair and mutagenesis (2nd ed.). Washington, D.C.: ASM Press. ISBN 978-1-55581-319-2.
External links[edit]
- DNA Repair
- DNA+Mismatch+Repair at the US National Library of Medicine Medical Subject Headings (MeSH)