Catálisis enzimática
La catálisis enzimática es el aumento de la velocidad de un proceso mediante una molécula biológica , una " enzima ". La mayoría de las enzimas son proteínas y la mayoría de estos procesos son reacciones químicas. Dentro de la enzima, generalmente la catálisis ocurre en un sitio localizado, llamado sitio activo .
La mayoría de las enzimas están formadas predominantemente por proteínas, ya sea una sola cadena de proteínas o muchas de esas cadenas en un complejo de múltiples subunidades . Las enzimas a menudo también incorporan componentes no proteicos, como iones metálicos o moléculas orgánicas especializadas conocidas como cofactor (por ejemplo, trifosfato de adenosina ). Muchos cofactores son vitaminas y su función como vitaminas está directamente relacionada con su uso en la catálisis del proceso biológico dentro del metabolismo. Catálisis de reacciones bioquímicas en la célula.es vital ya que muchas, pero no todas las reacciones metabólicamente esenciales, tienen tasas muy bajas cuando no están catalizadas. Un impulsor de la evolución de las proteínas es la optimización de tales actividades catalíticas, aunque solo las enzimas más cruciales operan cerca de los límites de eficiencia catalítica, y muchas enzimas están lejos de ser óptimas. Los factores importantes en la catálisis enzimática incluyen catálisis ácida y básica general , dirección orbital, restricción entrópica, efectos de orientación (es decir, catálisis de cerradura y llave), así como efectos de movimiento que involucran la dinámica de proteínas [1]
Los mecanismos de la catálisis enzimática varían, pero todos son similares en principio a otros tipos de catálisis química en que el factor crucial es una reducción de la (s) barrera (s) de energía que separan los reactivos (o sustratos de los productos). La reducción de la energía de activación ( E a) aumenta la fracción de moléculas reactivas que pueden superar esta barrera y formar el producto. Un principio importante es que, dado que solo reducen las barreras de energía entre los productos y los reactivos, las enzimas siempre catalizan reacciones en ambas direcciones y no pueden impulsar una reacción ni afectar la posición de equilibrio, solo la velocidad con la que se logra. Al igual que con otros catalizadores, la reacción no consume ni cambia la enzima (como un sustrato), sino que se recicla de manera que una sola enzima realice muchas rondas de catálisis.
Las enzimas suelen ser muy específicas y actúan solo sobre determinados sustratos. Algunas enzimas son absolutamente específicas, lo que significa que actúan sobre un solo sustrato, mientras que otras muestran especificidad de grupo y pueden actuar sobre grupos químicos similares pero no idénticos, como el enlace peptídico en diferentes moléculas. Muchas enzimas tienen especificidad estereoquímica y actúan sobre un estereoisómero pero no sobre otro. [2]
Ajuste inducido
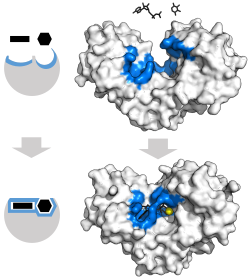
El modelo clásico para la interacción enzima- sustrato es el modelo de ajuste inducido. [3] Este modelo propone que la interacción inicial entre la enzima y el sustrato es relativamente débil, pero que estas interacciones débiles inducen rápidamente cambios conformacionales en la enzima que fortalecen la unión.
Las ventajas del mecanismo de ajuste inducido surgen debido al efecto estabilizador de una fuerte unión a la enzima. Hay dos mecanismos diferentes de unión al sustrato: unión uniforme, que tiene una fuerte unión al sustrato, y unión diferencial, que tiene una fuerte unión en estado de transición. El efecto estabilizador de la unión uniforme aumenta tanto la afinidad de unión del sustrato como del estado de transición, mientras que la unión diferencial aumenta solo la afinidad de unión del estado de transición. Ambos son utilizados por enzimas y han sido elegidos evolutivamente para minimizar la energía de activación de la reacción. Las enzimas que están saturadas, es decir, tienen una unión de sustrato de alta afinidad, requieren unión diferencial para reducir la energía de activación, mientras que las enzimas de sustrato pequeño no unidas pueden usar unión diferencial o uniforme. [4]
Estos efectos han llevado a la mayoría de las proteínas a utilizar el mecanismo de unión diferencial para reducir la energía de activación, por lo que la mayoría de los sustratos tienen una alta afinidad por la enzima mientras se encuentran en el estado de transición. La unión diferencial se lleva a cabo mediante el mecanismo de ajuste inducido: el sustrato primero se une débilmente, luego la enzima cambia de conformación aumentando la afinidad al estado de transición y estabilizándolo, reduciendo así la energía de activación para alcanzarlo.
Sin embargo, es importante aclarar que el concepto de ajuste inducido no se puede utilizar para racionalizar la catálisis. Es decir, la catálisis química se define como la reducción de E a ‡ (cuando el sistema ya está en el ES ‡ ) en relación con E a ‡ en la reacción no catalizada en agua (sin la enzima). El ajuste inducido solo sugiere que la barrera es más baja en la forma cerrada de la enzima, pero no nos dice cuál es la razón de la reducción de la barrera.
El ajuste inducido puede ser beneficioso para la fidelidad del reconocimiento molecular en presencia de competencia y ruido a través del mecanismo de corrección de pruebas conformacional . [5]
Mecanismos de una ruta de reacción alternativa.
Estos cambios conformacionales también acercan los residuos catalíticos en el sitio activo a los enlaces químicos en el sustrato que serán alterados en la reacción. Una vez que tiene lugar la unión, uno o más mecanismos de catálisis reducen la energía del estado de transición de la reacción , proporcionando una vía química alternativa para la reacción. Hay seis posibles mecanismos de catálisis "sobre la barrera", así como un mecanismo "a través de la barrera":
Proximidad y orientación
Las interacciones enzima-sustrato alinean los grupos químicos reactivos y los mantienen juntos en una geometría óptima, lo que aumenta la velocidad de la reacción. Esto reduce la entropíade los reactivos y, por lo tanto, hace que las reacciones de adición o transferencia sean menos desfavorables, ya que una reducción en la entropía general cuando dos reactivos se convierten en un solo producto. Sin embargo, este es un efecto general y se observa en reacciones de no adición o transferencia donde ocurre debido a un aumento en la "concentración efectiva" de los reactivos. Esto se entiende al considerar cómo los aumentos de concentración conducen a aumentos en la velocidad de reacción: esencialmente, cuando los reactivos están más concentrados, chocan con más frecuencia y, por lo tanto, reaccionan con más frecuencia. En la catálisis enzimática, la unión de los reactivos a la enzima restringe el espacio conformacional de los reactivos, manteniéndolos en la 'orientación adecuada' y cerca unos de otros, de modo que colisionen con mayor frecuencia, y con la geometría correcta, para facilitar la reacción deseada. Los "concentración efectiva "es la concentración que el reactivo tendría que estar, libre en solución, para experimentar la misma frecuencia de colisión. A menudo, tales concentraciones efectivas teóricas no son físicas e imposibles de realizar en la realidad, lo cual es un testimonio del gran poder catalítico de muchas enzimas. , con incrementos masivos de la tasa sobre el estado no catalizado.
| Por ejemplo: |
| Se producirán reacciones similares mucho más rápido si la reacción es intramolecular. |
| La concentración efectiva de acetato en la reacción intramolecular se puede estimar como k 2 / k 1 = 2 x 10 5 Molar. |
Sin embargo, la situación podría ser más compleja, ya que los estudios computacionales modernos han establecido que los ejemplos tradicionales de efectos de proximidad no pueden relacionarse directamente con los efectos entrópicos de las enzimas. [6] [7] [8] Además, se ha descubierto que la propuesta entrópica original [9] sobreestima en gran medida la contribución de la entropía de orientación a la catálisis. [10]
Donantes o aceptores de protones
Donantes y aceptores de protones, es decir, ácidos y basespuede donar y aceptar protones para estabilizar las cargas en desarrollo en el estado de transición. Esto está relacionado con el principio general de la catálisis, el de reducir las barreras energéticas, ya que en general los estados de transición son estados de alta energía, y al estabilizarlos se reduce esta alta energía, bajando la barrera. Una característica clave de la catálisis enzimática sobre muchas catálisis no biológica es que tanto la catálisis ácida como la básica se pueden combinar en la misma reacción. En muchos sistemas abióticos, los ácidos ([H +] grandes) o las bases (sumideros de H + de gran concentración, o especies con pares de electrones) pueden aumentar la velocidad de la reacción; pero, por supuesto, el medio ambiente solo puede tener un pH general (medida de acidez o basicidad (alcalinidad)). Sin embargo, dado que las enzimas son moléculas grandes,pueden colocar tanto los grupos ácidos como los grupos básicos en su sitio activo para interactuar con sus sustratos y emplear ambos modos independientemente del pH total.
A menudo se emplea catálisis ácida o básica general para activar grupos nucleófilos y / o electrófilos , o para estabilizar grupos salientes. En el sitio activo se emplean muchos aminoácidos con grupos ácidos o básicos, como el ácido glutámico y aspártico, histidina, cistina, tirosina, lisina y arginina, así como serina y treonina. Además, a menudo se emplea la cadena principal peptídica, con grupos N carbonilo y amida. La cistina y la histidina están involucradas con mucha frecuencia, ya que ambas tienen un pKa cercano al pH neutro y, por lo tanto, pueden aceptar y donar protones.
Muchos mecanismos de reacción que involucran catálisis ácido / base asumen un pKa sustancialmente alterado. Esta alteración de pKa es posible a través del entorno local del residuo [ cita requerida ] .
| Condiciones | Ácidos | Bases |
|---|---|---|
| Entorno hidrofóbico | Incrementar pKa | Disminuir pKa |
| Residuos adyacentes de carga similar | Incrementar pKa | Disminuir pKa |
Formación de puentes salinos (y puentes de hidrógeno ) | Disminuir pKa | Incrementar pKa |
El pKa también puede verse afectado significativamente por el entorno circundante, en la medida en que los residuos que son básicos en solución pueden actuar como donantes de protones y viceversa.
| Por ejemplo: |
| Tríada catalítica de una serina proteasa |
| El paso inicial del mecanismo catalítico de la serina proteasa implica que la histidina del sitio activo acepte un protón del residuo de serina. Esto prepara a la serina como nucleófilo para atacar el enlace amida del sustrato. Este mecanismo incluye la donación de un protón de serina (una base, pKa 14) a histidina (un ácido, pKa 6), posible gracias al entorno local de las bases. |
Es importante aclarar que la modificación de los pKa es una parte pura del mecanismo electrostático. [11] Además, el efecto catalítico del ejemplo anterior se asocia principalmente con la reducción del pKa del oxianión y el aumento del pKa de la histidina, mientras que la transferencia de protones de la serina a la histidina no se cataliza significativamente, ya que no es la barrera que determina la tasa. [12] Nótese que en el ejemplo mostrado, el ácido conjugado de histidina actúa como un catalizador ácido general para la posterior pérdida de la amina de un intermedio tetraédrico. Sin embargo, la evidencia que apoya este mecanismo propuesto (Figura 4 en la Ref. 13) [13] ha sido controvertida. [14]
Catálisis electrostática
La estabilización de los estados de transición cargados también puede ser por residuos en el sitio activo que forman enlaces iónicos (o interacciones de carga iónica parcial) con el intermedio. Estos enlaces pueden provenir de cadenas laterales ácidas o básicas que se encuentran en aminoácidos como lisina , arginina , ácido aspártico o ácido glutámico o provenir de cofactores metálicos como el zinc . Los iones metálicos son particularmente efectivos y pueden reducir el pKa del agua lo suficiente como para convertirlo en un nucleófilo efectivo.
Los estudios sistemáticos de simulación por computadora establecieron que los efectos electrostáticos son, con mucho, la mayor contribución a la catálisis. [11] Puede aumentar la velocidad de reacción en un factor de hasta 10 7 . [15] En particular, se ha descubierto que la enzima proporciona un entorno que es más polar que el agua y que los estados de transición iónicos se estabilizan mediante dipolos fijos. Esto es muy diferente de la estabilización del estado de transición en el agua, donde las moléculas de agua deben pagar con "energía de reorganización". [16] Para estabilizar estados iónicos y cargados. Por lo tanto, la catálisis está asociada con el hecho de que los grupos polares de la enzima están preorganizados [17].
Se ha demostrado que la magnitud del campo electrostático ejercido por el sitio activo de una enzima está altamente correlacionada con la mejora de la velocidad catalítica de la enzima [18] [19]
La unión del sustrato normalmente excluye el agua del sitio activo, reduciendo así la constante dieléctrica local a la de un disolvente orgánico. Esto refuerza las interacciones electrostáticas entre los sustratos cargados / polares y los sitios activos. Además, los estudios han demostrado que las distribuciones de carga alrededor de los sitios activos están organizadas para estabilizar los estados de transición de las reacciones catalizadas. En varias enzimas, estas distribuciones de carga aparentemente sirven para guiar a los sustratos polares hacia sus sitios de unión, de modo que las velocidades de estas reacciones enzimáticas son mayores que sus límites aparentes controlados por difusión [ cita requerida ] .
| Por ejemplo: |
| Mecanismo catalítico de carboxipeptidasa |
| El intermedio tetraédrico se estabiliza mediante un enlace iónico parcial entre el ion Zn 2+ y la carga negativa del oxígeno. |
Catálisis covalente
La catálisis covalente implica que el sustrato forme un enlace covalente transitorio con residuos en el sitio activo de la enzima o con un cofactor. Esto agrega un intermedio covalente adicional a la reacción y ayuda a reducir la energía de los estados de transición posteriores de la reacción. El enlace covalente debe, en una etapa posterior de la reacción, romperse para regenerar la enzima. Este mecanismo es utilizado por la tríada catalítica de enzimas como proteasas como quimotripsina y tripsina , donde se forma un intermedio acil-enzima. Un mecanismo alternativo es la formación de bases de Schiff utilizando la amina libre de un residuo de lisina , como se ve en la enzima.aldolasa durante la glucólisis .
Algunas enzimas utilizan cofactores que no son aminoácidos , como el fosfato de piridoxal (PLP) o el pirofosfato de tiamina (TPP) para formar intermedios covalentes con moléculas reactivas. [20] [21] Dichos intermedios covalentes funcionan para reducir la energía de los estados de transición posteriores, de manera similar a cómo los intermedios covalentes formados con los residuos de aminoácidos del sitio activo permiten la estabilización, pero las capacidades de los cofactores permiten que las enzimas lleven a cabo reacciones que los residuos del lado de los aminoácidos por sí solos. no pude. Las enzimas que utilizan tales cofactores incluyen la enzima aspartato transaminasa dependiente de PLP y la enzima piruvato deshidrogenasa dependiente de TPP . [22] [23]
En lugar de reducir la energía de activación para una vía de reacción, la catálisis covalente proporciona una vía alternativa para la reacción (a través del intermedio covalente) y, por lo tanto, es distinta de la verdadera catálisis. [11] Por ejemplo, la energía del enlace covalente a la molécula de serina en la quimotripsina debe compararse con el enlace covalente bien entendido al nucleófilo en la reacción en solución no catalizada. Una verdadera propuesta de catálisis covalente (donde la barrera es más baja que la barrera correspondiente en solución) requeriría, por ejemplo, un enlace covalente parcial al estado de transición por un grupo enzimático (por ejemplo, un enlace de hidrógeno muy fuerte), y tal los efectos no contribuyen significativamente a la catálisis.
Catálisis de iones metálicos
Un ión metálico en el sitio activo participa en la catálisis coordinando la estabilización de carga y el blindaje. Debido a la carga positiva de un metal, solo las cargas negativas pueden estabilizarse mediante iones metálicos. [24] Sin embargo, los iones metálicos son ventajosos en la catálisis biológica porque no se ven afectados por los cambios en el pH. [25] Los iones metálicos también pueden actuar para ionizar el agua actuando como un ácido de Lewis . [26] Los iones metálicos también pueden ser agentes de oxidación y reducción. [27]
Deformación de enlace
Este es el efecto principal de la unión por ajuste inducido, donde la afinidad de la enzima por el estado de transición es mayor que por el sustrato mismo. Esto induce reordenamientos estructurales que tensan los enlaces del sustrato en una posición más cercana a la conformación del estado de transición, reduciendo así la diferencia de energía entre el sustrato y el estado de transición y ayudando a catalizar la reacción.
Sin embargo, el efecto de deformación es, de hecho, un efecto de desestabilización del estado fundamental, más que un efecto de estabilización del estado de transición. [11] [28] [ página necesaria ] Además, las enzimas son muy flexibles y no pueden aplicar un gran efecto de tensión. [29]
Además de la tensión de unión en el sustrato, la tensión de unión también puede inducirse dentro de la propia enzima para activar residuos en el sitio activo.
| Por ejemplo: |
| Conformaciones de sustrato, sustrato unido y estado de transición de la lisozima . |
| El sustrato, al unirse, se distorsiona de la conformación de media silla del anillo de hexosa (debido al impedimento estérico con los aminoácidos de la proteína que obligan a la c6 ecuatorial a estar en la posición axial) en la conformación de silla [30] [ página necesaria ] |
Túneles cuánticos
Estos mecanismos tradicionales "sobre la barrera" han sido desafiados en algunos casos por modelos y observaciones de mecanismos "a través de la barrera" ( túnel cuántico ). Algunas enzimas operan con cinéticas que son más rápidas de lo que predeciría el ΔG ‡ clásico . En los modelos "a través de la barrera", un protón o un electrón pueden hacer un túnel a través de las barreras de activación. [31] [32] Se ha observado tunelización cuántica de protones en la oxidación de triptamina por amina deshidrogenasa aromática . [33]
La tunelización cuántica no parece proporcionar una ventaja catalítica importante, ya que las contribuciones de tunelización son similares en las reacciones catalizadas y no catalizadas en solución. [32] [34] [35] [36] Sin embargo, la contribución de la tunelización (que normalmente mejora las constantes de velocidad en un factor de ~ 1000 [33] en comparación con la velocidad de reacción de la ruta clásica 'sobre la barrera') es probablemente crucial a la viabilidad de los organismos biológicos. Esto enfatiza la importancia general de las reacciones de túnel en biología.
En 1971-1972 se formuló el primer modelo mecánico-cuántico de catálisis enzimática. [37] [38] [se necesita una fuente de terceros ]
Enzima activa
La energía de unión del complejo enzima-sustrato no puede considerarse como una energía externa necesaria para la activación del sustrato. La enzima de alto contenido energético puede transferir en primer lugar algún grupo energético específico X 1 desde el sitio catalítico de la enzima al lugar final del primer reactivo unido, luego otro grupo X 2 del segundo reactivo unido (o del segundo grupo del único reactivo reactivo) debe transferirse al sitio activo para finalizar la conversión del sustrato en producto y la regeneración enzimática. [39]
Podemos presentar toda la reacción enzimática como dos reacciones de acoplamiento:
( 1 )
( 2 )
Puede verse en la reacción ( 1 ) que el grupo X 1 de la enzima activa aparece en el producto debido a la posibilidad de una reacción de intercambio dentro de la enzima para evitar tanto la inhibición electrostática como la repulsión de los átomos. Por tanto, representamos a la enzima activa como un potente reactivo de la reacción enzimática. La reacción ( 2 ) muestra una conversión incompleta del sustrato porque su grupo X 2 permanece dentro de la enzima. Este enfoque como idea se había propuesto anteriormente basándose en las hipotéticas conversiones enzimáticas extremadamente altas (enzima catalíticamente perfecta). [40]
El punto crucial para la verificación del presente enfoque es que el catalizador debe ser un complejo de la enzima con el grupo de transferencia de la reacción. Este aspecto químico está respaldado por los mecanismos bien estudiados de las diversas reacciones enzimáticas. Considere la reacción de hidrólisis del enlace peptídico catalizada por una proteína pura α-quimotripsina (una enzima que actúa sin un cofactor), que es un miembro bien estudiado de la familia de las serina proteasas, ver. [41]
Presentamos los resultados experimentales de esta reacción como dos pasos químicos:
( 3 )
( 4 )
donde S 1 es un polipéptido, P 1 y P 2 son productos. El primer paso químico ( 3 ) incluye la formación de un intermedio de acil-enzima covalente. El segundo paso ( 4 ) es el paso de desacilación. Es importante señalar que el grupo H +, que se encuentra inicialmente en la enzima, pero no en el agua, aparece en el producto antes del paso de hidrólisis, por lo que puede considerarse como un grupo adicional de la reacción enzimática.
Por tanto, la reacción ( 3 ) muestra que la enzima actúa como un potente reactivo de la reacción. Según el concepto propuesto, el transporte de H de la enzima promueve la primera conversión de reactivo, ruptura del primer enlace químico inicial (entre los grupos P 1 y P 2 ). El paso de hidrólisis conduce a la ruptura del segundo enlace químico y la regeneración de la enzima.
El mecanismo químico propuesto no depende de la concentración de los sustratos o productos en el medio. Sin embargo, un cambio en su concentración provoca principalmente cambios de energía libre en el primer y último paso de las reacciones ( 1 ) y ( 2 ) debido a los cambios en el contenido de energía libre de cada molécula, ya sea S o P, en la solución acuosa. Este enfoque está de acuerdo con el siguiente mecanismo de contracción muscular. El paso final de la hidrólisis de ATP en el músculo esquelético es la liberación de producto causada por la asociación de cabezas de miosina con actina. [42] El cierre de la hendidura de unión a actina durante la reacción de asociación está estructuralmente acoplado con la apertura de la bolsa de unión a nucleótidos en el sitio activo de la miosina. [43]
En particular, los pasos finales de la hidrólisis de ATP incluyen la liberación rápida de fosfato y la liberación lenta de ADP. [44] [45] La liberación de un anión fosfato del anión ADP unido a una solución acuosa puede considerarse una reacción exergónica porque el anión fosfato tiene una masa molecular baja.
Así, llegamos a la conclusión de que la liberación primaria del fosfato inorgánico H 2 PO 4 - conduce a la transformación de una parte significativa de la energía libre de la hidrólisis del ATP en la energía cinética del fosfato solvatado, produciendo un flujo activo. Esta suposición de una transducción mecanoquímica local está de acuerdo con el mecanismo de contracción muscular de Tirosh, donde la fuerza muscular se deriva de una acción integrada de flujo activo creado por la hidrólisis de ATP. [46] [47]
Ejemplos de mecanismos catalíticos
En realidad, la mayoría de los mecanismos enzimáticos implican una combinación de varios tipos diferentes de catálisis.
Triosa fosfato isomerasa
La triosa fosfato isomerasa ( EC 5.3.1.1 ) cataliza la interconversión reversible de los dos isómeros de triosa fosfatos, dihidroxiacetona fosfato y D- gliceraldehído 3-fosfato .
Tripsina
La tripsina ( EC 3.4.21.4 ) es una serina proteasa que escinde los sustratos de proteínas después de los residuos de lisina o arginina utilizando una tríada catalítica para realizar una catálisis covalente y un orificio de oxianión para estabilizar la acumulación de carga en los estados de transición .
Aldolasa
La aldolasa ( EC 4.1.2.13 ) cataliza la descomposición de la fructosa 1,6-bisfosfato (F-1,6-BP) en gliceraldehído 3-fosfato y dihidroxiacetona fosfato ( DHAP ).
Difusividad enzimática
El advenimiento de los estudios de una sola molécula en la década de 2010 llevó a la observación de que el movimiento de enzimas no atadas aumenta con el aumento de la concentración de sustrato y el aumento de la entalpía de reacción . [48] Las observaciones posteriores sugieren que este aumento en la difusividad es impulsado por el desplazamiento transitorio del centro de masa de la enzima , lo que resulta en un "efecto de retroceso que impulsa la enzima". [49]
Similitud de reacción
La similitud entre reacciones enzimáticas ( EC ) se puede calcular utilizando cambios de enlace, centros de reacción o métricas de subestructura ( EC-BLAST ). [50]
Ver también
- Tríada catalítica
- Ensayo de enzimas
- Inhibidor de enzimas
- La cinética de enzimas
- Promiscuidad enzimática
- Dinámica proteica
- Pseudoenzimas , cuya ubicuidad a pesar de su inactividad catalítica sugiere implicaciones ómicas
- Túneles cuánticos
- El mapa de proteólisis
- Cristalografía resuelta en el tiempo
Referencias
- ^ Kamerlin, SC; Warshel, A (2010). "En los albores del siglo XXI: ¿Es la dinámica el eslabón perdido para comprender la catálisis enzimática?" . Proteínas: estructura, función y bioinformática . 78 (6): 1339–75. doi : 10.1002 / prot.22654 . PMC 2841229 . PMID 20099310 .
- ^ Laidler, Keith J. (1978). Química Física con Aplicaciones Biológicas . Benjamin / Cummings. pag. 427. ISBN 0-8053-5680-0.
- ^ Koshland DE (febrero de 1958). "Aplicación de una teoría de la especificidad enzimática a la síntesis de proteínas" . Proc. Natl. Acad. Sci. USA 44 (2): 98–104. Código Bibliográfico : 1958PNAS ... 44 ... 98K . doi : 10.1073 / pnas.44.2.98 . PMC 335371 . PMID 16590179 .
- ^ Anslyn, EV; Dougherty, DA (2006). Química orgánica física moderna . Libros universitarios de ciencia. ISBN 978-1-891389-31-3.
- ^ Savir Y; Tlusty T (2007). Scalas, Enrico (ed.). "Corrección conformacional: el impacto de los cambios conformacionales en la especificidad del reconocimiento molecular" . PLOS One . 2 (5): e468. Código Bibliográfico : 2007PLoSO ... 2..468S . doi : 10.1371 / journal.pone.0000468 . PMC 1868595 . PMID 17520027 .
- ^ Stanton, RV; Perakyla, M .; Bakowies, D .; Kollman, PA (1998). "Cálculos combinados ab initio y de energía libre para estudiar reacciones en enzimas y solución: hidrólisis de amidas en tripsina y solución acuosa". Mermelada. Chem. Soc . 120 (14): 3448–3457. doi : 10.1021 / ja972723x .
- ^ Kuhn, B .; Kollman, PA (2000). "QM-FE y cálculos de dinámica molecular sobre catecol O-metiltransferasa: energía libre de activación en la enzima y en solución acuosa y regioselectividad de la reacción catalizada por enzimas". Mermelada. Chem. Soc . 122 (11): 2586–2596. doi : 10.1021 / ja992218v .
- ^ Bruice, TC; Lightstone, FC (1999). "Contribuciones del estado fundamental y del estado de transición a las tasas de reacciones intramoleculares y enzimáticas". Acc. Chem. Res . 32 (2): 127-136. doi : 10.1021 / ar960131y .
- ^ Página, MI; Jencks, WP (1971). "Contribuciones entrópicas para acelerar las aceleraciones en las reacciones enzimáticas e intramoleculares y el efecto quelato" . Proc. Natl. Acad. Sci. USA . 68 (8): 1678–1683. Código bibliográfico : 1971PNAS ... 68.1678P . doi : 10.1073 / pnas.68.8.1678 . PMC 389269 . PMID 5288752 .
- ^ Warshel, A .; Parson, WW (2001). "Dinámica de reacciones bioquímicas y biofísicas: conocimiento de las simulaciones por computadora". Cuarto de galón. Rev. Biophys . 34 (4): 563–679. doi : 10.1017 / s0033583501003730 . PMID 11852595 . S2CID 28961992 .
- ^ a b c d Warshel, A .; Sharma, PK; Kato, M .; Xiang, Y .; Liu, H .; Olsson, MHM (2006). "Bases electrostáticas de la catálisis enzimática". Chem. Rev . 106 (8): 3210–3235. doi : 10.1021 / cr0503106 . PMID 16895325 .
- ^ Warshel, A .; Naray-Szabo, G .; Sussman, F .; Hwang, J.-K. (1989). "¿Cómo funcionan realmente las serina proteasas?". Bioquímica . 28 (9): 3629–37. doi : 10.1021 / bi00435a001 . PMID 2665806 .
- ^ Fersht, AR; Requena, Y. (diciembre de 1971). "Mecanismo de la hidrólisis de amidas catalizada por quimotripsina. Dependencia del pH de Kc y Km. Detección cinética de un intermedio". Mermelada. Chem. Soc . 93 (25): 7079–7087. doi : 10.1021 / ja00754a066 . PMID 5133099 .
- ^ Zeeberg, Barry; Caplow, Michael (abril de 1973). "Concerniente a un cambio informado en el paso determinante de la velocidad en la catálisis de quimotripsina". Mermelada. Chem. Soc . 95 (8): 2734–2735. doi : 10.1021 / ja00789a081 . PMID 4694533 .
- ^ Voet, Donald. (cop. 2011). Bioquímica . John Wiley e hijos. OCLC 808679090 . Verifique los valores de fecha en:
|date=( ayuda ) - ^ Marcus, RA (1965). "Sobre la teoría de las reacciones de transferencia de electrones. VI. Tratamiento unificado para reacciones homogéneas y de electrodos" (PDF) . J. Chem. Phys . 43 (2): 679–701. Código bibliográfico : 1965JChPh..43..679M . doi : 10.1063 / 1.1696792 .
- ^ Warshel, A (1978). "Energética de la catálisis enzimática" . Proc. Natl. Acad. Sci. USA . 75 (11): 5250–5254. Código Bibliográfico : 1978PNAS ... 75.5250W . doi : 10.1073 / pnas.75.11.5250 . PMC 392938 . PMID 281676 .
- ^ "Cómo funcionan las enzimas"
- ^ "CATÁLISIS DE POTENCIA DE CAMPOS ELÉCTRICOS EXTREMOS EN EL SITIO ACTIVO DE ISOMERASA DE CETOSTEROIDES", [1]
- ^ Toney, MD "Especificidad de reacción en enzimas piridoxal". Archivos de bioquímica y biofísica (2005) 433: 279-287
- ^ Centro de información sobre micronutrientes, Universidad Estatal de Oregon
- ^ Voet, Donald; Judith Voet (2004). Bioquímica . John Wiley & Sons Inc. págs. 986–989 . ISBN 978-0-471-25090-6.
- ^ Voet, Donald; Judith Voet (2004). Bioquímica . John Wiley & Sons Inc. págs. 604–606 . ISBN 978-0-471-25090-6.
- ↑ Piccirilli, Joseph A .; Vyle, Joseph S .; Caruthers, Marvin H .; Cech, Thomas R. (7 de enero de 1993). "Catálisis de iones metálicos en la reacción de ribozima de Tetrahymena". Naturaleza . 361 (6407): 85–88. Código Bibliográfico : 1993Natur.361 ... 85P . doi : 10.1038 / 361085a0 . PMID 8421499 . S2CID 4326584 .
- ^ BENDER, MYRON L. (1 de enero de 1962). "Catálisis de iones metálicos de reacciones orgánicas nucleofílicas en solución". Reacciones de ligandos coordinados . Avances en Química. 37 . SOCIEDAD QUÍMICA AMERICANA. págs. 19–36. doi : 10.1021 / ba-1963-0037.ch002 . ISBN 978-0841200388.
- ^ Fife, Thomas H .; Przystas, Theodore J. (1 de febrero de 1985). "Catálisis de iones metálicos divalentes en la hidrólisis de ésteres de ácido picolínico. Iones metálicos promovidos por iones hidróxido y reacciones catalizadas por agua". Revista de la Sociedad Química Estadounidense . 107 (4): 1041–1047. doi : 10.1021 / ja00290a048 . ISSN 0002-7863 .
- ^ Stadtman, ER (1 de enero de 1990). "Oxidación de proteínas catalizada por iones metálicos: mecanismo bioquímico y consecuencias biológicas" . Biología y Medicina de Radicales Libres . 9 (4): 315–325. doi : 10.1016 / 0891-5849 (90) 90006-5 . ISSN 0891-5849 . PMID 2283087 .
- ^ Jencks, William P. (1987) [1969]. Catálisis en Química y Enzimología . Serie McGraw-Hill en química avanzada (reimpresión ed.). Nueva York: Publicaciones de Dover . ISBN 9780486654607.
- ^ Warshel, A .; Levitt, M. (1976). "Estudios teóricos de reacciones enzimáticas: estabilización dieléctrica electrostática y estérica del ion carbonio en la reacción de lisozima". Revista de Biología Molecular . 103 (2): 227–49. doi : 10.1016 / 0022-2836 (76) 90311-9 . PMID 985660 .
- ^ <fundamentos de la bioquímica Voet, voet y Pratt 4ª edición>, que tiene una forma similar al estado de transición.
- ↑ García-Viloca, M; Gao, J; Karplus, M; Truhlar, DG (2004). "Cómo funcionan las enzimas: análisis por la teoría de la tasa moderna y simulaciones por computadora". Ciencia . 303 (5655): 186–95. Código Bibliográfico : 2004Sci ... 303..186G . doi : 10.1126 / science.1088172 . PMID 14716003 . S2CID 17498715 .
- ^ a b Olsson, MH; Siegbahn, PE; Warshel, A (2004). "Simulaciones del gran efecto de isótopos cinéticos y la dependencia de la temperatura de la transferencia del átomo de hidrógeno en la lipoxigenasa". Revista de la Sociedad Química Estadounidense . 126 (9): 2820–8. doi : 10.1021 / ja037233l . PMID 14995199 .
- ^ a b Masgrau, L; Roujeinikova, A; Johannissen, LO; Hothi, P; Basran, J; Ranaghan, KE; Mulholland, AJ; Sutcliffe, MJ; et al. (2006). "Descripción atómica de una reacción enzimática dominada por tunelización de protones". Ciencia . 312 (5771): 237–41. Código bibliográfico : 2006Sci ... 312..237M . doi : 10.1126 / science.1126002 . PMID 16614214 . S2CID 27201250 .
- ^ Hwang, J.-K .; Warshel, A. (1996). "¿Qué importancia tienen los movimientos nucleares de la mecánica cuántica en la catálisis enzimática?". Mermelada. Chem. Soc . 118 (47): 11745-11751. doi : 10.1021 / ja962007f .
- ↑ Ball, P. (2004). "Enzimas: ¿por casualidad o por diseño?" . Naturaleza . 431 (7007): 396–397. Código Bibliográfico : 2004Natur.431..396B . doi : 10.1038 / 431396a . PMID 15385982 . S2CID 228263 .
- ^ Olsson, MHM; Parson, WW; Warshel, A. (2006). "Contribuciones dinámicas a la catálisis enzimática: pruebas críticas de una hipótesis popular". Chem. Rev . 106 (5): 1737-1756. doi : 10.1021 / cr040427e . PMID 16683752 .
- ^ Volkenshtein MV, Dogonadze RR, Madumarov AK, Urushadze ZD, Kharkats Yu.I. Teoría de la catálisis enzimática. - Molekuliarnaya Biologia, Moscú, 6, 1972, 431-439
- ^ Volkenshtein MV, Dogonadze RR, Madumarov AK, Urushadze ZD, Kharkats Yu.I. Interacciones electrónicas y conformacionales en la catálisis enzimática. En: EL Andronikashvili (Ed.), Konformatsionnie Izmenenia Biopolimerov v Rastvorakh, Editorial "Nauka", Moscú, 1973, 153-157
- ^ Foigel, Alexander G. (2011). "¿Es la enzima un reactivo potente de la reacción bioquímica?". Bioquímica molecular y celular . 352 (1–2): 87–9. doi : 10.1007 / s11010-011-0742-4 . PMID 21318350 . S2CID 11133081 .
- ^ Fogel, AG (1982). "Cooperatividad de reacciones enzimáticas y aspectos moleculares de la transducción de energía". Mol. Celda. Biochem . 47 (1): 59–64. doi : 10.1007 / bf00241567 . PMID 7132966 . S2CID 21790380 .
- ^ Hengge, AC; Stein, RL (2004). "Papel de la movilidad conformacional de proteínas en la catálisis enzimática: acilación de alfa-quimotripsina por sustratos de péptidos específicos". Bioquímica . 43 (3): 742–747. doi : 10.1021 / bi030222k . PMID 14730979 .
- ^ Lymn, RW; Taylor, EW. (1971). "Mecanismo de hidrólisis de trifosfato de adenosina por actomiosina". Bioquímica . 10 (25): 4617–4624. doi : 10.1021 / bi00801a004 . PMID 4258719 .
- ^ Holmes, KC; Angert, yo; Kull, FG; Jahn, W; Schroder, RR. (2003). "La crio-microscopía electrónica muestra cómo la fuerte unión de la miosina a la actina libera nucleótidos". Naturaleza . 425 (6956): 423–427. Código bibliográfico : 2003Natur.425..423H . doi : 10.1038 / nature02005 . PMID 14508495 . S2CID 2686184 .
- ^ Siemankowski, RF; Wiseman, MO; Blanco, HD. (1985). "La disociación de ADP del subfragmento 1 de actomiosina es lo suficientemente lenta como para limitar la velocidad de acortamiento sin carga en el músculo de vertebrados" . Proc. Natl. Acad. Sci. USA . 82 (3): 658–662. Código Bibliográfico : 1985PNAS ... 82..658S . doi : 10.1073 / pnas.82.3.658 . PMC 397104 . PMID 3871943 .
- ^ Blanco, HD; Belknap, B; Webb, señor. (1997). "Cinética de escisión de nucleósido trifosfato y pasos de liberación de fosfato por actomiosina esquelética de conejo asociada, medida usando una nueva sonda fluorescente para fosfato". Bioquímica . 36 (39): 11828-11836. doi : 10.1021 / bi970540h . PMID 9305974 .
- ^ Tirosh, R; Bajo, WZ; Oplatka, A. (1990). "Movimiento de traslación de los filamentos de actina en presencia de meromiosina pesada y MgATP según lo medido por el ensanchamiento Doppler de la dispersión de la luz láser". Biochim. Biophys. Acta . 1037 (3): 274–280. doi : 10.1016 / 0167-4838 (90) 90025-b . PMID 2178685 .
- ^ Tirosh, R. (2006). "Protones balísticos y soluciones de agua inducidas por microondas (solitones) en transformaciones bioenergéticas" . En t. J. Mol. Sci . 7 (9): 320–345. doi : 10.3390 / i7090320 .
- ^ Muddana, Hari S .; Sengupta, Samudra; Mallouk, Thomas E .; et al. (28 de enero de 2010). "La catálisis de sustrato mejora la difusión de una sola enzima" . Revista de la Sociedad Química Estadounidense . 132 (7): 2110–1. doi : 10.1021 / ja908773a . PMC 2832858 . PMID 20108965 .
- ^ Riedel, Clemente; Gabizon, Ronen; Wilson, Christian AM; et al. (8 de enero de 2015). "El calor liberado durante el recambio catalítico mejora la difusión de una enzima" . Naturaleza . 517 (7533): 227–30. Código Bibliográfico : 2015Natur.517..227R . doi : 10.1038 / nature14043 . PMC 4363105 . PMID 25487146 .
- ^ Rahman, SA; Cuesta, SM; Furnham, N; Holliday, GL; Thornton, JM (2014). "EC-BLAST: una herramienta para buscar y comparar automáticamente reacciones enzimáticas" . Métodos de la naturaleza . 11 (2): 171-174. doi : 10.1038 / nmeth.2803 . PMC 4122987 . PMID 24412978 .
Otras lecturas
- Alan Fersht, Estructura y mecanismo en la ciencia de las proteínas: una guía para la catálisis enzimática y el plegamiento de proteínas. WH Freeman, 1998. ISBN 0-7167-3268-8
- Número dedicado de Philosophical Transactions B sobre catálisis cuántica en enzimas disponible gratuitamente. [ enlace muerto permanente ]
enlaces externos
- Medios relacionados con la catálisis enzimática en Wikimedia Commons
- Enzimas
- Catálisis




