Micropéptido
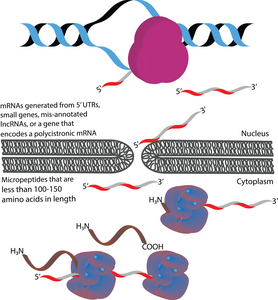
Los micropéptidos (también denominados microproteínas) son polipéptidos con una longitud de menos de 100-150 aminoácidos que están codificados por marcos de lectura abiertos cortos (sORF). [1] [2] [3] A este respecto, se diferencian de muchos otros polipéptidos pequeños activos, que se producen a través de la escisión postraduccional de polipéptidos más grandes. [1] [4] En términos de tamaño, los micropéptidos son considerablemente más cortos que las proteínas "canónicas", que tienen una longitud promedio de 330 y 449 aminoácidos en procariotas y eucariotas, respectivamente. [5]Los micropéptidos a veces se nombran de acuerdo con su ubicación genómica. Por ejemplo, el producto traducido de un marco de lectura abierto aguas arriba (uORF) podría denominarse péptido codificado por uORF (uPEP). [6] Los micropéptidos carecen de una secuencia de señalización N-terminal, lo que sugiere que es probable que estén localizados en el citoplasma . [1] Sin embargo, se han encontrado algunos micropéptidos en otros compartimentos celulares, como lo indica la existencia de micropéptidos transmembrana . [7] [8] Se encuentran tanto en procariotas como en eucariotas. [1] [9] [10] Los sORF a partir de los cuales se traducen los micropéptidos se pueden codificar en 5 'UTR, genes pequeños o ARNm policistrónicos . Algunos genes codificadores de micropéptidos originalmente se anotaron erróneamente como ARN largos no codificantes (lncRNA). [11]
Dado su pequeño tamaño, originalmente se pasaron por alto los sORF. Sin embargo, se han identificado cientos de miles de supuestos micropéptidos mediante diversas técnicas en una multitud de organismos. Solo una pequeña fracción de estos con potencial de codificación ha tenido su expresión y función confirmadas. Aquellos que se han caracterizado funcionalmente, en general, tienen papeles en la señalización celular , organogénesis y fisiología celular . A medida que se descubren más micropéptidos, también aumentan sus funciones. Una función reguladora es la de los peptoswitches, que inhiben la expresión de secuencias codificantes aguas abajo al estancar los ribosomas , a través de su activación directa o indirecta por moléculas pequeñas. [11]
Existen varias técnicas experimentales para identificar los ORF potenciales y sus productos de traducción. Estas técnicas solo son útiles para la identificación de sORF que pueden producir micropéptidos y no para la caracterización funcional directa.
Un método para encontrar posibles sORF y, por lo tanto, micropéptidos, es mediante la secuenciación de ARN ( RNA-Seq ). RNA-Seq utiliza la secuenciación de próxima generación (NGS) para determinar qué ARN se expresan en una célula, tejido u organismo determinados en un momento específico. Esta recopilación de datos, conocida como transcriptoma , se puede utilizar como un recurso para encontrar posibles sORF. [1] Debido a la gran probabilidad de que ocurran por casualidad sORF inferiores a 100 aa, es necesario realizar más estudios para determinar la validez de los datos obtenidos mediante este método. [11]
El perfil de ribosomas se ha utilizado para identificar posibles micropéptidos en un número creciente de organismos, incluidas moscas de la fruta, peces cebra, ratones y humanos. [11] Un método utiliza compuestos como harringtonina, puromicina o lactimidomicina para detener los ribosomas en los sitios de inicio de la traducción. [12] Esto indica dónde se está llevando a cabo la traducción activa. Los inhibidores de la elongación de la traducción, como la emetina o la cicloheximida, también pueden usarse para obtener huellas de ribosomas que tienen más probabilidades de dar como resultado un ORF traducido. [13] Si un ribosoma está unido a un sORF o cerca de él, supuestamente codifica un micropéptido. [1] [2] [14]
La espectrometría de masas (MS) es el estándar de oro para identificar y secuenciar proteínas. Usando esta técnica, los investigadores pueden determinar si los polipéptidos son, de hecho, traducidos de un sORF.