| Proteorrodopsina | ||||||||
|---|---|---|---|---|---|---|---|---|
 Visualización de dibujos animados de proteorodopsina de ELViture | ||||||||
| Identificadores | ||||||||
| Símbolo | Bac_rodopsina | |||||||
| Pfam | PF01036 | |||||||
| InterPro | TCDB | |||||||
| PROSITE | PDOC00291 | |||||||
| SCOP2 | 2brd / SCOPe / SUPFAM | |||||||
| TCDB | 3.E.1 | |||||||
| Superfamilia OPM | 6 | |||||||
| Proteína OPM | 4hyj | |||||||
| ||||||||
La proteorodopsina (también conocida como pRhodopsin ) es una familia de más de 50 proteínas de retinilideno fotoactivas , una familia más grande de proteínas transmembrana que utilizan la retina como cromóforo para la funcionalidad mediada por la luz, en este caso, una bomba de protones . Algunos homólogos existen como pentámeros o hexámeros. [ aclaración necesaria ] La prodopsina se encuentra en bacterias planctónicas marinas , arqueas y eucariotas ( protae ), pero se descubrió por primera vez en bacterias. [1] [2] [3] [4]
Su nombre se deriva de las proteobacterias que llevan el nombre del griego antiguo Πρωτεύς ( Proteus ), un dios del mar temprano mencionado por Homero como " Viejo del mar ", Ῥόδος (rhódon) para " rosa ", debido a su color rosado, y ὄψις ( opsis ) para "vista". Algunos miembros de la familia, los pigmentos homólogos similares a la rodopsina , es decir, la bacteriorrodopsina (de los cuales hay más de 800 tipos) tienen funciones sensoriales como las opsinas , integrales para la fototransducción visual. . Muchas de estas funciones sensoriales se desconocen, por ejemplo, la función de la neuropsina en la retina humana. [5] Se sabe que los miembros tienen diferentes espectros de absorción, incluida la luz visible verde y azul . [6] [7] [8] [9] [10] [11]
Descubrimiento
La proteorodopsina (PR o prodopsina) se descubrió por primera vez en 2000 dentro de un cromosoma artificial bacteriano de γ-proteobacterias marinas no cultivadas previamente , a las que todavía se hace referencia únicamente por sus datos metagenómicos de ribotipo , SAR86. La investigación fue un esfuerzo cooperativo entre cuatro partes: Oded Beja , Marcelino T. Suzuki y Edward F. DeLong en el Instituto de Investigación del Acuario de la Bahía de Monterey (Mosslanding, CA), L. Aravind y Eugene V Kooninen el Centro Nacional de Información Biotecnológica (Bethesda, MD), Andrew Hadd, Linh P. Nguyen, Stevan B. Jovanovich, Christian M. Gates y Rober A Feldman en Molecular Dynamics (Sunnyvale, CA), y finalmente John y Elena Spudich en el Departamento de Microbiología y Genética Molecular del Centro de Ciencias de la Salud de la Universidad de Texas en Houston . Se encontró que más especies de γ-proteobacterias, tanto Gram positivas como negativas, expresan la proteína. [1]
Distribución
Se han obtenido muestras de bacterias que expresan proteorhodopsina del Océano Pacífico Oriental , Océano Pacífico Central Norte y Océano Austral , Antártida . [12] Posteriormente, se identificaron genes de variantes de proteorrodopsina en muestras del Mediterráneo , el Mar Rojo , el Mar de los Sargazos y el Mar de Japón y el Mar del Norte . [4] [6]
Las variantes de proteorodopsina no se esparcen al azar, sino que se dispersan a lo largo de gradientes de profundidad basados en el ajuste de absorción máximo de la secuencia de holoproteína particular ; esto se debe principalmente a la absorción electromagnética por el agua que crea gradientes de longitud de onda en relación con la profundidad. Oxyrrhis marina es un protista dinoflagelado con proteorrodopsina absorbente de verde (un resultado del Grupo L109) que existe principalmente en pozas de marea y costas poco profundas, donde la luz verde todavía está disponible. Karlodinium micrum, otro dinolagelato, expresa una proteorrodopsina sintonizada en azul (E109) que puede estar relacionada con sus migraciones verticales en aguas profundas . [3] Originalmente se creía que O.Marina era un heterótrofo , sin embargo, la proteorodopsina bien puede participar de una manera funcionalmente significativa, ya que era el gen nuclear expresado con mayor abundancia y, además, se dispersa de manera desigual en el organismo, lo que sugiere alguna función de la membrana del orgánulo . Anteriormente, las únicas proteínas transductoras de energía solar eucariotas eran el Fotosistema I y el Fotosistema II . Se ha planteado la hipótesis de que la transferencia lateral de genes es el método por el cual la proteorodopsina se ha abierto camino en numerosos filos. Las bacterias, archea y eukarya colonizan la zona fótica.donde salen a la luz; La proteorodopsina se ha podido diseminar a través de esta zona, pero no a otras porciones de la columna de agua. [3] [4] [9] [13] [14]
Taxonomía
La proteorodopsina pertenece a una familia de proteínas de retinilideno similares, muy similares a sus homólogos arqueales, la clorhodopsina y la bacteriorrodopsina. La rodopsina sensorial fue descubierta por Franz Christian Boll en 1876. [11] [15] La bacteriorrodopsina fue descubierta en 1971 y nombrada en 1973 y actualmente solo se sabe que existe en el dominio archea, no en bacterias. [16] Halorhodopsin fue descubierta y nombrada por primera vez en 1977. [17] Bacteriorhodopsin y Halorhodopsin ambas solo existen en el dominio Archea mientras que la proteorhodopsina abarca bacterias, archea y eucariotas. La proteorodopsina comparte siete hélices α transmembrana retinianas unidas covalentemente por un mecanismo de base de Schiff a una lisinaresiduo en la séptima hélice (hélice G). La bacteriorrodopsina, como la proteorodopsina, es una bomba de protones impulsada por luz. La rodopsina sensorial es una proteína acoplada a G involucrada en la vista. [1] [17]
Sitio activo
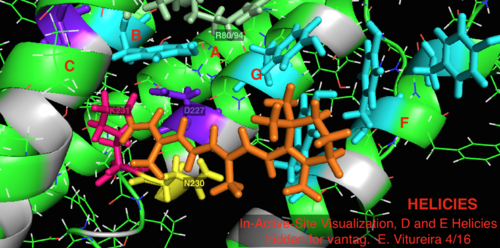
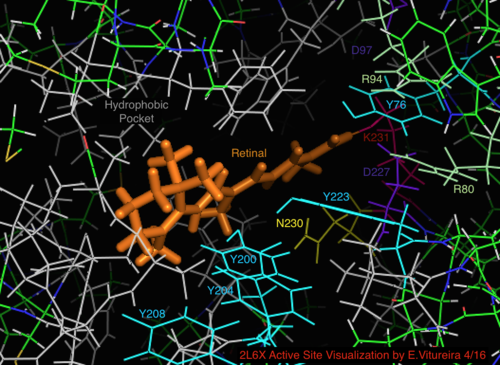
En comparación con su homólogo de arqueas bacteriorrodopsina más conocido, la mayoría de los residuos del sitio activo de importancia conocida para el mecanismo de la bacteriorrodopsina se conservan en la proteorrodopsina. Sin embargo, la similitud de secuencia no se conserva de manera significativa, ya sea de halo o bacteriorrodopsina. Los homólogos de los residuos del sitio activo Arg 82, Asp 85 (el aceptor de protones primario ), Asp 212 y Lys 216 (el sitio de unión de la base de Schiff retiniana ) en la bacteriorrodopsina se conservan como Arg94, Asp97, Asp227 y Lys231 en la proteorrodopsina. Sin embargo, en la proteorodopsina, no hay residuos de ácido carboxílico directamente homólogos a Glu 194 o Glu204 de bacteriorrodopsina (o Glu 108 y 204 dependiendo de la variante de bacrodopsina), que se cree que están involucrados en la vía de liberación de protones en la superficie extracelular. Sin embargo, Asp97 y Arg94 pueden reemplazar esta funcionalidad sin la proximidad de residuos cercana como en la bacteriorrodopsina. El departamento de química de la Universidad de Syracuse demostró de manera decisiva que Asp97 no puede ser el grupo de liberación de protones, ya que la liberación se produjo en condiciones forzadas en las que el grupo de ácido aspártico permaneció protonado. [18] [19] [20] [21]
Ligando
Las acciones rodopsina familia haloprotein el ligando de retina, aldehído de vitamina A, uno de los muchos tipos de vitamina A. retina es un conjugado de poli- insaturado cromóforo ( polieno ), obtenidos a partir de carnívoros dieta o por la vía de caroteno ( β-caroteno 15,15 '-monoxigenasa ).
Función
La proteorrodopsina funciona en todos los océanos de la Tierra como una bomba de H + impulsada por la luz, mediante un mecanismo similar al de la bacteriorrodopsina. Como en la bacteriorrodopsina, el cromóforo retiniano de la proteorrodopsina se une covalentemente a la apoproteína a través de una base de Schiff protonada en Lys231. La configuración del cromóforo retiniano en la proteorrodopsina no fotolizada es predominantemente trans [18] , y se isomeriza a 13-cis tras la iluminación con luz. Se han propuesto varios modelos del fotociclo completo de proteorodopsina, basados en FTIR y espectroscopía UV-visible ; se asemejan a los modelos de fotociclo establecidos para la bacteriorrodopsina.[18] [20] [21] [22] Se han descubierto y expresado fotosistemas completos basados en proteorrodopsina en E. coli, lo que les otorga una capacidad adicional de gradiente de energía mediada por la luz para la generación de ATP sin necesidad externa de retina o precursores; con el PR, el gen de otras cinco proteínas codifica la vía biosintética del fotopigmento. [23]
Ingeniería genética
Si el gen de la proteorrodopsina se inserta en E. coli y se administra retinal a estas bacterias modificadas , entonces incorporarán el pigmento en su membrana celular y bombearán H + en presencia de luz. Un púrpura intenso es representativo de colonias claramente transformadas, debido a la absorción de luz. Los gradientes de protones pueden usarse para alimentar otras estructuras de proteínas de membrana o usarse para acidificar un orgánulo de tipo vesícula. [1] Se demostró además que el gradiente de protones generado por proteorrodopsina podría usarse para generar ATP. [23]
Galería

Holoenzima (verde) con hélices AG etiquetadas (violeta) y ligando retiniano (naranja)

Visualización de superficie de proteorodopsina mostrando terminales

Visualización del sitio activo unido a la retina de la estructura de la proteína 2L6X de la pRhodopsina, residuos codificados por colores y marcados por actividad, el ligando es naranja.

Visualización etiquetada y codificada por colores de dibujos animados en el sitio activo 2L6x, hélices D y E ocultas para una ventaja, sitio de unión del ligando retiniano
Referencias
- ^ a b c d Béjà O, Aravind L, Koonin EV, Suzuki MT, Hadd A, Nguyen LP, Jovanovich SB, Gates CM, Feldman RA, Spudich JL, Spudich EN, DeLong EF (septiembre de 2000). "Rodopsina bacteriana: evidencia de un nuevo tipo de fototrofia en el mar". Ciencia . 289 (5486): 1902–6. Código Bibliográfico : 2000Sci ... 289.1902B . doi : 10.1126 / science.289.5486.1902 . PMID 10988064 .
- ^ Lin S, Zhang H, Zhuang Y, Tran B, Gill J (noviembre de 2010). "Los análisis metatranscriptómicos basados en líderes empalmados conducen al reconocimiento de características genómicas ocultas en dinoflagelados" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 107 (46): 20033–8. Código Bibliográfico : 2010PNAS..10720033L . doi : 10.1073 / pnas.1007246107 . PMC 2993343 . PMID 21041634 .
- ↑ a b c Slamovits CH, Okamoto N, Burri L, James ER, Keeling PJ (2011). "Una bomba de protones de proteorrodopsina bacteriana en eucariotas marinos" . Comunicaciones de la naturaleza . 2 (2): 183. Código Bibliográfico : 2011NatCo ... 2E.183S . doi : 10.1038 / ncomms1188 . PMID 21304512 .
- ↑ a b c Frigaard NU, Martinez A, Mincer TJ, DeLong EF (febrero de 2006). "Transferencia de genes laterales de proteorodopsina entre bacterias planctónicas marinas y arqueas". Naturaleza . 439 (7078): 847–50. Código Bibliográfico : 2006Natur.439..847F . doi : 10.1038 / nature04435 . PMID 16482157 .
- ^ Buhr ED, Yue WW, Ren X, Jiang Z, Liao HW, Mei X, Vemaraju S, Nguyen MT, Reed RR, Lang RA, Yau KW, Van Gelder RN (20 de octubre de 2015). " " Fotoentrenamiento mediado por neuropsina (OPN6) de osciladores circadianos locales en la retina y córnea de mamíferos " " . Actas de la Academia Nacional de Ciencias . 112 (42): 13093-13098. Código bibliográfico : 2015PNAS..11213093B . doi : 10.1073 / pnas.1516259112 . PMC 4620855 . PMID 26392540 .
- ↑ a b Béjà O, Spudich EN, Spudich JL, Leclerc M, DeLong EF (junio de 2001). "Fototrofia de proteorodopsina en el océano". Naturaleza . 411 (6839): 786–9. doi : 10.1038 / 35081051 . PMID 11459054 .
- ^ Man D, Wang W, Sabehi G, Aravind L, Post AF, Massana R, Spudich EN, Spudich JL, Béjà O (abril de 2003). "Diversificación y sintonía espectral en proteorrodopsinas marinas" . El diario EMBO . 22 (8): 1725–31. doi : 10.1093 / emboj / cdg183 . PMC 154475 . PMID 12682005 .
- ^ Kelemen BR, Du M, Jensen RB (diciembre de 2003). "Proteorrodopsina en colores vivos: diversidad de propiedades espectrales dentro de las células bacterianas vivas" . Biochimica et Biophysica Acta (BBA) - Biomembranas . 1618 (1): 25–32. doi : 10.1016 / j.bbamem.2003.10.002 . PMID 14643930 .
- ↑ a b Sabehi G, Kirkup BC, Rozenberg M, Stambler N, Polz MF, Béjà O (mayo de 2007). "Adaptación y sintonía espectral en proteorrodopsinas marinas divergentes del Mediterráneo oriental y los mares de los Sargazos" . El diario ISME . 1 (1): 48–55. doi : 10.1038 / ismej.2007.10 . PMID 18043613 .
- ^ Enciclopedia de las Ciencias Neruológicas . Prensa académica. 29 de abril de 2014. p. 441. ISBN 978-0-12-385158-1.
- ↑ a b Giese, Arthur C (septiembre de 2013). Fotofisiología: principios generales; Acción de la luz sobre las plantas . Elsevier. pag. 9. ISBN 978-1-4832-6227-7.
- ^ Venter JC, Remington K, Heidelberg JF, Halpern AL, Rusch D, Eisen JA, Wu D, Paulsen I, Nelson KE, Nelson W, Fouts DE, Levy S, Knap AH, Lomas MW, Nealson K, White O, Peterson J, Hoffman J, Parsons R, Baden-Tillson H, Pfannkoch C, Rogers YH, Smith HO (abril de 2004). "Secuenciación escopeta del genoma ambiental del Mar de los Sargazos". Ciencia . 304 (5667): 66–74. Código Bibliográfico : 2004Sci ... 304 ... 66V . CiteSeerX 10.1.1.124.1840 . doi : 10.1126 / science.1093857 . PMID 15001713 .
- ^ Giovannoni, SJ; Bibbs, L; Cho, JC; Stapels, MD; Desiderio, R; Vergin, KL; Rappé, MS; Laney, S; Wilhelm, LJ; Tripp, HJ; Mathur, EJ; Barofsky, DF (3 de noviembre de 2005). "Proteorrodopsina en la ubicua bacteria marina SAR11". Naturaleza . 438 (7064): 82–5. Código bibliográfico : 2005Natur.438 ... 82G . doi : 10.1038 / nature04032 . PMID 16267553 .
- ^ Kushwaha, SC; Kates, M (23 de agosto de 1973). "Aislamiento e identificación de" bacteriorrodopsina "y carotenoides C40 menores en Halobacterium cutirubrum". Biochimica et Biophysica Acta (BBA) - Lípidos y metabolismo de lípidos . 316 (2): 235–43. doi : 10.1016 / 0005-2760 (73) 90013-1 . PMID 4741911 .
- ^ Enciclopedia de las Ciencias Neurológicas . Prensa académica. Abril de 2014. p. 441. ISBN 978-0-12-385158-1.
- ^ Oesterhelt, D; Stoeckenius, W (29 de septiembre de 1971). "Proteína similar a la rodopsina de la membrana púrpura de Halobacterium halobium". Nature New Biology . 233 (39): 149–52. doi : 10.1038 / newbio233149a0 . PMID 4940442 .
- ^ a b Matsuno-Yagi, A; Mukohata, Y (9 de septiembre de 1977). "Dos posibles funciones de la bacteriorrodopsina; un estudio comparativo de cepas de Halobacterium halobium que difieren en la pigmentación". Comunicaciones de investigación bioquímica y biofísica . 78 (1): 237–43. doi : 10.1016 / 0006-291x (77) 91245-1 . PMID 20882 .
- ^ a b c Dioumaev AK, Brown LS, Shih J, Spudich EN, Spudich JL, Lanyi JK (abril de 2002). "Transferencias de protones en el ciclo de reacción fotoquímica de la proteorodopsina". Bioquímica . 41 (17): 5348–58. doi : 10.1021 / bi025563x . PMID 11969395 .
- ^ Partha R, Krebs R, Caterino TL, Braiman MS (junio de 2005). "Acoplamiento debilitado de arginina conservada al cromóforo de proteorrodopsina y su contraión implica diferencias estructurales de bacteriorrodopsina" . Biochimica et Biophysica Acta (BBA) - Bioenergética . 1708 (1): 6–12. doi : 10.1016 / j.bbabio.2004.12.009 . PMID 15949979 .
- ↑ a b Dioumaev AK, Wang JM, Bálint Z, Váró G, Lanyi JK (junio de 2003). "El transporte de protones por la proteorrodopsina requiere que el contraión de la base de Schiff retinal Asp-97 sea aniónico". Bioquímica . 42 (21): 6582–7. doi : 10.1021 / bi034253r . PMID 12767242 .
- ^ a b Krebs RA, Alexiev U, Partha R, DeVita AM, Braiman MS (abril de 2002). "Detección de liberación rápida de H + activada por luz y formación de intermedio M a partir de proteorrodopsina" . Fisiología de BMC . 2 : 5. doi : 10.1186 / 1472-6793-2-5 . PMC 103662 . PMID 11943070 .
- ^ Xiao Y, Partha R, Krebs R, Braiman M (enero de 2005). "Espectroscopía FTIR resuelta en el tiempo de los fotointermedios implicados en la liberación transitoria rápida de H + por la proteorrodopsina". El Journal of Physical Chemistry B . 109 (1): 634–41. doi : 10.1021 / jp046314g . PMID 16851056 .
- ↑ a b Martinez A, Bradley AS, Waldbauer JR, Summons RE, DeLong EF (2007). "La expresión del gen del fotosistema de proteorodopsina permite la fotofosforilación en un huésped heterólogo" . PNAS . 104 (13): 5590–5595. Código Bibliográfico : 2007PNAS..104.5590M . doi : 10.1073 / pnas.0611470104 . PMC 1838496 . PMID 17372221 .



