ARN condicional pequeño
Un ARN condicional pequeño ( ARNc ) es una molécula o complejo de ARN pequeño (normalmente menos de aproximadamente 100 nt) diseñado para interactuar y cambiar la conformación condicionalmente en respuesta a entradas moleculares afines para realizar la transducción de señales in vitro , in situ o in vivo . . [1]
En ausencia de moléculas de entrada afines, los scRNA se modifican para coexistir de forma metaestable o estable sin interactuar. La detección de las entradas afines inicia cambios conformacionales aguas abajo de uno o más scRNA que conducen a la generación de la señal de salida deseada. La señal de salida puede estar destinada a leer el estado de los circuitos biológicos endógenos (p. ej., mapear la expresión génica para investigación biológica o diagnóstico médico), [2] o para regular el estado de los circuitos biológicos endógenos (p. ej., expresión génica perturbadora para investigación biológica o tratamiento médico). [1] Las secuencias de scRNA se pueden programar para reconocer diferentes entradas o para activar diferentes salidas,[1] [2] [3] logrando incluso selectividad de secuencia de un solo nucleótido. [3] La transducción de señales de scRNA aprovecha los principios de las disciplinas emergentes de la nanotecnología de ARN dinámico, la programación molecular y la biología sintética .
Los scRNA marcados con fluoróforo se diseñaron para transducir entre la detección de objetivos de mRNA y la generación de polímeros de amplificación fluorescentes brillantes in situ (Figura 1). [2] En este contexto, la transducción de señales de scRNA permite el mapeo multiplexado de la expresión de mRNA dentro de embriones de vertebrados intactos (Figura 2). [2]
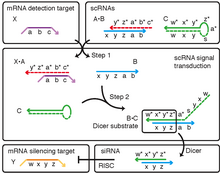
Los scRNA se han diseñado para realizar la transducción de forma y secuencia para producir condicionalmente un sustrato de Dicer dirigido al ARNm Y del 'objetivo de silenciamiento' tras la detección de un ARNm X del 'objetivo de detección' independiente, con el procesamiento posterior de Dicer que produce un pequeño ARN de interferencia ( siRNA ) apuntando al ARNm Y para su destrucción (Figura 3). [1] En este contexto, la transducción de señales de scRNA proporciona un paso hacia la implementación de la interferencia de RNA condicional (Figura 4).

Figura 1 . Amplificación de señales fluorescentes utilizando pequeños ARN condicionales. Los scRNA fluorescentes metaestables se autoensamblan en polímeros de amplificación fluorescentes tras la detección de un iniciador de ARN afín. El iniciador I se nuclea con la horquilla H1 a través del emparejamiento de bases con el toehold '1' monocatenario, mediando una migración de rama de 3 vías que abre la horquilla para formar el complejo I·H1 que contiene el segmento monocatenario '3*-2*'. Este complejo se nuclea con la horquilla H2 por medio del apareamiento de bases hasta el toehold '3', mediando una migración de rama que abre la horquilla para formar el complejo I·H1·H2 que contiene el segmento monocatenario '2*-1*'. Por lo tanto, la secuencia iniciadora se regenera, proporcionando la base para una reacción en cadena de pasos de polimerización alternativos de H1 y H2. Las estrellas rojas denotan fluoróforos. Imagen de Choi et al. 2010;
usado con permiso de Nature Publishing Group.

Figura 2 . Mapeo simultáneo de cinco ARNm objetivo diferentes dentro de un embrión de pez cebra intacto usando amplificación de señal de ARNc y microscopía confocal. Las sondas de ARN complementarias a los objetivos de ARNm desencadenan reacciones en cadena en las que los scRNA marcados con fluoróforo se autoensamblan en polímeros de amplificación fluorescentes atados. La programabilidad y la selectividad de secuencia de estas cascadas de amplificación permiten que cinco amplificadores de scRNA funcionen de forma independiente al mismo tiempo en la misma muestra, cada uno de los cuales se tiñe para la expresión de uno de los cinco mRNA objetivo. Barra de escala: 50 μm. Imagen de Choi et al. 2010;
[2] usado con permiso de Nature Publishing Group.

Figura 3 . Formación condicional de sustrato de Dicer a través de transducción de forma y secuencia con pequeños ARN condicionales. El scRNA A·B detecta el objetivo de detección de ARNm X (que contiene la subsecuencia 'abc'), lo que conduce a la producción del sustrato Dicer B·C dirigido al objetivo de silenciamiento del ARNm Y (que contiene la subsecuencia independiente 'wxyz'). Los scRNA A·B y C son estables en ausencia de X. A intercambia B por X (paso 1) a través de la migración de rama de 3 vías mediada por toehold y la disociación espontánea. B se ensambla con C (paso 2) a través de la nucleación de bucle/toehold y la migración de rama de 3 vías para formar el sustrato Dicer B·C. Las modificaciones químicas (2'OMe-RNA) evitan la degradación: A y parte de C (esqueleto discontinuo). Imagen de Hochrein et al. 2013;
[1] usado con permiso de la American Chemical Society.

Figura 4 . El ARNi condicional (si se transcribe el gen X, silencia el gen Y independiente) proporciona un marco conceptual para ejercer un control espaciotemporal sobre la eliminación de genes. Con este fin, los pequeños ARN condicionales (scRNA) interactúan y cambian la conformación para transducir entre la unión del 'objetivo de detección' X del ARNm y la producción de un sustrato de Dicer que se dirige al 'objetivo silenciador' Y del ARNm independiente. Imagen de Hochrein et al. 2013;
[1] usado con permiso de la American Chemical Society.