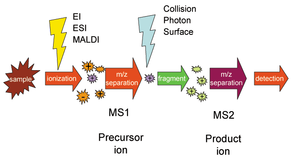
El monitoreo de reacciones seleccionadas ( SRM ) es un método utilizado en espectrometría de masas en tándem en el que se selecciona un ión de una masa particular en la primera etapa de un espectrómetro de masas en tándem y un producto iónico de una reacción de fragmentación del ión precursor se selecciona en la segunda etapa. Etapa de espectrómetro de masas para detección. [1]
Variantes [ editar ]
Un caso general de SRM puede representarse mediante
donde el ión precursor ABCD + se selecciona mediante la primera etapa de espectrometría de masas (MS1), se disocia en la molécula AB y el ión producto CD + , y este último se selecciona mediante la segunda etapa de espectrometría de masas (MS2) y se detecta. El par de iones precursor y producto se denomina "transición" de SRM. [2]
El monitoreo de reacción consecutiva ( CRM ) es la aplicación en serie de tres o más etapas de espectrometría de masas a SRM, representada en un caso simple por
donde ABCD + es seleccionado por MS1, se disocia en la molécula AB y el ion CD + . [3] El ion se selecciona en la segunda etapa de espectrometría de masas MS2 y luego se somete a una fragmentación adicional para formar el ion D + que se selecciona en la tercera etapa de espectrometría de masas MS3 y se detecta.
La monitorización de reacciones múltiples ( MRM ) es la aplicación de la monitorización de reacciones seleccionadas a múltiples iones de producto de uno o más iones precursores, [3] [4] por ejemplo
donde ABCD + es seleccionado por MS1 y se disocia por dos vías, formando AB + o CD + . Los iones son seleccionados secuencialmente por MS2 y detectados. El monitoreo de reacciones en paralelo ( PRM ) es la aplicación de SRM con detección en paralelo de todas las transiciones en un solo análisis utilizando un espectrómetro de masas de alta resolución. [5]
Proteómica [ editar ]
SRM se puede utilizar para proteómica cuantitativa dirigida mediante espectrometría de masas . [6] Después de la ionización en, por ejemplo, una fuente de electropulverización , primero se aísla un precursor de péptido para obtener una población de iones sustancial de la mayoría de las especies deseadas. A continuación, esta población se fragmenta para producir iones producto cuyas abundancias de señales son indicativas de la abundancia del péptido en la muestra. Este experimento se puede realizar en espectrómetros de masas de triple cuadrupolo , donde Q 1 de resolución de masas aísla el precursor, q 2actúa como una celda de colisión, y la masa de resolución de Q 3 se cicla a través de los iones de productos que son detectadas a la salida de la última cuadrupolo por un multiplicador de electrones . Un par de precursor / producto a menudo se denomina transición . Se trabaja mucho para garantizar que las transiciones se seleccionen con la máxima especificidad.
Usando el etiquetado isotópico con péptidos fuertemente marcados (p. Ej., D , 13 C o 15 N ) en una matriz compleja como estándares de concentración , SRM se puede usar para construir una curva de calibración que puede proporcionar la cuantificación absoluta (es decir, número de copias por célula ) del péptido ligero nativo y, por extensión, su proteína original . [2]
SRM se ha utilizado para identificar las proteínas codificadas por genes de tipo salvaje y mutantes ( proteínas mutantes ) y cuantificar sus números absolutos de copias en tumores y fluidos biológicos, respondiendo así las preguntas básicas sobre el número absoluto de copias de proteínas en una sola célula, que será esencial en el modelado digital de células de mamíferos y el cuerpo humano, y los niveles relativos de proteínas genéticamente anormales en tumores, y resultará útil para aplicaciones de diagnóstico. [7] [8] SRM también se ha utilizado como un método para desencadenar escaneos de iones de productos completos de péptidos para a) confirmar la especificidad de la transición de SRM, ob) detectar modificaciones postraduccionales específicas que están por debajo del límite de detección de análisis estándar de EM. [9]En 2017, SRM se desarrolló para ser una plataforma de detección dirigida de proteínas basada en espectrometría de masas altamente sensible y reproducible (titulada "SAFE-SRM"), y se ha demostrado que la nueva tubería basada en SRM tiene grandes ventajas en aplicaciones de proteómica clínica. sobre las tuberías tradicionales de SRM, y ha demostrado un rendimiento de diagnóstico drásticamente mejorado con respecto a los métodos de diagnóstico de biomarcadores de proteínas basados en anticuerpos, como ELISA . [10]
Ver también [ editar ]
- Espectrometría de masas de proteínas
Referencias [ editar ]
- ^ E. de Hoffmann (1996). "Espectrometría de masas en tándem: una cartilla" (PDF) . Revista de espectrometría de masas . 31 (2): 129-137. doi : 10.1002 / (SICI) 1096-9888 (199602) 31: 2 <129 :: AID-JMS305> 3.0.CO; 2-T .
- ↑ a b Lange, Vinzenz; Picotti, Paola; Domon, Bruno; Aebersold, Ruedi (2008). "Monitoreo de reacción seleccionada para proteómica cuantitativa: un tutorial" . Biología de sistemas moleculares . 4 : 222. doi : 10.1038 / msb.2008.61 . ISSN 1744-4292 . PMC 2583086 . PMID 18854821 .
- ^ a b Murray, Kermit K .; Boyd, Robert K .; Eberlin, Marcos N .; Langley, G. John; Li, Liang; Naito, Yasuhide (2013). "Definiciones de términos relacionados con la espectrometría de masas (Recomendaciones de la IUPAC 2013)" . Química pura y aplicada . 85 (7): 1515–1609. doi : 10.1351 / PAC-REC-06-04-06 . ISSN 1365-3075 .
- ^ Kondrat, RW; McClusky, GA; Cooks, RG (1978). "Monitorización de reacciones múltiples en espectrometría de masas / espectrometría de masas para el análisis directo de mezclas complejas". Química analítica . 50 (14): 2017-2021. doi : 10.1021 / ac50036a020 . ISSN 0003-2700 .
- ^ Peterson, AC; Russell, JD; Bailey, DJ; Westphall, MS; Coon, JJ (2012). "Monitoreo de reacciones paralelas para proteómica dirigida cuantitativa de alta resolución y alta precisión de masa" . Proteómica molecular y celular . 11 (11): 1475–1488. doi : 10.1074 / mcp.O112.020131 . ISSN 1535-9476 . PMC 3494192 . PMID 22865924 .
- ↑ Picotti, Paola; Aebersold, Ruedi (2012). "Proteómica basada en la monitorización de reacciones seleccionadas: flujos de trabajo, potencial, dificultades y direcciones futuras". Métodos de la naturaleza . 9 (6): 555–566. doi : 10.1038 / nmeth.2015 . ISSN 1548-7091 . PMID 22669653 .
- ^ Wang Q, Chaerkady R, Wu J, et al. (Febrero de 2011). "Proteínas mutantes como biomarcadores específicos del cáncer" . Proc. Natl. Acad. Sci. USA . 108 (6): 2444–9. Código bibliográfico : 2011PNAS..108.2444W . doi : 10.1073 / pnas.1019203108 . PMC 3038743 . PMID 21248225 .
- ^ "Espectrometría de masas de monitorización de reacción seleccionada para cuantificación absoluta de proteínas" . Revista de experimentos visualizados .
- ^ Unwin RD, Griffiths JG, et al. (Agosto de 2005). "Monitoreo de reacciones múltiples para identificar sitios de fosforilación de proteínas con alta sensibilidad" . Proteómica molecular y celular . 4 (8): 1134–44. doi : 10.1074 / mcp.M500113-MCP200 . PMID 15923565 .
- ^ Wang Q, Zhang M, Tomita T, et al. (Diciembre de 2017). "Enfoque de seguimiento de reacción seleccionado para validar biomarcadores de péptidos" . Proc. Natl. Acad. Sci. USA . 114 (51): 13519-13524. doi : 10.1073 / pnas.1712731114 . PMC 5754789 . PMID 29203663 .