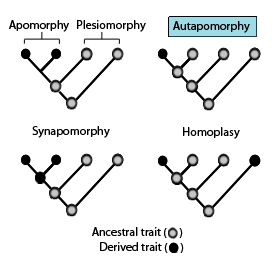
En filogenética , una autapomorfia es un rasgo distintivo, conocido como rasgo derivado , que es exclusivo de un taxón determinado. Es decir, se encuentra solo en un taxón , pero no se encuentra en ningún otro taxón externo , ni siquiera en los más estrechamente relacionados con el taxón focal (que puede ser una especie , familia o, en general, cualquier clado). [2] Por lo tanto, puede considerarse una apomorfia en relación con un solo taxón. [3] La palabra autapomorfia , introducida por primera vez en 1950 por el entomólogo alemán Willi Hennig , se deriva de la Palabras griegas αὐτός, aut- = "self"; ἀπό, apo = "lejos de"; y μορφή, morphḗ = "forma". [4]
Discusión
Debido a que las autapomorfias solo están presentes en un único taxón, no transmiten información sobre la relación. Por lo tanto, las autapomorfias no son útiles para inferir relaciones filogenéticas. Sin embargo, la autapomorfía, como la sinapomorfia y la plesiomorfia, es un concepto relativo que depende del taxón en cuestión. Una autapomorfia en un nivel dado bien puede ser una sinapomorfia en un nivel menos inclusivo. [5] Un ejemplo de autapomorfia se puede describir en serpientes modernas. Las serpientes han perdido los dos pares de patas que caracterizan a todos los Tetrapoda y los taxones más cercanos a Ophidia.- así como sus ancestros comunes - todos tienen dos pares de patas. Por tanto, el taxón Ophidia presenta una autapomorfia con respecto a su ausencia de patas. [3]
El concepto de especie autapomórfica es uno de los muchos métodos que los científicos pueden utilizar para definir y distinguir especies entre sí. Esta definición asigna especies sobre la base de la cantidad de divergencia asociada con la incompatibilidad reproductiva, que se mide esencialmente por el número de autapomorfias. [6] Este método de agrupación se conoce a menudo como el " concepto de especie monofilética " o el concepto de "filoespecie" y fue popularizado por DE Rosen en 1979. Dentro de esta definición, una especie se considera "el grupo monofilético menos inclusivo definido por al al menos una autapomorfia ". [7]Si bien este modelo de especiación es útil porque evita agrupaciones no monofiléticas, también tiene sus críticas. NI Platnick, por ejemplo, cree que el concepto de especie autapomórfica es inadecuado porque permite la posibilidad de aislamiento reproductivo y especiación al tiempo que revoca el estado de "especie" de la población madre. En otras palabras, si una población periférica se separa y se aísla reproductivamente, posiblemente necesitaría desarrollar al menos una autapomorfia para ser reconocida como una especie diferente. Si esto puede suceder sin que la población madre más grande también desarrolle una nueva autapomorfia, entonces la población madre no puede seguir siendo una especie bajo el concepto de especie autapomórfica: ya no tendría ninguna apomorfia que no también sea compartida por la especie hija.[8]
Similitudes filogenéticas: estos términos filogenéticos se utilizan para describir diferentes patrones de estados de carácter o rasgo ancestrales y derivados como se indica en el diagrama anterior en asociación con sinapomorfias. [1]
- La homoplasia en la sistemática biológica es cuando un rasgo se ha ganado o perdido de forma independiente en linajes separados durante la evolución. Esta evolución convergente lleva a que las especies compartan de forma independiente un rasgo que es diferente del rasgo que se infiere que estuvo presente en su ancestro común. [9] [10] [11]
- Rasgo derivado de la homoplasia paralela presente en dos grupos o especies sin un ancestro común debido a la evolución convergente . [12]
- Homoplasia inversa : rasgo presente en un antepasado pero no en descendientes directos que reaparece en descendientes posteriores. [13]
- Apomorfia : un rasgo derivado. La apomorfia compartida por dos o más taxones y heredada de un ancestro común es la sinapomorfia. La apomorfia exclusiva de un taxón determinado es la autapomorfia. [14] [15] [16] [17]
- Sinapomorfia / Homología : un rasgo derivado que se encuentra en algunos o todos los grupos terminales de un clado y se hereda de un ancestro común, para el cual era una autapomorfia (es decir, no está presente en su ancestro inmediato).
- Sinapomorfia subyacente : una sinapomorfia que se ha vuelto a perder en muchos miembros del clado. Si se pierde en todos menos uno, puede ser difícil distinguirlo de una autapomorfia.
- Autapomorphy: un rasgo derivado distintivo que es exclusivo de un taxón o grupo determinado. [3]
- Symplesiomorphy : un rasgo ancestral compartido por dos o más taxones.
- Plesiomorfia - una simmplesiomorfia discutida en referencia a un estado más derivado.
- Pseudoplesiomorfia: es un rasgo que no puede identificarse ni como una plesiomorfia ni como una apomorfia que sea una reversión. [18]
- Reversión: es la pérdida de un rasgo derivado presente en el antepasado y el restablecimiento de un rasgo plesiomórfico.
- Convergencia: evolución independiente de un rasgo similar en dos o más taxones.
- Hemiplasia [19] [20]
Referencias
- ^ a b Page RD, Holmes EC (14 de julio de 2009). Evolución molecular: un enfoque filogenético . John Wiley e hijos. ISBN 978-1-4443-1336-9. OCLC 609843839 .
- ^ Futuyma DJ (1998). Biología evolutiva (3ª ed.). Sinauer Associates, Inc. pág. 95.
- ↑ a b c Appel RD, Feytmans E (2009). "Capítulo 3: Introducción a la filogenética y sus aspectos moleculares". Bioinformática: una perspectiva suiza (1ª ed.). Compañía Editorial Científica Mundial.
- ^ Calow PP (2009). Enciclopedia de Ecología y Gestión Ambiental . John Wiley e hijos. ISBN 978-1-4443-1324-6. OCLC 1039167559 .
- ^ Forey PL (1997). Historia de los peces celacantos (1ª ed.). Velocista.
- ^ Howard DJ, Berlocher SH (1998). Formas sin fin: especies y especiación (1ª ed.). Estados Unidos: Oxford University Press. ISBN 978-0-19-510901-6. OCLC 60181901 .
- ^ Toro en (2004). Diversidad microbiana y bioprospección . Prensa ASM.
- ^ Platnick NI (2001). " De los cladogramas a las clasificaciones: el camino hacia DePhylocode " . (PDF) . La Asociación de Sistemática.
- ^ Gauger A (17 de abril de 2012). "¡La similitud ocurre! El problema de la homoplastia" . Evolution Today y Science News .
- ^ Sanderson MJ, Hufford L (21 de octubre de 1996). Homoplasia: la recurrencia de la similitud en la evolución . Elsevier. ISBN 978-0-08-053411-4. OCLC 173520205 .
- ^ Brandley MC, Warren DL, Leaché AD, McGuire JA (abril de 2009). "Soporte de homoplastia y clado" . Biología sistemática . 58 (2): 184–98. doi : 10.1093 / sysbio / syp019 . PMID 20525577 .
- ^ Archie JW (septiembre de 1989). "Razones de exceso de homoplasia: nuevos índices para medir los niveles de homoplasia en la sistemática filogenética y una crítica del índice de coherencia". Zoología sistemática . 38 (3): 253–269. doi : 10.2307 / 2992286 . JSTOR 2992286 .
- ^ Wake DB, Wake MH, Specht CD (febrero de 2011). "Homoplasia: de la detección del patrón a la determinación del proceso y mecanismo de evolución". Ciencia . 331 (6020): 1032–5. doi : 10.1126 / science.1188545 . PMID 21350170 . S2CID 26845473 . Resumen de Lay - Science Daily .
- ^ Simpson MG (9 de agosto de 2011). Sistemática de plantas . Elsevier . Amsterdam: Elsevier. ISBN 9780080514048.
- ^ Russell PJ, Hertz PE, McMillan B (2013). Biología: la ciencia dinámica . Aprendizaje Cengage. ISBN 978-1-285-41534-5.
- ^ Lipscomb D (1998). "Conceptos básicos del análisis cladístico" (PDF) . Washington DC: Universidad George Washington.
- ↑ Choudhuri S (9 de mayo de 2014). Bioinformática para principiantes: genes, genomas, evolución molecular, bases de datos y herramientas analíticas (1ª ed.). Prensa académica. pag. 51. ISBN 978-0-12-410471-6. OCLC 950546876 .
- ↑ Williams D, Schmitt M, Wheeler Q (21 de julio de 2016). El futuro de la sistemática filogenética: el legado de Willi Hennig . ISBN 978-1-107-11764-8. OCLC 951563305 .
- ^ Avise JC, Robinson TJ (junio de 2008). "Hemiplasia: un nuevo término en el léxico de la filogenia" . Biología sistemática . 57 (3): 503–7. doi : 10.1080 / 10635150802164587 . PMID 18570042 .
- ^ Copetti D, Búrquez A, Bustamante E, Charboneau JL, Childs KL, Eguiarte LE, et al. (Noviembre de 2017). "Amplia discordancia del árbol de genes y hemiplasia dieron forma a los genomas de cactus columnares de América del Norte" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 114 (45): 12003–12008. doi : 10.1073 / pnas.1706367114 . PMC 5692538 . PMID 29078296 .