
Las condensinas son grandes complejos de proteínas que desempeñan un papel central en el ensamblaje y la segregación de los cromosomas durante la mitosis y la meiosis (Figura 1). [1] [2] Sus subunidades se identificaron originalmente como componentes principales de los cromosomas mitóticos ensamblados en extractos de huevos de Xenopus . [3]
Composición de subunidades
Tipos eucariotas

Muchas células eucariotas poseen dos tipos diferentes de complejos de condensina, conocidos como condensina I y condensina II , cada uno de los cuales se compone de cinco subunidades (Figura 2). [4] Las condensinas I y II comparten el mismo par de subunidades centrales, SMC2 y SMC4, ambas pertenecientes a una gran familia de ATPasas cromosómicas , conocidas como proteínas SMC (SMC significa Mantenimiento estructural de los cromosomas). [5] [6] Cada uno de los complejos contiene un conjunto distinto de subunidades reguladoras no SMC (una subunidad de kleisina [7] y un par de subunidades repetidas HEAT ). [8]Ambos complejos son grandes y tienen una masa molecular total de 650-700 kDa.
| Complejo | Subunidad | Clasificación | Vertebrados | D. melanogaster | C. elegans | S. cerevisiae | S. pombe | A. thaliana | C. merolae | T. thermophila |
|---|---|---|---|---|---|---|---|---|---|---|
| condensina I y II | SMC2 | ATPasa | CAP-E / SMC2 | SMC2 | MEZCLA-1 | Smc2 | Cortar14 | CAP-E1 y -E2 | SMC2 | Scm2 |
| condensina I y II | SMC4 | ATPasa | CAP-C / SMC4 | SMC4 / Gluon | SMC-4 | Smc4 | Cut3 | CAP-C | SMC4 | Smc4 |
| condensin I | CAP-D2 | HEAT repetir | CAP-D2 | CAP-D2 | DPY-28 | Ycs4 | Cnd1 | CAB72176 | CAP-D2 | Cpd1 y 2 |
| condensin I | CAP-G | HEAT repetir | CAP-G | CAP-G | CAP-G1 | Ycg1 | Cnd3 | BAB08309 | CAP-G | Cpg1 |
| condensin I | CAP-H | Kleisin | CAP-H | CAP-H / Estéril | DPY-26 | Brn1 | Cnd2 | AAC25941 | CAP-H | Cph1,2,3,4 y 5 |
| condensina II | CAP-D3 | HEAT repetir | CAP-D3 | CAP-D3 | HCP-6 | - | - | At4g15890.1 | CAP-D3 | - |
| condensina II | CAP-G2 | HEAT repetir | CAP-G2 | - | CAP-G2 | - | - | CAP-G2 / HEB1 | CAP-G2 | - |
| condensina II | CAP-H2 | Kleisin | CAP-H2 | CAP-H2 | KLE-2 | - | - | CAP-H2 / HEB2 | CAP-H2 | - |
| condensin I DC | Variante SMC4 | ATPasa | - | - | DPY-27 | - | - | - | - | - |
Las subunidades centrales condensinas (SMC2 y SMC4) se conservan entre todas las especies eucariotas que se han estudiado hasta la fecha. Las subunidades no SMC exclusivas de la condensina I también se conservan entre los eucariotas, pero la aparición de subunidades no SMC exclusivas de la condensina II es muy variable entre las especies.
- Por ejemplo, la mosca de la fruta Drosophila melanogaster no tiene el gen de la subunidad CAP-G2 de la condensina II. [9] Otras especies de insectos a menudo también carecen de los genes para las subunidades CAP-D3 y / o CAP-H, lo que indica que las subunidades que no son SMC exclusivas de la condensina II han estado sometidas a una alta presión de selección durante la evolución del insecto. [10]
- El nematodo Caenorhabditis elegans posee condensinas I y II. Sin embargo, esta especie es única en el sentido de que tiene un tercer complejo (estrechamente relacionado con la condensina I) que participa en la regulación génica de todo el cromosoma , es decir, la compensación de dosis . [11] En este complejo, conocido como condensina I DC , la subunidad SMC4 auténtica se reemplaza con su variante, DPY-27 (Figura 2).
- Algunas especies, como los hongos (p. Ej., La levadura en ciernes Saccharomyces cerevisiae y la levadura de fisión Schizosaccharomyces pombe ), carecen de todas las subunidades reguladoras exclusivas de la condensina II. [12] [13] Por otro lado, el alga roja primitiva unicelular Cyanidioschyzon merolae , cuyo tamaño de genoma es comparable al de la levadura, tiene condensinas I y II. [14] Por lo tanto, no existe una relación aparente entre la aparición de condensina II y el tamaño de los genomas eucariotas.
- El ciliado Tetrahymena thermophila tiene condensina I solamente. Sin embargo, existen múltiples parálogos para dos de sus subunidades reguladoras (CAP-D2 y CAP-H), y algunos de ellos se localizan específicamente en el macronúcleo (responsable de la expresión génica) o en el micronúcleo (responsable de la reproducción). [15] Por lo tanto, esta especie tiene múltiples complejos de condensina I que tienen diferentes subunidades reguladoras y muestran una localización nuclear distinta. [16] Esta es una propiedad única que no se encuentra en otras especies.
Tipos procariotas
Las especies procariotas también tienen complejos de tipo condensina que desempeñan un papel importante en la organización y segregación de los cromosomas ( nucleoides ). Las condensinas procariotas se pueden clasificar en dos tipos: SMC-ScpAB [17] y MukBEF . [18] Muchas especies de eubacterias y arqueas tienen SMC-ScpAB, mientras que un subgrupo de eubacterias (conocidas como γ-proteobacterias ), incluida Escherichia coli, tiene MukBEF. ScpA y MukF pertenecen a una familia de proteínas llamadas "kleisins", [7] mientras que ScpB y MukF se han clasificado recientemente en una nueva familia de proteínas llamada "kite". [19]
| Complejo | Subunidad | Clasificación | B. subtilis | Caulobacter | E. coli |
|---|---|---|---|---|---|
| SMC-ScpAB | SMC | ATPasa | SMC / BsSMC | SMC | - |
| SMC-ScpAB | ScpA | Kleisin | ScpA | ScpA | - |
| SMC-ScpAB | ScpB | cometa | ScpB | ScpB | - |
| MukBEF | MukB | ATPasa | - | - | MukB |
| MukBEF | MukE | cometa | - | - | MukE |
| MukBEF | MukF | Kleisin | - | - | MukF |
A pesar de las estructuras primarias altamente divergentes de sus subunidades correspondientes entre SMC-ScpAB y MukBEF, es razonable considerar que los dos complejos desempeñan funciones similares, si no idénticas, en la organización y dinámica de los cromosomas procariotas, en función de su arquitectura molecular y sus fenotipos celulares defectuosos. Por tanto, ambos complejos se denominan a menudo condensinas procariotas (o bacterianas). Estudios recientes informan de la aparición de un tercer complejo relacionado con MukBEF (denominado MksBEF) en algunas especies bacterianas. [20]
Mecanismos moleculares
Estructuras moleculares

Los dímeros de SMC que actúan como subunidades centrales de las condensinas muestran una forma de V muy característica, cada brazo de la cual se compone de espirales en espiral antiparalelas (Figura 3; consulte las proteínas SMC para obtener más detalles). [21] [22] La longitud de cada brazo en espiral alcanza ~ 50 nm, que corresponde a la longitud de ~ 150 pb de ADN bicatenario (dsDNA). En los complejos eucariotas de condensina I y II, una subunidad de kleisina une los dos dominios principales de un dímero de SMC y se une a dos subunidades de repetición HEAT (Figura 1). [23] [24]
Los primeros estudios aclararon la estructura de partes de las condensinas bacterianas, como MukBEF [25] [26] y SMC-ScpA. [27] [28] En los complejos eucariotas, se han informado varias estructuras de subcomplejos y subdominios, incluidos los dominios de bisagra y brazo de un dímero SMC2-SMC4, [29] [30] un CAP-G (ycg1) / CAP-H (brn1) subcomplejo, [31] [32] y un subcomplejo CAP-D2 (ycs4) / CAP-H (brn1). [24] Por otro lado, la microscopía de fuerza atómica de alta velocidad ha demostrado que los brazos de un dímero SMC son mucho más flexibles de lo esperado. [33]
Actividades moleculares
Condensina I purificada a partir de Xenopus extractos de huevo es un ADN estimulada ATPasa y muestra la capacidad de introducir positivo tensión superhelical en dsDNA en un ATP (positivo manera -hydrolysis dependiente de superenrollamiento actividad). [34] [35] Se han detectado actividades similares en las condensinas de otros organismos. [36] [37] La actividad de superenrollamiento positivo se activa in vitro mediante la fosforilación de Cdk1 , lo que sugiere que es probable que sea una de las actividades fisiológicas directamente involucradas en el ensamblaje del cromosoma mitótico. [38] Se postula que esta actividad de la condensina I ayuda a plegar el ADN y promueve la resolución de las cromátidas hermanas mediada por la topoisomerasa II . [39] Los primeros experimentos de una sola molécula de ADN también demostraron en tiempo real que la condensina I es capaz de compactar el ADN de una manera dependiente de la hidrólisis de ATP. [40]
Más recientemente, los experimentos de una sola molécula han demostrado que la condensación I de levadura en ciernes es capaz de translocarse a lo largo del dsDNA ( actividad motora ) [41] y "extruir" los bucles de ADN ( actividad de extrusión del bucle ) [42] de una manera dependiente de la hidrólisis del ATP. En los últimos experimentos, la actividad de los complejos de condensina individuales en el ADN se visualizó mediante imágenes de fluorescencia en tiempo real , lo que reveló que la condensina I de hecho es un motor de extrusión de bucle rápido y que un solo complejo de condensina I puede extruir 1.500 pb de ADN por segundo en de una manera estrictamente dependiente de ATP. Se ha propuesto que la condensina I ancla el ADN entre las subunidades Ycg1-Brn1 [31].y tira del ADN asimétricamente para formar grandes bucles. Además, se ha demostrado que los complejos de condensina pueden atravesarse entre sí, formando estructuras de bucle dinámico y cambiando sus tamaños. [43]
Se desconoce cómo podrían actuar las condensinas sobre el ADN nucleosómico . El desarrollo reciente de un sistema de reconstitución ha identificado la histona chaperona FACT como un componente esencial del ensamblaje de cromosomas mediado por condensina I in vitro , lo que proporciona una pista importante para este problema. [44] También se ha demostrado que las condensinas pueden ensamblar estructuras similares a cromosomas en extractos libres de células incluso bajo la condición en la que el ensamblaje de nucleosomas está en gran parte suprimido. [45] Esta observación indica que las condensinas pueden funcionar, al menos en parte, en el ADN no nucleosómico en un entorno fisiológico.
Actualmente, solo se dispone de información limitada sobre la contribución funcional de las subunidades individuales de condensinas a sus actividades. Un dímero SMC2-SMC4 tiene la capacidad de volver a recocer ADN monocatenario complementario . [46] Esta actividad no requiere ATP . Para los complejos eucariotas, se ha informado que las subunidades de repetición HEAT contribuyen en parte a la unión del ADN [31] [47] y al ensamblaje de los ejes cromosómicos. [48] La naturaleza flexible y extensible de las repeticiones HEAT podría ser la base de la acción dinámica de las condensinas y la arquitectura de los cromosomas mitóticos. [49] [50]
Modelado matemático
Se han informado varios intentos de modelado matemático y simulación por computadora del ensamblaje de cromosomas mitóticos, basados en actividades moleculares de las condensinas. Los representativos incluyen el modelado basado en la extrusión de bucle, [51] contactos estocásticos por pares [52] y una combinación de atracciones de bucle e intercondensina. [53]
Funciones en el ensamblaje y la segregación de cromosomas
Mitosis

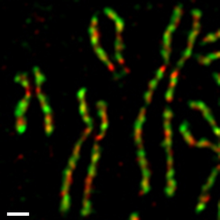
En las células de cultivo de tejidos humanos, los dos complejos de condensina se regulan de manera diferente durante el ciclo celular mitótico (Figura 4). [54] [55] La condensina II está presente dentro del núcleo celular durante la interfase y participa en una etapa temprana de la condensación cromosómica dentro del núcleo de la profase . Por otro lado, la condensina I está presente en el citoplasma durante la interfase y accede a los cromosomas solo después de que la envoltura nuclear se rompe (NEBD) al final de la profase. Durante la prometafase y la metafase, la condensina I y la condensina II cooperan para ensamblar cromosomas en forma de varilla, en los que dos cromátidas hermanas se resuelven completamente. Tales dinámica diferencial de los dos complejos se observa en Xenopus extractos de huevo , [56] ovocitos de ratón, [57] y las células madre neurales, [58] que indica que es parte de un mecanismo regulador fundamental conservada entre diferentes organismos y tipos de células. Lo más probable es que este mecanismo asegure la acción ordenada de los dos complejos, a saber, condensina II primero y condensina I después. [59]
En los cromosomas en metafase , las condensinas I y II se enriquecen en el eje central de forma no superpuesta (Figura 5). Los experimentos de agotamiento in vivo [4] [58] [60] y los experimentos de inmunodepleción en extractos de huevos de Xenopus [56] demuestran que los dos complejos tienen funciones distintas en el ensamblaje de cromosomas en metafase. Las células deficientes en funciones de condensina no se detienen en una etapa específica del ciclo celular, mostrando defectos de segregación cromosómica (es decir, puentes anafase) y progresando a través de una citocinesis anormal. [61] [62]
La contribución relativa de las condensinas I y II a la mitosis varía entre las diferentes especies eucariotas. Por ejemplo, cada una de las condensinas I y II juega un papel esencial en el desarrollo embrionario en ratones. [58] Tienen funciones superpuestas y no superpuestas durante el ciclo celular mitótico. Por otro lado, la condensina II no es esencial para la mitosis en el alga primitiva C. merolae [14] y la planta terrestre A. thaliana . [63] Curiosamente, la condensina II juega un papel dominante sobre la condensina I en los embriones tempranos de C. elegans . [11] Esta peculiaridad podría deberse al hecho de que C. elegans tiene una estructura cromosómica especializada conocida comocromosomas holocéntricos . Los hongos, como S. cerevisiae [13] y S. pombe [12] no tienen condensina II del primero. Estas diferencias entre las especies eucariotas proporcionan importantes implicaciones en la evolución de la arquitectura cromosómica (ver la sección de "Implicaciones evolutivas" más adelante).
| especies | M. musculus | D. melanogaster | C. elegans | S. cerevisiae | S. pombe | A. thaliana | C. merolae |
|---|---|---|---|---|---|---|---|
| tamaño del genoma | ~ 2500 Mb | 140 Mb | 100 Mb | 12 Mb | 14 Mb | 125 Mb | 16 Mb |
| condensin I | esencial | esencial | menor | esencial | esencial | esencial | esencial |
| condensina II | esencial | no esencial | esencial | - | - | no esencial | no esencial |
Recientemente se ha hecho posible que los cambios estructurales de los cromosomas dependientes del ciclo celular se controlen mediante un método basado en la genómica conocido como Hi-C ( captura de conformación cromosómica de alto rendimiento ). [64] El impacto de la deficiencia de condensina en la conformación cromosómica se ha abordado en la levadura en gemación, [65] [66] levadura de fisión, [67] [68] y las células DT40 de pollo. [69]El resultado de estos estudios apoya firmemente la noción de que las condensinas juegan un papel crucial en el ensamblaje de los cromosomas mitóticos y que las condensinas I y II tienen funciones distintas en este proceso. Además, los análisis de imágenes cuantitativas permiten a los investigadores contar el número de complejos de condensina presentes en los cromosomas humanos en metafase. [70]
Meiosis
Las condensinas también juegan un papel importante en el ensamblaje y segregación de cromosomas en la meiosis . Se han informado estudios genéticos en S. cerevisiae , [71] D. melanogaster , [72] [73] y C. elegans . [74] En ratones, los requisitos para las subunidades de condensina en la meiosis se han abordado mediante experimentos de bloqueo mediados por anticuerpos [57] y análisis de desactivación genética condicional . [75] En la meiosis I de mamíferos, la contribución funcional de la condensina II parece mayor que la de la condensina I. Como se ha demostrado en la mitosis, [58]sin embargo, los dos complejos de condensina también tienen funciones superpuestas y no superpuestas en la meiosis. A diferencia de la cohesina , hasta ahora no se han identificado subunidades de condensinas específicas de la meiosis.
Funciones cromosómicas fuera de la mitosis o la meiosis
Estudios recientes han demostrado que las condensinas participan en una amplia variedad de funciones cromosómicas fuera de la mitosis o la meiosis . [59]
- En la levadura en ciernes , la condensina I (la única condensina de este organismo) participa en la regulación del número de copias de la repetición del ADNr [76] , así como en la agrupación de los genes del ARNt . [77]
- En la levadura de fisión , la condensina I participa en la regulación del punto de control replicativo [78] y la agrupación de genes transcritos por la ARN polimerasa III. [79]
- En C. elegans , un tercer complejo de condensina (condensina I DC ) relacionado con la condensina I regula la estructura de orden superior de los cromosomas X como un regulador principal de la compensación de dosis . [80]
- En D. melanogaster , las subunidades de condensina II contribuyen a la disolución de los cromosomas politénicos [81] ya la formación de territorios cromosómicos [82] en las células nodrizas del ovario . Hay evidencia disponible de que regulan negativamente la transvección en células diploides. También se ha informado de que se requieren componentes de condensina I para asegurar la expresión génica correcta en las neuronas después de la salida del ciclo celular. [83]
- En A. thaliana , la condensina II es esencial para la tolerancia del exceso de estrés por boro, posiblemente aliviando el daño del ADN. [63]
- En células de mamíferos, es probable que la condensina II esté implicada en la regulación de la arquitectura y función del cromosoma en interfase. Por ejemplo, en las células humanas, la condensina II participa en el inicio de la resolución de las cromátidas hermanas durante la fase S, mucho tiempo antes de la profase mitótica cuando las cromátidas hermanas se vuelven citológicamente visibles. [84]
- En los núcleos de interfase de ratón , la heterocromatina pericentromérica en diferentes cromosomas se asocia entre sí, formando una gran estructura conocida como cromocentros. Las células deficientes en condensina II, pero no en condensina I, muestran hiperagrupación de cromocentros, lo que indica que la condensina II tiene un papel específico en la supresión de la agrupación de cromocentros. [58]
- Si bien los primeros estudios sugirieron la posibilidad de que las condensinas puedan participar directamente en la regulación de la expresión génica , algunos estudios recientes argumentan en contra de esta hipótesis. [85] [86]
Modificaciones postraduccionales
Las subunidades de condensina se someten a diversas modificaciones postraduccionales de una manera dependiente del ciclo celular. Entre ellos, el ejemplo mejor estudiado es la fosforilación . [87] Por ejemplo, Cdk1 (quinasa dependiente de ciclina 1) activa la condensina I, [38] mientras que CK2 (caseína quinasa 2) regula negativamente su actividad. [88]
| complejo | subunidad | especies | sitio de fosforilación | quinasa | referencia |
|---|---|---|---|---|---|
| condensina I y II | SMC4 | S. pombe | T19 | Cdk1 | [12] |
| S. cerevisiae | muchos | Cdk1 | [89] | ||
| condensin I | CAP-D2 | X. laevis | T1314, T1348, T1353 | Cdk1 | [38] [44] |
| CAP-H | H. sapiens | S570 | CK2 | [88] | |
| H. sapiens | S70 | Aurora B | [90] | ||
| S. pombe | S5, S41, S52 | Aurora B | [90] [91] | ||
| CAP-D2, -G, -H | H. sapiens | - | Aurora B | [92] | |
| S. cerevisiae | muchos | polo / Cdc5 | [37] | ||
| condensina II | CAP-D3 | H. sapiens | T1415 | Cdk1 | [93] |
| H. sapiens | S1419 | Plk1 | [93] | ||
| CAP-G2 | H. sapiens | T1010 (unión PBD) | ? | [94] | |
| CAP-H2 | H. sapiens | S492 | Mps1 | [95] | |
| D. melanogaster | - | CK1α | [96] |
Se ha informado que, en D. melanogaster , la subunidad CAP-H2 de la condensina II se degrada mediante la acción de la ubiquitina ligasa SCF Slimb . [97]
Relevancia para las enfermedades
Se demostró que MCPH1, una de las proteínas responsables de la microcefalia primaria humana , tiene la capacidad de regular negativamente la condensina II. [98] En las células de pacientes con mcph1 , la condensina II (pero no la condensina I) está hiperactivada, lo que lleva a una condensación cromosómica prematura en la fase G2 (es decir, antes de entrar en la mitosis). [99] Sin embargo, no hay evidencia de que la mala regulación de la condensina II esté directamente relacionada con la etiología de la microcefalia mcph1 . Más recientemente, se ha informado que las mutaciones hipomórficas en las subunidades de condensina I o II causan microcefalia en humanos. [100] En ratones, hipomórficomutaciones en Condensina II subunidades causan defectos específicos en las células T de desarrollo, [101] que lleva a linfoma de células T . [102] Es interesante observar que los tipos de células con modos de división celular especializados, como las células madre neurales y las células T , son particularmente susceptibles a las mutaciones en las subunidades de condensina.
Implicaciones evolutivas
Los procariotas tienen tipos primitivos de condensinas, [17] [18] lo que indica que el origen evolutivo de las condensinas precede al de las histonas. El hecho de que las condensinas I y II se conserven ampliamente entre las especies eucariotas existentes implica fuertemente que el último ancestro común eucariota ( LECA ) tenía ambos complejos. [59] Por lo tanto, es razonable especular que algunas especies, como los hongos, han perdido condensina II durante la evolución .
Entonces, ¿por qué muchos eucariotas tienen dos complejos de condensina diferentes? Como se discutió anteriormente, la contribución relativa de las condensinas I y II a la mitosis varía entre diferentes organismos. Desempeñan papeles igualmente importantes en la mitosis de mamíferos, mientras que la condensina I tiene un papel predominante sobre la condensina II en muchas otras especies. En esas especies, la condensina II podría haberse adaptado para diversas funciones no esenciales distintas de la mitosis . [63] [81] Aunque no existe una relación aparente entre la aparición de condensina II y el tamaño de los genomas, parece que la contribución funcional de la condensina II aumenta a medida que aumenta el tamaño del genoma. [14] [58]La contribución relativa de los dos complejos de condensina a la arquitectura del cromosoma mitótico también cambia durante el desarrollo, lo que tiene un impacto en la morfología de los cromosomas mitóticos. [56] Por lo tanto, el acto de equilibrio de las condensinas I y II aparentemente está afinado tanto en la evolución como en el desarrollo.
Familiares
Las células eucariotas tienen dos clases adicionales de complejos de proteínas SMC . Cohesin contiene SMC1 y SMC3 y participa en la cohesión de cromátidas hermanas. El complejo SMC5 / 6 contiene SMC5 y SMC6 y está implicado en la reparación recombinacional.
Ver también
- cromosoma
- nucleoide
- mitosis
- mitosis
- ciclo celular
- cohesin
- Proteína SMC
- ATPasa
- HEAT repetir
- Topoisomerasa II
- Superenrollamiento de ADN
Referencias
- ^ Hirano T (2016). "Organización cromosómica basada en condensina de bacterias a vertebrados" . Celular . 164 (5): 847–857. doi : 10.1016 / j.cell.2016.01.033 . PMID 26919425 .
- ↑ Kalitsis P, Zhang T, Marshall KM, Nielsen CF, Hudson DF (2017). "Condensina, organizador maestro del genoma". Cromosoma Res . 25 (1): 61–76. doi : 10.1007 / s10577-017-9553-0 . PMID 28181049 . S2CID 28241964 . Mantenimiento de CS1: utiliza el parámetro de autores ( enlace )
- ^ Hirano T, Kobayashi R, Hirano M (1997). "Condensinas, complejo de condensación cromosómica que contiene XCAP-C, XCAP-E y un homólogo de Xenopus de la proteína Drosophila Barren" . Celular . 89 (4): 511-21. doi : 10.1016 / S0092-8674 (00) 80233-0 . PMID 9160743 . S2CID 15061740 .
- ↑ a b Ono T, Losada A, Hirano M, Myers MP, Neuwald AF, Hirano T (2003). "Contribuciones diferenciales de condensina I y condensina II a la arquitectura del cromosoma mitótico en células de vertebrados" . Celular . 115 (1): 109–21. doi : 10.1016 / s0092-8674 (03) 00724-4 . PMID 14532007 . S2CID 18811084 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Uhlmann F (2016). "Complejos SMC: del ADN a los cromosomas". Nat. Rev. Mol. Cell Biol . 17 (7): 399–412. doi : 10.1038 / nrm.2016.30 . PMID 27075410 . S2CID 20398243 .
- ^ Yatskevich S, Rhodes J, Nasmyth K (2019). "Organización del ADN cromosómico por complejos SMC" . Annu. Rev. Genet . 53 : 445–482. doi : 10.1146 / annurev-genet-112618-043633 . PMID 31577909 . Mantenimiento de CS1: utiliza el parámetro de autores ( enlace )
- ↑ a b Schleiffer A, Kaitna S, Maurer-Stroh S, Glotzer M, Nasmyth K, Eisenhaber F (2003). "Kleisins: una superfamilia de socios de proteínas SMC bacterianas y eucariotas" . Mol. Celular . 11 (3): 571–5. doi : 10.1016 / S1097-2765 (03) 00108-4 . PMID 12667442 .
- ^ Neuwald AF, Hirano T (2000). "HEAT repite asociadas con condensinas, cohesinas y otros complejos involucrados en funciones relacionadas con los cromosomas" . Genome Res . 10 (10): 1445–52. doi : 10.1101 / gr.147400 . PMC 310966 . PMID 11042144 .
- ^ Herzog S, Nagarkar Jaiswal S, Urban E, Riemer A, Fischer S, Heidmann SK (2013). "La disección funcional de la subunidad Cap-G de condensina de Drosophila melanogaster revela su asociación exclusiva con condensina I" . PLOS Genet . 9 (4): e1003463. doi : 10.1371 / journal.pgen.1003463 . PMC 3630105 . PMID 23637630 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Rey, Thomas D; Leonard, Christopher J; Cooper, Jacob C; Nguyen, hijo; Joyce, Eric F; Phadnis, Nitin; Takahashi, Aya (octubre de 2019). "Pérdidas recurrentes y rápida evolución del complejo Condensina II en insectos" . Biología Molecular y Evolución . 36 (10): 2195–2204. doi : 10.1093 / molbev / msz140 . PMC 6759200 . PMID 31270536 .
- ^ a b Csankovszki G, Collette K, Spahl K, Carey J, Snyder M, Petty E, Patel U, Tabuchi T, Liu H, McLeod I, Thompson J, Sarkeshik A, Yates J, Meyer BJ, Hagstrom K (2009) . "Tres complejos de condensina distintos controlan la dinámica del cromosoma de C. elegans" . Curr. Biol . 19 (1): 9-19. doi : 10.1016 / j.cub.2008.12.006 . PMC 2682549 . PMID 19119011 .
- ↑ a b c Sutani T, Yuasa T, Tomonaga T, Dohmae N, Takio K, Yanagida M (1999). "Complejo de condensina de levadura de fisión: funciones esenciales de las subunidades no SMC para la condensación y la fosforilación de Cdc2 de Cut3 / SMC4" . Genes Dev . 13 (17): 2271–83. doi : 10.1101 / gad.13.17.2271 . PMC 316991 . PMID 10485849 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b Freeman L, Aragon-Alcaide L, Strunnikov A (2000). "El complejo de condensina gobierna la condensación cromosómica y la transmisión mitótica del ADNr" . J. Cell Biol . 149 (4): 811–824. doi : 10.1083 / jcb.149.4.811 . PMC 2174567 . PMID 10811823 .
- ↑ a b c Fujiwara T, Tanaka K, Kuroiwa T, Hirano T (2013). "Dinámica espacio-temporal de condensinas I y II: conocimientos evolutivos de la primitiva alga roja Cyanidioschyzon merolae" . Mol. Biol. Celular . 24 (16): 2515-27. doi : 10.1091 / mbc.E13-04-0208 . PMC 3744952 . PMID 23783031 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ Howard-Till R, Loidl J (2018). "Las condensinas promueven la individualización cromosómica y la segregación durante la mitosis, meiosis y amitosis en Tetrahymena thermophila" . Mol. Biol. Celular . 29 (4): 466–478. doi : 10.1091 / mbc.E17-07-0451 . PMC 6014175 . PMID 29237819 .
- ^ Howard-Till, Rachel; Tian, Miao; Loidl, Josef; Cohen-Fix, Orna (15 de mayo de 2019). "Un complejo de condensina especializado participa en la maduración nuclear somática en" . Biología molecular de la célula . 30 (11): 1326-1338. doi : 10.1091 / mbc.E18-08-0487 . PMC 6724606 . PMID 30893010 .
- ↑ a b Mascarenhas J, Soppa J, Strunnikov AV, Graumann PL (2002). "Localización dependiente del ciclo celular de dos nuevas proteínas de condensación y segregación de cromosomas procarióticos en Bacillus subtilis que interactúan con la proteína SMC" . EMBO J . 21 (12): 3108–18. doi : 10.1093 / emboj / cdf314 . PMC 126067 . PMID 12065423 .
- ↑ a b Yamazoe M, Onogi T, Sunako Y, Niki H, Yamanaka K, Ichimura T, Hiraga S (1999). "Formación de complejos de proteínas MukB, MukE y MukF implicadas en la partición cromosómica en Escherichia coli" . EMBO J . 18 (21): 5873–84. doi : 10.1093 / emboj / 18.21.5873 . PMC 1171653 . PMID 10545099 .
- ^ Palecek JJ, Gruber S (2015). "Proteínas de la cometa: una superfamilia de socios de SMC / kleisin conservados en bacterias, arqueas y eucariotas" . Estructura . 23 (12): 2183–2190. doi : 10.1016 / j.str.2015.10.004 . PMID 26585514 .
- ^ Petrushenko ZM, Ella W, Rybenkov VV (2011). "Una nueva familia de condensinas bacterianas" . Mol. Microbiol . 81 (4): 881–896. doi : 10.1111 / j.1365-2958.2011.07763.x . PMC 3179180 . PMID 21752107 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Melby TE, Ciampaglio CN, Briscoe G, Erickson HP (1998). "La estructura simétrica del mantenimiento estructural de los cromosomas (SMC) y proteínas MukB: bobinas enroscadas antiparalelas largas, plegadas en una bisagra flexible" . J. Cell Biol . 142 (6): 1595–1604. doi : 10.1083 / jcb.142.6.1595 . PMC 2141774 . PMID 9744887 .
- ^ Anderson DE, Losada A, Erickson HP, Hirano T (2002). "La condensación y la cohesina muestran diferentes conformaciones de brazos con ángulos de bisagra característicos" . J. Cell Biol . 156 (6): 419–424. doi : 10.1083 / jcb.200111002 . PMC 2173330 . PMID 11815634 .
- ^ Onn I, Aono N, Hirano M, Hirano T (2007). "Reconstitución y geometría de subunidades de complejos de condensina humana" . EMBO J . 26 (4): 1024–1034. doi : 10.1038 / sj.emboj.7601562 . PMC 1852836 . PMID 17268547 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ a b Hassler M, Shaltiel IA, Kschonsak M, Simon B, Merkel F, Thärichen L, Bailey HJ, Macošek J, Bravo S, Metz J, Hennig J, Haering CH (2019). "Base estructural de un ciclo de ATPasa de condensina asimétrica" . Mol Cell . 74 (6): 1175–1188.e24. doi : 10.1016 / j.molcel.2019.03.037 . PMC 6591010 . PMID 31226277 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Fennell-Fezzie R, Gradia SD, Akey D, Berger JM (2005). "La subunidad MukF de la condensina de Escherichia coli: arquitectura y relación funcional con las kleisinas" . EMBO J . 24 (11): 1921-1930. doi : 10.1038 / sj.emboj.7600680 . PMC 1142612 . PMID 15902272 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Woo JS, Lim JH, Shin HC, Suh MK, Ku B, Lee KH, Joo K, Robinson H, Lee J, Park SY, Ha NC, Oh BH (2009). "Los estudios estructurales de un complejo de condensina bacteriana revelan una alteración dependiente de ATP de las interacciones entre subunidades". Celular . 136 (1): 85–96. doi : 10.1016 / j.cell.2008.10.050 . PMID 19135891 . S2CID 4608756 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Bürmann F, Shin HC, Basquin J, Soh YM, Giménez-Oya V, Kim YG, Oh BH, Gruber S (2013). "Un puente asimétrico SMC-kleisin en condensina procariota". Nat. Struct. Mol. Biol . 20 (3): 371–379. doi : 10.1038 / nsmb.2488 . PMID 23353789 . S2CID 21584205 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Kamada K, Miyata M, Hirano T (2013). "Base molecular de la activación de SMC ATPasa: papel de los cambios estructurales internos del subcomplejo regulador ScpAB" . Estructura . 21 (4): 581–594. doi : 10.1016 / j.str.2013.02.016 . PMID 23541893 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Griese JJ, Witte G, Hopfner KP (2010). "La estructura y la actividad de unión al ADN del dominio de bisagra de condensina de ratón destacan características comunes y diversas de las proteínas SMC" . Ácidos nucleicos Res . 38 (10): 3454–3465. doi : 10.1093 / nar / gkq038 . PMC 2879519 . PMID 20139420 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Soh Y, Bürmann F, Shin H, Oda T, Jin KS, Toseland CP, Kim C, Lee H, Kim SJ, Kong M, Durand-Diebold M, Kim Y, Kim HM, Lee NK, Sato M, Oh B , Gruber S (2015). "Base molecular para la formación de varillas SMC y su disolución al unirse al ADN" . Mol. Celular . 57 (2): 290-303. doi : 10.1016 / j.molcel.2014.11.023 . PMC 4306524 . PMID 25557547 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b c Kschonsak M, Merkel F, Bisht S, Metz J, Rybin V, Hassler M, Haering CH (2017). "Base estructural para un mecanismo de cinturón de seguridad que ancla la condensación a los cromosomas" . Celular . 171 (3): 588–600.e24. doi : 10.1016 / j.cell.2017.09.008 . PMC 5651216 . PMID 28988770 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Hara, Kodai; Kinoshita, Kazuhisa; Migita, Tomoko; Murakami, Kei; Shimizu, Kenichiro; Takeuchi, Kozo; Hirano, Tatsuya; Hashimoto, Hiroshi (12 de marzo de 2019). "Base estructural de las interacciones CALOR-kleisina en el subcomplejo de condensina I humana" . Informes EMBO . 20 (5). doi : 10.15252 / embr.201847183 . PMC 6501013 . PMID 30858338 .
- ^ Eeftens JM, Katan AJ, Kschonsak M, Hassler M, de Wilde L, Dief EM, Haering CH, Dekker C (2016). "Los dímeros Condensin Smc2-Smc4 son flexibles y dinámicos" . Rep . Celular 14 (8): 1813–1818. doi : 10.1016 / j.celrep.2016.01.063 . PMC 4785793 . PMID 26904946 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Kimura K, Hirano T (1997). "Superenrollamiento positivo dependiente de ATP de ADN por condensina 13S: una implicación bioquímica para la condensación cromosómica" . Celular . 90 (4): 625–634. doi : 10.1016 / s0092-8674 (00) 80524-3 . PMID 9288743 . S2CID 15876604 .
- ^ Kimura K, Rybenkov VV, Crisona NJ, Hirano T, Cozzarelli NR (1999). "13S condensin reconfigura activamente el ADN mediante la introducción de retorcimiento global positivo: implicaciones para la condensación cromosómica" . Celular . 98 (2): 239–248. doi : 10.1016 / s0092-8674 (00) 81018-1 . PMID 10428035 . S2CID 16671030 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Hagstrom KA, Holmes VF, Cozzarelli NR, Meyer BJ (2002). "La condensina de C. elegans promueve la arquitectura del cromosoma mitótico, la organización del centrómero y la segregación de las cromátidas hermanas durante la mitosis y la meiosis" . Genes Dev . 16 (6): 729–742. doi : 10.1101 / gad.968302 . PMC 155363 . PMID 11914278 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b St-Pierre J, Douziech M, Bazile F, Pascariu M, Bonneil E, Sauvé V, Ratsima H, D'Amours D (2009). "Polo quinasa regula la condensación del cromosoma mitótico por hiperactivación de la actividad de superenrollamiento del ADN de condensina" . Mol Cell . 120 (Parte 7): 1245-1255. doi : 10.1016 / j.molcel.2009.04.013 . PMID 19481522 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b c Kimura K, Hirano M, Kobayashi R, Hirano T (1998). "Fosforilación y activación de condensina 13S por Cdc2 in vitro". Ciencia . 282 (5388): 487–490. doi : 10.1126 / science.282.5388.487 . PMID 9774278 .
- ^ Baxter J, Sen N, Martínez VL, De Carandini ME, Schvartzman JB, Diffley JF, Aragón L (2011). "El superenrollamiento positivo de ADN mitótico impulsa la decantación por la topoisomerasa II en eucariotas". Ciencia . 331 (6022): 1328-1332. doi : 10.1126 / science.1201538 . PMID 21393545 . S2CID 34081946 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Strick TR, Kawaguchi T, Hirano T (2004). "Detección en tiempo real de la compactación de ADN de una sola molécula por condensina I" . Curr. Biol . 14 (10): 874–880. doi : 10.1016 / j.cub.2004.04.038 . PMID 15186743 . S2CID 10078994 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Terakawa T, Bisht S, Eeftens JM, Dekker C, Haering CH, Greene EC (2017). "El complejo de condensina es un motor mecanoquímico que se desplaza a lo largo del ADN" . Ciencia . 358 (6363): 672–676. doi : 10.1126 / science.aan6516 . PMC 5862036 . PMID 28882993 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Ganji M, Shaltiel IA, Bisht S, Kim E, Kalichava A, Haering CH, Dekker C (2018). "Imágenes en tiempo real de extrusión de bucle de ADN por condensina" . Ciencia . 360 (6384): 102–105. doi : 10.1126 / science.aar7831 . PMC 6329450 . PMID 29472443 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Kim E, Kerssemakers J, Shaltiel IA, Haering CH, Dekker C (2020). "Los complejos de condensina de extrusión de bucle de ADN pueden atravesarse entre sí" . Naturaleza . 579 (7799): 438–442. doi : 10.1038 / s41586-020-2067-5 . PMID 32132705 . S2CID 212407150 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b Shintomi K, Takahashi TS, Hirano T (2015). "Reconstitución de cromátidas mitóticas con un conjunto mínimo de factores purificados". Nat Cell Biol . 17 (8): 1014–1023. doi : 10.1038 / ncb3187 . PMID 26075356 . S2CID 8332012 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Shintomi K, Inoue F, Watanabe H, Ohsumi K, Ohsugi M, Hirano T (2017). "Ensamblaje de cromosomas mitóticos a pesar del agotamiento del nucleosoma en extractos de huevo de Xenopus " . Ciencia . 356 (6344): 1284–1287. doi : 10.1126 / science.aam9702 . PMID 28522692 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Sutani T, Yanagida M (1997). "Actividad de renaturalización del ADN del complejo SMC implicado en la condensación cromosómica". Naturaleza . 388 (6644): 798–801. doi : 10.1038 / 42062 . PMID 9285594 . S2CID 4332572 .
- ↑ Piazza I, Rutkowska A, Ori A, Walczak M, Metz J, Pelechano V, Beck M, Haering CH (2014). "La asociación de condensina con cromosomas depende de la unión del ADN por sus subunidades de repetición HEAT". Nat Struct Mol Biol . 21 (6): 560–568. doi : 10.1038 / nsmb.2831 . PMID 24837193 . S2CID 10741875 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Kinoshita K, Kobayashi TJ, Hirano T (2015). "Actos de equilibrio de dos subunidades de HEAT de condensina apoyo el ensamblaje dinámico de los ejes cromosómicos" . Dev Cell . 33 (1): 94–106. doi : 10.1016 / j.devcel.2015.01.034 . PMID 25850674 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Yoshimura SH, Hirano T (2016). "Repeticiones de HEAT: ¿arreglos versátiles de hélices anfifílicas que trabajan en entornos abarrotados?" . J. Cell Sci . 129 (21): 3963–3970. doi : 10.1242 / jcs.185710 . PMID 27802131 .
- ^ Kappel C, Zachariae U, Dölker N, Grubmüller H (2010). "Un núcleo hidrofóbico inusual confiere una flexibilidad extrema a las proteínas de repetición HEAT" . Biophys. J . 99 (5): 1596–1603. doi : 10.1016 / j.bpj.2010.06.032 . PMC 2931736 . PMID 20816072 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Goloborodko, Anton; Imakaev, Maxim V; Marko, John F; Mirny, Leonid (18 de mayo de 2016). "Compactación y segregación de cromátidas hermanas mediante extrusión de bucle activo" . eLife . 5 . doi : 10.7554 / eLife.14864 . PMC 4914367 . PMID 27192037 .
- ^ Cheng, Tammy MK; Heeger, Sebastian; Chaleil, Raphaël AG; Matthews, Nik; Stewart, Aengus; Wright, Jon; Lim, Carmay; Bates, Paul A; Uhlmann, Frank (29 de abril de 2015). "Un modelo biofísico simple emula la condensación del cromosoma de levadura en ciernes" . eLife . 4 : e05565. doi : 10.7554 / eLife.05565 . PMC 4413874 . PMID 25922992 .
- ^ Sakai, Yuji; Mochizuki, Atsushi; Kinoshita, Kazuhisa; Hirano, Tatsuya; Tachikawa, Masashi; Morozov, Alexandre V. (18 de junio de 2018). "Modelado de las funciones de la condensina en la segregación y conformación de cromosomas" . PLOS Biología Computacional . 14 (6): e1006152. doi : 10.1371 / journal.pcbi.1006152 . PMC 6005465 . PMID 29912867 .
- ^ Ono T, Fang Y, Spector DL, Hirano T (2004). "Regulación espacial y temporal de las condensinas I y II en el ensamblaje de cromosomas mitóticos en células humanas" . Mol. Biol. Celular . 15 (7): 3296-308. doi : 10.1091 / mbc.E04-03-0242 . PMC 452584 . PMID 15146063 .
- ^ Hirota T, Gerlich D, Koch B, Ellenberg J, Peters JM (2004). "Funciones distintas de la condensina I y II en el ensamblaje del cromosoma mitótico" . J. Cell Sci . 117 (Pt 26): 6435–45. doi : 10.1242 / jcs.01604 . PMID 15572404 .
- ↑ a b c Shintomi K, Hirano T (2011). "La relación relativa de condensina I a II determina las formas de los cromosomas" . Genes Dev . 25 (14): 1464-1469. doi : 10.1101 / gad.2060311 . PMC 3143936 . PMID 21715560 .
- ↑ a b Lee J, Ogushi S, Saitou M, Hirano T (2011). "Las condensinas I y II son esenciales para la construcción de cromosomas bivalentes en ovocitos de ratón" . Mol. Biol. Celular . 22 (18): 3465–3477. doi : 10.1091 / mbc.E11-05-0423 . PMC 3172270 . PMID 21795393 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b c d e f Nishide K, Hirano T (2014). "Funciones superpuestas y no superpuestas de condensinas I y II en divisiones de células madre neurales" . PLOS Genet . 10 (12): e1004847. doi : 10.1371 / journal.pgen.1004847 . PMC 4256295 . PMID 25474630 .
- ↑ a b c Hirano T (2012). "Condensinas: organizadores universales de cromosomas con diversas funciones" . Genes Dev . 26 (4): 1659–1678. doi : 10.1101 / gad.194746.112 . PMC 3418584 . PMID 22855829 .
- ^ Verde LC, Kalitsis P, Chang TM, Cipetic M, Kim JH, Marshall O, Turnbull L, Whitchurch CB, Vagnarelli P, Samejima K, Earnshaw WC, Choo KH, Hudson DF (2012). "Funciones contrastantes de la condensina I y la condensina II en la formación de cromosomas mitóticos" . J. Cell Sci . 125 (Pt6): 1591–1604. doi : 10.1242 / jcs.097790 . PMC 3336382 . PMID 22344259 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Saka Y, Sutani T, Yamashita Y, Saitoh S, Takeuchi M, Nakaseko Y, Yanagida M (1994). "La levadura de fisión cut3 y cut14, miembros de una familia de proteínas ubicua, son necesarias para la condensación y segregación de cromosomas en la mitosis" . EMBO J . 13 (20): 4938–4952. doi : 10.1002 / j.1460-2075.1994.tb06821.x . PMC 395434 . PMID 7957061 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Hudson DF, Vagnarelli P, Gassmann R, Earnshaw WC (2003). "La condensina es necesaria para el ensamblaje de proteínas no histonas y la integridad estructural de los cromosomas mitóticos de vertebrados" . Dev. Celular . 5 (2): 323–336. doi : 10.1016 / s1534-5807 (03) 00199-0 . PMID 12919682 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b c Sakamoto T, Inui YT, Uraguchi S, Yoshizumi T, Matsunaga S, Mastui M, Umeda M, Fukui K, Fujiwara T (2011). "La condensina II alivia el daño del ADN y es esencial para la tolerancia del estrés por sobrecarga de boro en Arabidopsis" . Célula vegetal . 23 (9): 3533–3546. doi : 10.1105 / tpc.111.086314 . PMC 3203421 . PMID 21917552 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ Naumova N, Imakaev M, Fudenberg G, Zhan Y, Lajoie BR, Mirny LA, Dekker J (2013). "Organización del cromosoma mitótico" . Ciencia . 342 (6161): 948–953. doi : 10.1126 / science.1236083 . PMC 4040465 . PMID 24200812 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Schalbetter SA, Goloborodko A, Fudenberg G, Belton JM, Miles C, Yu M, Dekker J, Mirny L, Baxter J (2017). "SMC complejos cromosomas mitóticos diferencialmente compactos según contexto genómico" . Nat Cell Biol . 19 (9): 1071–1080. doi : 10.1038 / ncb3594 . PMC 5640152 . PMID 28825700 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Lazar-Stefanita L, Scolari VF, Mercy G, Muller H, Guérin TM, Thierry A, Mozziconacci J, Koszul R (2017). "Cohesinas y condensinas orquestan la dinámica 4D de los cromosomas de levadura durante el ciclo celular" . EMBO J . 36 (18): 2684–2697. doi : 10.15252 / embj.201797342 . PMC 5599795 . PMID 28729434 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Kakui Y, Rabinowitz A, Barry DJ, Uhlmann F (2017). "Remodelación mediada por condensina del paisaje de cromatina mitótica en levadura de fisión" . Nat Genet . 49 (10): 1553-1557. doi : 10.1038 / ng.3938 . PMC 5621628 . PMID 28825727 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ Tanizawa H, Kim KD, Iwasaki O, Noma KI (2017). "Alteraciones arquitectónicas del genoma de la levadura de fisión durante el ciclo celular" . Nat Struct Mol Biol . 24 (11): 965–976. doi : 10.1038 / nsmb.3482 . PMC 5724045 . PMID 28991264 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Gibcus, Johan H .; Samejima, Kumiko; Goloborodko, Anton; Samejima, Itaru; Naumova, Natalia; Nuebler, Johannes; Kanemaki, Masato T .; Xie, Linfeng; Paulson, James R .; Earnshaw, William C .; Mirny, Leonid A .; Dekker, Job (9 de febrero de 2018). "Una vía para la formación de cromosomas mitóticos" . Ciencia . 359 (6376): eaao6135. doi : 10.1126 / science.aao6135 . PMC 5924687 . PMID 29348367 .
- ^ Walther, Nike; Hossain, M. Julius; Politi, Antonio Z .; Koch, Birgit; Kueblbeck, Moritz; Ødegård-Fougner, Øyvind; Lampe, Marko; Ellenberg, Jan (2 de julio de 2018). "Un mapa cuantitativo de las condensinas humanas proporciona nuevos conocimientos sobre la arquitectura del cromosoma mitótico" . Revista de biología celular . 217 (7): 2309–2328. doi : 10.1083 / jcb.201801048 . PMC 6028534 . PMID 29632028 .
- ^ Yu HG, Koshland DE (2003). "Se requiere condensina meiótica para la compactación cromosómica adecuada, el ensamblaje SC y la resolución de los enlaces cromosómicos dependientes de la recombinación" . J. Cell Biol . 163 (5): 937–947. doi : 10.1083 / jcb.200308027 . PMC 2173617 . PMID 14662740 .
- ^ Hartl TA, Sweeney SJ, Knepler PJ, Bosco G (2008). "Condensina II resuelve asociaciones cromosómicas para permitir la segregación anafase I en la meiosis masculina de Drosophila" . PLOS Genet . 4 (10): e1000228. doi : 10.1371 / journal.pgen.1000228 . PMC 2562520 . PMID 18927632 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Resnick TD, Dej KJ, Xiang Y, Hawley RS, Ahn C, Orr-Weaver TL (2009). "Las mutaciones en el complejo pasajero cromosómico y el complejo condensina afectan diferencialmente el desmontaje del complejo sinaptonémico y la configuración de la metafase I en la meiosis femenina de Drosophila" . Genética . 181 (3): 875–887. doi : 10.1534 / genetics.108.097741 . PMC 2651061 . PMID 19104074 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Chan RC, Severson AF, Meyer BJ (2004). "La condensación reestructura los cromosomas en preparación para las divisiones meióticas" . J. Cell Biol . 167 (4): 613–625. doi : 10.1083 / jcb.200408061 . PMC 2172564 . PMID 15557118 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Houlard M, Godwin J, Metson J, Lee J, Hirano T, Nasmyth K (2015). "La condensina confiere la rigidez longitudinal de los cromosomas" . Nat Cell Biol . 17 (6): 771–81. doi : 10.1038 / ncb3167 . PMC 5207317 . PMID 25961503 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Johzuka K, Terasawa M, Ogawa H, Ogawa T, Horiuchi T (2006). "La condensación cargada en el sitio de barrera de la horquilla de replicación en el gen del ARNr se repite durante la fase S de una manera dependiente de FOB1 para evitar la contracción de una matriz repetitiva larga en Saccharomyces cerevisiae" . Mol Cell Biol . 26 (6): 2226–2236. doi : 10.1128 / MCB.26.6.2226-2236.2006 . PMC 1430289 . PMID 16507999 .
- ^ Haeusler RA, Pratt-Hyatt M, Buen PD, Gipson TA, Engelke DR (2008). "La agrupación de genes de ARNt de levadura está mediada por la asociación específica de condensina con complejos de transcripción de genes de ARNt" . Genes Dev . 22 (16): 2204–2214. doi : 10.1101 / gad.1675908 . PMC 2518813 . PMID 18708579 .
- ↑ Aono N, Sutani T, Tomonaga T, Mochida S, Yanagida M (2002). "Cnd2 tiene funciones duales en la condensación mitótica y la interfase". Naturaleza . 417 (6885): 197–202. doi : 10.1038 / 417197a . PMID 12000964 . S2CID 4332524 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ Iwasaki O, Tanaka A, Tanizawa H, Grewal SI, Noma K (2010). "Localización centromérica de genes Pol III dispersos en levadura de fisión" . Mol. Biol. Celular . 21 (2): 254-265. doi : 10.1091 / mbc.e09-09-0790 . PMC 2808234 . PMID 19910488 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Grúa E, Bian Q, McCord RP, Lajoie BR, Wheeler BS, Ralston EJ, Uzawa S, Dekker J, Meyer BJ (2015). "Remodelación impulsada por la condensación de la topología del cromosoma X durante la compensación de la dosis" . Naturaleza . 523 (7559): 210–244. doi : 10.1038 / nature14450 . PMC 4498965 . PMID 26030525 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b Hartl TA, Smith HF, Bosco G (2008). "La alineación y la transvección cromosómicas son antagonizadas por la condensina II". Ciencia . 322 (5906): 1384-1387. doi : 10.1126 / science.1164216 . PMID 19039137 . S2CID 5154197 .
- ^ Bauer CR, Hartl TA, Bosco G (2012). "La condensina II promueve la formación de territorios cromosómicos induciendo la compactación axial de los cromosomas en interfase poliploide" . PLOS Genet . 8 (8): e1002873. doi : 10.1371 / journal.pgen.1002873 . PMC 3431300 . PMID 22956908 .
- ^ Hassan A, Araguás Rodríguez P, Heidmann SK, Walmsley EL, Aughey GN, Southall TD. (2020). "La subunidad Cap-G de la condensina I es esencial para la expresión génica adecuada durante la maduración de las neuronas posmitóticas" . eLife . 9 : e55159. doi : 10.7554 / eLife.55159 . PMC 7170655 . PMID 32255428 . Mantenimiento de CS1: utiliza el parámetro de autores ( enlace )
- ^ Ono T, Yamashita D, Hirano T (2013). "La condensina II inicia la resolución de la cromátida hermana durante la fase S" . J. Cell Biol . 200 (4): 429–441. doi : 10.1083 / jcb.201208008 . PMC 3575537 . PMID 23401001 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ Paul MR, Markowitz TE, Hochwagen A, Ercan S (2018). "El agotamiento de la condensación provoca la descompactación del genoma sin alterar el nivel de expresión génica global en Saccharomyces cerevisiae" . Genética . 210 (1): 331–344. doi : 10.1534 / genetics.118.301217 . PMC 6116964 . PMID 29970489 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Hocquet C, Robellet X, Modolo L, Sun XM, Burny C, Cuylen-Haering S, Toselli E, Clauder-Münster S, Steinmetz L, Haering CH, Marguerat S, Bernard P (2018). "La condensina controla los niveles de ARN celular mediante la segregación precisa de los cromosomas en lugar de regular directamente la transcripción" . eLife . 7 : e38517. doi : 10.7554 / eLife.38517 . PMC 6173581 . PMID 30230473 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Bazile F, St-Pierre J, D'Amours D (2010). "Modelo de tres pasos para la activación de la condensina durante la condensación del cromosoma mitótico" . Ciclo celular . 9 (16): 3243–3255. doi : 10.4161 / cc.9.16.12620 . PMID 20703077 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b Takemoto A, Kimura K, Yanagisawa J, Yokoyama S, Hanaoka F. (2006). "Regulación negativa de condensina I por fosforilación mediada por CK2" . EMBO J . 25 (22): 5339–5348. doi : 10.1038 / sj.emboj.7601394 . PMC 1636611 . PMID 17066080 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Robellet X, Thattikota Y, Wang F, Wee TL, Pascariu M, Shankar S, Bonneil É, Brown CM, D'Amours D (2015). "Un interruptor de fosfo de alta sensibilidad activado por Cdk1 gobierna la morfogénesis de los cromosomas durante la división celular" . Genes Dev . 29 (4): 426–439. doi : 10.1101 / gad.253294.114 . PMC 4335297 . PMID 25691469 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b Tada K, Susumu H, Sakuno T, Watanabe Y. (2011). "La asociación de la condensina con la histona H2A da forma a los cromosomas mitóticos". Naturaleza . 474 (7352): 477–483. doi : 10.1038 / nature10179 . PMID 21633354 . S2CID 205225378 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Nakazawa N, Mehrotra R, Ebe M, Yanagida M. (2011). "La condensina fosforilada por la quinasa Ark1 similar a Aurora-B se requiere continuamente hasta la telofase en un modo distinto de Top2" . J Cell Sci . 124 (Parte 11): 1795–1807. doi : 10.1242 / jcs.078733 . PMID 21540296 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Lipp JJ, Hirota T, Poser I, Peters JM (2007). "Aurora B controla la asociación de condensina I pero no condensina II con cromosomas mitóticos" . J Cell Sci . 120 (Parte 7): 1245-1255. doi : 10.1242 / jcs.03425 . PMID 17356064 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ a b Abe S, Nagasaka K, Hirayama Y, Kozuka-Hata H, Oyama M, Aoyagi Y, Obuse C, Hirota T (2011). "La fase inicial de la condensación cromosómica requiere la fosforilación mediada por Cdk1 de la subunidad CAP-D3 de la condensina II" . Genes Dev . 25 (8): 863–874. doi : 10.1101 / gad.2016411 . PMC 3078710 . PMID 21498573 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Kim JH, Shim J, Ji MJ, Jung Y, Bong SM, Jang YJ, Yoon EK, Lee SJ, Kim KG, Kim YH, Lee C, Lee BI, Kim KT (2014). "El componente de condensina NCAPG2 regula la unión microtúbulo-cinetocoro mediante el reclutamiento de la quinasa 1 similar a Polo a los cinetocoros" . Nat Commun . 5 : 4588. doi : 10.1038 / ncomms5588 . PMID 25109385 . CS1 maint: varios nombres: lista de autores ( enlace )
- ↑ Kagami Y, Nihira K, Wada S, Ono M, Honda M, Yoshida K (2014). "La fosforilación de Mps1 de condensina II controla la condensación del cromosoma en el inicio de la mitosis" . J. Cell Biol . 205 (6): 781–790. doi : 10.1083 / jcb.201308172 . PMC 4068140 . PMID 24934155 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Nguyen HQ, Nye J, Buster DW, Klebba JE, Rogers GC, Bosco G (2015). "La caseína quinasa I alfa de Drosophila regula el emparejamiento de homólogos y la organización del genoma modulando los niveles de la subunidad Cap-H2 de la condensina II" . PLOS Genet . 11 (2): e1005014. doi : 10.1371 / journal.pgen.1005014 . PMC 4344196 . PMID 25723539 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Buster DW, Daniel SG, Nguyen HQ, Windler SL, Skwarek LC, Peterson M, Roberts M, Meserve JH, Hartl T, Klebba JE, Bilder D, Bosco G, Rogers GC (2013). "SCFSlimb ubiquitin ligasa suprime la reorganización nuclear mediada por condensina II degradando Cap-H2" . J. Cell Biol . 201 (1): 49–63. doi : 10.1083 / jcb.201207183 . PMC 3613687 . PMID 23530065 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Yamashita D, Shintomi K, Ono T, Gavvovidis I, Schindler D, Neitzel H, Trimborn M, Hirano T (2011). "MCPH1 regula la condensación de cromosomas y la forma como un modulador compuesto de condensina II" . J. Cell Biol . 194 (6): 841–854. doi : 10.1083 / jcb.201106141 . PMC 3207293 . PMID 21911480 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Trimborn M, Schindler D, Neitzel H, Hirano T (2006). "La condensación cromosómica mal regulada en la microcefalia primaria MCPH1 está mediada por la condensina II" . Ciclo celular . 5 (3): 322–326. doi : 10.4161 / cc.5.3.2412 . PMID 16434882 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Martin CA, Murray JE, Carroll P, Leitch A, Mackenzie KJ, Halachev M, Fetit AE, Keith C, Bicknell LS, Fluteau A, Gautier P, Hall EA, Joss S, Soares G, Silva J, Bober MB, Duker A, Wise CA, Quigley AJ, Phadke SR, The Deciphering Developmental Disorders Study., Wood AJ, Vagnarelli P, Jackson AP (2016). "Las mutaciones en los genes que codifican proteínas del complejo de condensina causan microcefalia a través de un fallo de decantación en la mitosis" . Genes Dev . 30 (19): 2158–2172. doi : 10.1101 / gad.286351.116 . PMC 5088565 . PMID 27737959 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Gosling KM, Makaroff LE, Theodoratos A, Kim YH, Whittle B, Rui L, Wu H, Hong NA, Kennedy GC, Fritz JA, Yates AL, Goodnow CC, Fahrer AM (2007). "Una mutación en una subunidad de la condensina II del cromosoma, kleisina beta, interrumpe específicamente el desarrollo de las células T" . Proc. Natl. Acad. Sci. USA . 104 (30): 12445-12450. doi : 10.1073 / pnas.0704870104 . PMC 1941488 . PMID 17640884 . CS1 maint: varios nombres: lista de autores ( enlace )
- ^ Woodward J, Taylor GC, Soares DC, Boyle S, Sie D, Read D, Chathoth K, Vukovic M, Tarrats N, Jamieson D, Campbell KJ, Blyth K, Acosta JC, Ylstra B, Arends MJ, Kranc KR, Jackson AP, Bickmore WA, Wood AJ (2016). "La mutación de condensina II causa linfoma de células T a través de la inestabilidad del genoma específico de tejido" . Genes Dev . 30 (19): 2173–2186. doi : 10.1101 / gad.284562.116 . PMC 5088566 . PMID 27737961 . CS1 maint: varios nombres: lista de autores ( enlace )
Enlaces externos
- condensina en los encabezamientos de materias médicas de la Biblioteca Nacional de Medicina de EE. UU. (MeSH)