Hibridación in situ fluorescente
La hibridación in situ con fluorescencia ( FISH ) es una técnica de citogenética molecular que utiliza sondas fluorescentes que se unen sólo a partes particulares de una secuencia de ácido nucleico con un alto grado de complementariedad de secuencia . Fue desarrollado por investigadores biomédicos a principios de la década de 1980 [1] para detectar y localizar la presencia o ausencia de secuencias de ADN específicas en los cromosomas . La microscopía de fluorescencia se puede utilizar para averiguar dónde se une la sonda fluorescente a los cromosomas. FISH se usa a menudo para encontrar características específicas en el ADN para su uso enasesoramiento genético , medicina e identificación de especies. [2] FISH también se puede utilizar para detectar y localizar objetivos de ARN específicos ( mRNA , lncRNA y miRNA ) [ cita requerida ] en células, células tumorales circulantes y muestras de tejido. En este contexto, puede ayudar a definir los patrones espacio-temporales de expresión génica dentro de células y tejidos.
En biología, una sonda es una sola hebra de ADN o ARN que es complementaria a una secuencia de nucleótidos de interés.
Las sondas de ARN se pueden diseñar para cualquier gen o cualquier secuencia dentro de un gen para la visualización de ARNm , [3] [4] [5] lncRNA [6] [7] [8] y miARN en tejidos y células. FISH se usa examinando el ciclo de reproducción celular, específicamente la interfase de los núcleos para detectar anomalías cromosómicas. [9] FISH permite el análisis de una gran serie de casos archivados mucho más fácil de identificar el cromosoma identificado mediante la creación de una sonda con una base cromosómica artificial que atraerá cromosomas similares. [9] Las señales de hibridación para cada sonda cuando se detecta una anomalía nucleica. [9]Cada sonda para la detección de mRNA y lncRNA se compone de aproximadamente 20 a 50 pares de oligonucleótidos, y cada par cubre un espacio de 40 a 50 pb. Los detalles dependen de la técnica FISH específica utilizada. Para la detección de miARN, las sondas usan química patentada para la detección específica de miARN y cubren toda la secuencia de miARN.
Las sondas a menudo se derivan de fragmentos de ADN que fueron aislados, purificados y amplificados para su uso en el Proyecto Genoma Humano . El tamaño del genoma humano es tan grande, comparado con la longitud que podría secuenciarse directamente, que fue necesario dividir el genoma en fragmentos. (En el análisis final, estos fragmentos se ordenaron digiriendo una copia de cada fragmento en fragmentos aún más pequeños usando endonucleasas específicas de secuencia, midiendo el tamaño de cada fragmento pequeño usando cromatografía de exclusión por tamaño)., y usar esa información para determinar dónde se superponen los fragmentos grandes). Para preservar los fragmentos con sus secuencias de ADN individuales, los fragmentos se agregaron a un sistema de poblaciones de bacterias que se replican continuamente. Las poblaciones clonales de bacterias, cada una de las cuales mantiene un único cromosoma artificial, se almacenan en varios laboratorios de todo el mundo. Los cromosomas artificiales ( BAC ) se pueden cultivar, extraer y etiquetar en cualquier laboratorio que contenga una biblioteca. Las bibliotecas genómicas a menudo reciben el nombre de la institución en la que se desarrollaron. Un ejemplo es la biblioteca RPCI-11, que lleva el nombre de Roswell Park Comprehensive Cancer Center (anteriormente conocido como Roswell Park Cancer Institute) en Buffalo, Nueva York.. Estos fragmentos son del orden de 100 mil pares de bases y son la base para la mayoría de las sondas FISH.
El propósito de usar RNA FISH es detectar transcritos de mRNA objetivo en células, secciones de tejido o incluso montajes completos. [10] El proceso se realiza en 3 procedimientos principales: preparación del tejido (pre-hibridación), hibridación y lavado (post-hibridación).

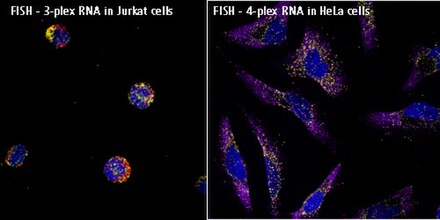

.jpg/440px-FISH_(Fluorescent_In_Situ_Hybridization).jpg)