visualización de ARNm
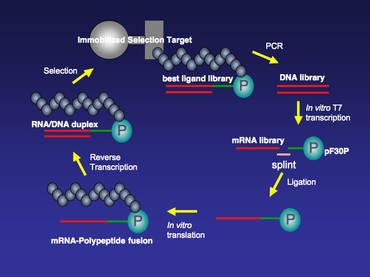
La visualización de ARNm es una técnica de visualización utilizada para la evolución de proteínas y/o péptidos in vitro para crear moléculas que puedan unirse a un objetivo deseado. El proceso da como resultado péptidos o proteínas traducidos que están asociados con su progenitor de ARNm a través de un enlace de puromicina . Luego, el complejo se une a un objetivo inmovilizado en un paso de selección ( cromatografía de afinidad ). Las fusiones de mRNA-proteína que se unen bien se transcriben de forma inversa a cDNA y su secuencia se amplifica mediante una reacción en cadena de la polimerasa . El resultado es un nucleótido.secuencia que codifica un péptido con alta afinidad por la molécula de interés.
La puromicina es un análogo del extremo 3' de un tirosil-tRNA con una parte de su estructura que imita una molécula de adenosina y la otra parte que imita una molécula de tirosina . En comparación con el enlace éster escindible en un tirosil-tRNA, la puromicina tiene un enlace amida no hidrolizable. Como resultado, la puromicina interfiere con la traducción y provoca la liberación prematura de los productos de traducción.
Todas las plantillas de ARNm utilizadas para la tecnología de visualización de ARNm tienen puromicina en su extremo 3'. A medida que avanza la traducción, el ribosoma se mueve a lo largo de la plantilla de ARNm y, una vez que alcanza el extremo 3' de la plantilla, la puromicina fusionada entrará en el sitio A del ribosoma y se incorporará al péptido naciente. La fusión de ARNm-polipéptido luego se libera del ribosoma (Figura 1).
Para sintetizar una fusión de ARNm-polipéptido, la puromicina fusionada no es la única modificación de la plantilla de ARNm. [1] Es necesario reclutar oligonucleótidos y otros espaciadores junto con la puromicina para proporcionar flexibilidad y la longitud adecuada para que la puromicina ingrese al sitio A. Idealmente, el conector entre el extremo 3' de un ARNm y la puromicina tiene que ser lo suficientemente flexible y largo para permitir que la puromicina entre en el sitio A tras la traducción del último codón. Esto permite la producción eficiente de una fusión de ARNm-polipéptido de longitud completa y alta calidad. Rihe Liu et al.optimizó el espaciador de oligonucleótidos 3'-puromicina. Informaron que dA25 en combinación con un Spacer 9 (Glen Research) y dAdCdCP en el extremo 5' funcionaron mejor para la reacción de fusión. Descubrieron que los enlazadores de más de 40 nucleótidos y de menos de 16 nucleótidos mostraban una eficacia muy reducida en la formación de fusiones. Además, cuando la secuencia rUrUP se presentaba junto a la puromicina, la fusión no se formaba de forma eficiente. [2]
Además de proporcionar flexibilidad y longitud, la porción poli dA del enlazador también permite una mayor purificación de la fusión de ARNm-polipéptido debido a su alta afinidad por la resina de celulosa dT. [3] Las fusiones de ARNm-polipéptido se pueden seleccionar sobre objetivos de selección inmovilizados durante varias rondas con una rigurosidad creciente. Después de cada ronda de selección, los miembros de la biblioteca que permanecen unidos al objetivo inmovilizado se amplifican por PCR y los que no se unen se eliminan por lavado.
La síntesis de una biblioteca de presentación de ARNm comienza a partir de la síntesis de una biblioteca de ADN. Se puede sintetizar una biblioteca de ADN para cualquier proteína o péptido pequeño de interés mediante síntesis en fase sólida seguida de amplificación por PCR. Por lo general, cada miembro de esta biblioteca de ADN tiene un sitio de transcripción de ARN polimerasa T7 y un sitio de unión al ribosoma en el extremo 5'. La región promotora de T7 permite la transcripción de T7 in vitro a gran escala para transcribir la biblioteca de ADN en una biblioteca de ARNm, que proporciona plantillas para la reacción de traducción in vitro posterior. El sitio de unión al ribosoma en la región 5' no traducida (5'UTR) se diseña de acuerdo con el sistema de traducción in vitro que se va a utilizar. Hay dos populares disponibles comercialmente in vitrosistemas de traducción. Uno es E. Coli S30 Extract System (Promega) que requiere una secuencia Shine-Dalgarno en la UTR 5' como sitio de unión al ribosoma; [4] el otro es Red Nova Lysate (Novagen), que necesita un sitio de unión ribosomal ΔTMV.