Coenzima Q - citocromo c reductasa
| UCR_TM | ||||||||
|---|---|---|---|---|---|---|---|---|
 Estructura cristalina del complejo citocromo bc mitocondrial unido con ubiquinona . [1] | ||||||||
| Identificadores | ||||||||
| Símbolo | UCR_TM | |||||||
| Pfam | PF02921 | |||||||
| InterPro | IPR004192 | |||||||
| SCOP2 | 1be3 / SCOPe / SUPFAM | |||||||
| TCDB | 3.D.3 | |||||||
| Superfamilia OPM | 92 | |||||||
| Proteína OPM | 3cx5 | |||||||
| Membranome | 258 | |||||||
| ||||||||
| ubiquinol — citocromo-c reductasa | ||||||||
|---|---|---|---|---|---|---|---|---|
| Identificadores | ||||||||
| CE no. | 7.1.1.8 | |||||||
| No CAS. | 9027-03-6 | |||||||
| Bases de datos | ||||||||
| IntEnz | Vista IntEnz | |||||||
| BRENDA | Entrada BRENDA | |||||||
| FÁCIL | NiceZyme vista | |||||||
| KEGG | Entrada KEGG | |||||||
| MetaCyc | camino metabólico | |||||||
| PRIAM | perfil | |||||||
| Estructuras PDB | RCSB PDB PDBe PDBsum | |||||||
| Ontología de genes | AmiGO / QuickGO | |||||||
| ||||||||
La coenzima Q: citocromo c - oxidorreductasa , a veces llamado el citocromo bc 1 complejo , y en otros momentos el complejo III , es el tercer complejo en la cadena de transporte de electrones ( EC 1.10.2.2 ), que juega un papel crítico en la generación de bioquímica de ATP ( fosforilación oxidativa ). El complejo III es una proteína transmembrana de múltiples subunidades codificada por los genomas mitocondrial ( citocromo b ) y nuclear (todas las demás subunidades). El complejo III está presente en las mitocondrias de todos los animales y todos los eucariotas aeróbicos y en las membranas internas de la mayoríaeubacterias . Las mutaciones en el Complejo III causan intolerancia al ejercicio , así como trastornos multisistémicos. El BC1 complejo contiene 11 subunidades, 3 respiratorias subunidades (citocromo b, citocromo C1, proteína Rieske), 2 de núcleo proteínas y 6 de bajo peso molecular proteínas .
Ubiquinol: la citocromo-c reductasa cataliza la reacción química.
- QH 2 + 2 ferricitocromo c Q + 2 ferrocitocromo c + 2 H +
Así, los dos sustratos de esta enzima son quinol (QH 2 ) y ferri- (Fe 3+ ) citocromo c , mientras que sus 3 productos son quinona (Q), ferro- (Fe 2+ ) citocromo cy H + .
Esta enzima pertenece a la familia de las oxidorreductasas , concretamente las que actúan sobre difenoles y sustancias afines como donante con un citocromo como aceptor. Esta enzima participa en la fosforilación oxidativa . Tiene cuatro cofactores : citocromo c 1 , citocromo b-562, citocromo b-566 y una ferredoxina 2-hierro del tipo Rieske .
Nomenclatura
El nombre sistemático de esta clase de enzimas es ubiquinol: ferricitocromo-c oxidorreductasa . Otros nombres de uso común incluyen:
|
|
Estructura
En comparación con las otras subunidades principales de bombeo de protones de la cadena de transporte de electrones , el número de subunidades encontradas puede ser pequeño, tan pequeño como tres cadenas polipeptídicas. Este número aumenta y se encuentran once subunidades en animales superiores. [2] Tres subunidades tienen grupos protésicos . El citocromo b subunidad tiene dos b de tipo hemes ( b L y b H ), el citocromo c subunidad tiene uno c de tipo hemo ( c 1 ), y la subunidad de la proteína de azufre Rieske de hierro (ISP) tiene un hierro dos, dos de azufregrupo de hierro-azufre (2Fe • 2S).
Estructuras de complejo III: PDB : 1KYO , PDB : 1L0L
Composición de complejo
En los vertebrados, el complejo bc 1 , o Complejo III, contiene 11 subunidades: 3 subunidades respiratorias, 2 proteínas centrales y 6 proteínas de bajo peso molecular. [3] [4] Los complejos proteobacterianos pueden contener tan solo tres subunidades. [5]
Tabla de composición de subunidades del complejo III
| No. | Nombre de la subunidad | Proteína humana | Descripción de proteínas de UniProt | Familia Pfam con proteína humana |
|---|---|---|---|---|
| Proteínas de subunidades respiratorias | ||||
| 1 | MT-CYB / Cyt b | CYB_HUMAN | Citocromo b | Pfam PF13631 |
| 2 | CYC1 / Cyt c1 | CY1_HUMAN | Citocromo c1, proteína hemo, mitocondrial | Pfam PF02167 |
| 3 | Rieske / UCR1 | UCRI_HUMAN | Subunidad del complejo del citocromo b-c1 Rieske, EC mitocondrial 1.10.2.2 | Pfam PF02921 , Pfam PF00355 |
| Subunidades de proteínas centrales | ||||
| 4 | QCR1 / SU1 | QCR1_HUMAN | Subunidad 1 del complejo del citocromo b-c1, mitocondrial | Pfam PF00675 , Pfam PF05193 |
| 5 | QCR2 / SU2 | QCR2_HUMAN | Subunidad 2 del complejo del citocromo b-c1, mitocondrial | Pfam PF00675 , Pfam PF05193 |
| Subunidades de proteínas de bajo peso molecular | ||||
| 6 | QCR6 / SU6 | QCR6_HUMAN | Subunidad 6 del complejo del citocromo b-c1, mitocondrial | Pfam PF02320 |
| 7 | QCR7 / SU7 | QCR7_HUMAN | Subunidad 7 del complejo del citocromo b-c1 | Pfam PF02271 |
| 8 | QCR8 / SU8 | QCR8_HUMAN | Subunidad 8 del complejo del citocromo b-c1 | Pfam PF02939 |
| 9 | QCR9 / SU9 / UCRC | QCR9_HUMAN a | Subunidad 9 del complejo del citocromo b-c1 | Pfam PF09165 |
| 10 | QCR10 / SU10 | QCR10_HUMAN | Subunidad 10 del complejo del citocromo b-c1 | Pfam PF05365 |
| 11 | QCR11 / SU11 | QCR11_HUMAN | Subunidad 11 del complejo del citocromo b-c1 | Pfam PF08997 |
- a En los vertebrados, un producto de escisión de 8 kDa del extremo N de la proteína Rieske ( péptido señal ) se retiene en el complejo como subunidad 9. Por tanto, las subunidades 10 y 11 corresponden a los hongos QCR9p y QCR10p.
Reacción
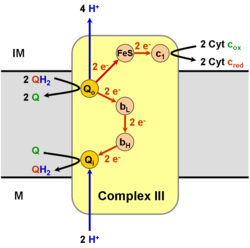
Cataliza la reducción del citocromo c por oxidación de la coenzima Q (CoQ) y el bombeo concomitante de 4 protones desde la matriz mitocondrial al espacio intermembrana:
- QH 2 + 2 citocromo c (Fe III ) + 2 H+
en→ Q + 2 citocromo c (Fe II ) + 4 H+
fuera
En el proceso llamado ciclo Q , [6] [7] se consumen dos protones de la matriz (M), se liberan cuatro protones en el espacio entre membranas (IM) y dos electrones se pasan al citocromo c .
Mecanismo de reacción
El mecanismo de reacción del complejo III (citocromo bc1, coenzima Q: citocromo C oxidorreductasa) se conoce como ciclo de ubiquinona ("Q"). En este ciclo, cuatro protones se liberan en el lado positivo "P" (espacio entre membranas), pero solo dos protones se toman del lado negativo "N" (matriz). Como resultado, se forma un gradiente de protones a través de la membrana. En la reacción general, dos ubiquinol se oxidan a ubiquinonas y una ubiquinona se reduce a ubiquinol . En el mecanismo completo, dos electrones se transfieren de ubiquinol a ubiquinona, a través de dos intermedios del citocromo c.
General :
- 2 x QH 2 oxidado a Q
- 1 x Q reducido a QH 2
- 2 x Cyt c reducido
- 4 x H + liberado en el espacio intermembrana
- 2 x H + recogido de la matriz
La reacción procede de acuerdo con los siguientes pasos:
Ronda 1 :
- El citocromo se une a un ubiquinol y una ubiquinona.
- El centro 2Fe / 2S y el hemo B L extraen cada uno un electrón del ubiquinol unido, liberando dos hidrógenos en el espacio intermembrana.
- Se transfiere un electrón al citocromo c 1 desde el centro 2Fe / 2S, mientras que otro se transfiere desde el B L heme a la B H hemo.
- El citocromo c 1 transfiere su electrón al citocromo c (que no debe confundirse con el citocromo c1) y el B H Heme transfiere su electrón a una ubiquinona cercana, lo que da lugar a la formación de una ubisemiquinona.
- El citocromo c se difunde. Se libera el primer ubiquinol (ahora oxidado a ubiquinona), mientras que la semiquinona permanece unida.
Ronda 2 :
- Un segundo ubiquinol se une al citocromo b.
- El centro 2Fe / 2S y el hemo B L extraen cada uno un electrón del ubiquinol unido, liberando dos hidrógenos en el espacio intermembrana.
- Se transfiere un electrón al citocromo c 1 desde el centro 2Fe / 2S, mientras que otro se transfiere desde el B L heme a la B H hemo.
- El citocromo c 1 luego transfiere su electrón al citocromo c , mientras que la semiquinona cercana producida en la ronda 1 recoge un segundo electrón del hemo B H , junto con dos protones de la matriz.
- Se libera el segundo ubiquinol (ahora oxidado a ubiquinona), junto con el ubiquinol recién formado. [8]
Inhibidores del complejo III
Hay tres grupos distintos de inhibidores del complejo III.
- La antimicina A se une al sitio Q i e inhibe la transferencia de electrones en el Complejo III desde el hem b H al Q oxidado (inhibidor del sitio Qi).
- El mixotiazol y la estigmatelina se unen al sitio Q o e inhiben la transferencia de electrones desde el QH 2 reducido a la proteína de azufre de hierro de Rieske. El mixotiazol y la estigmatelina se unen a bolsas distintas pero superpuestas dentro del sitio Q o .
- El mixotiazol se une más cerca al citocromo bL (de ahí que se lo denomine inhibidor " proximal ").
- La estigmatelina se une más lejos del hemo bL y más cerca de la proteína de azufre de hierro de Rieske, con la que interactúa fuertemente.
Algunos se han comercializado como fungicidas (los derivados de la estrobilurina , el más conocido de los cuales es la azoxistrobina ; inhibidores de QoI ) y como agentes antipalúdicos ( atovacuona ).
Además, la propilhexedrina inhibe la citocromo c reductasa. [9]
Radicales libres de oxígeno
Una pequeña fracción de electrones abandona la cadena de transporte de electrones antes de llegar al complejo IV . La fuga prematura de electrones al oxígeno da como resultado la formación de superóxido . La relevancia de esta reacción secundaria, por lo demás menor, es que el superóxido y otras especies reactivas del oxígeno son altamente tóxicas y se cree que desempeñan un papel en varias patologías, así como en el envejecimiento (la teoría de los radicales libres del envejecimiento ). [10] Electrón fuga se produce principalmente en la Q o sitio y es estimulada por A antimicina . La antimicina A bloquea la bhemas en el estado reducido impidiendo su re-oxidación en el sitio Q i , lo que, a su vez, hace que aumenten las concentraciones en estado estacionario de la semiquinona Q o , reaccionando esta última especie con el oxígeno para formar superóxido . Se cree que el efecto del alto potencial de membrana tiene un efecto similar. [11] El superóxido producido en el sitio Qo puede liberarse tanto en la matriz mitocondrial [12] [13] como en el espacio intermembrana, donde luego puede llegar al citosol. [12] [14] Esto podría explicarse por el hecho de que el Complejo III podría producir superóxido como HOO permeable a la membrana• en lugar de O impermeable a la membrana-. 2. [13]
Nombres de genes humanos
MT-CYB : citocromo b codificado por ADNmt ; mutaciones asociadas con la intolerancia al ejercicio
CYC1 : citocromo c1
CYCS : citocromo c
UQCRFS1 : proteína de azufre de hierro de Rieske
UQCRB : proteína de unión de ubiquinona, mutación ligada a la deficiencia del complejo mitocondrial III nuclear tipo 3
UQCRH : proteína bisagra
UQCRC2 : Core 2, mutaciones relacionadas con la deficiencia del complejo mitocondrial III, tipo nuclear 5
UQCRC1 : Núcleo 1
UQCR : subunidad 6.4KD
UQCR10 : subunidad de 7.2KD
TTC19 : subunidad recientemente identificada, mutaciones vinculadas a la deficiencia del complejo III nuclear tipo 2.
Mutaciones en genes del complejo III en enfermedades humanas
Las mutaciones en genes relacionados con el complejo III se manifiestan típicamente como intolerancia al ejercicio. [15] [16] Se notificó que otras mutaciones causan displasia septoóptica [17] y trastornos multisistémicos. [18] Sin embargo, las mutaciones en BCS1L , un gen responsable de la maduración adecuada del complejo III, pueden provocar el síndrome de Björnstad y el síndrome GRACILE , que en los recién nacidos son afecciones letales que tienen manifestaciones neurológicas y multisistémicas que tipifican trastornos mitocondriales graves. La patogenicidad de varias mutaciones se ha verificado en sistemas modelo como la levadura. [19]
Actualmente se desconoce hasta qué punto estas diversas patologías se deben a déficits bioenergéticos o sobreproducción de superóxido .
Ver también
- Respiración celular
- Centro de reacción fotosintética
Imágenes Adicionales
ETC
Referencias
- ^ PDB : 1 nz ; Gao X, Wen X, Esser L, Quinn B, Yu L, Yu CA, Xia D (agosto de 2003). "Base estructural para la reducción de quinonas en el complejo bc1: un análisis comparativo de las estructuras cristalinas del citocromo bc1 mitocondrial con sustrato unido e inhibidores en el sitio Qi". Bioquímica . 42 (30): 9067–80. doi : 10.1021 / bi0341814 . PMID 12885240 .
- ^ Iwata S, Lee JW, Okada K, Lee JK, Iwata M, Rasmussen B, Link TA, Ramaswamy S, Jap BK (julio de 1998). "Estructura completa del complejo de citocromo bc1 mitocondrial bovino de 11 subunidades". Ciencia . 281 (5373): 64–71. Código Bibliográfico : 1998Sci ... 281 ... 64I . doi : 10.1126 / science.281.5373.64 . PMID 9651245 .
- ^ Zhang Z, Huang L, Shulmeister VM, Chi YI, Kim KK, Hung LW, et al. (1998). "Transferencia de electrones por movimiento de dominio en el citocromo bc1". Naturaleza . 392 (6677): 677–84. Código Bibliográfico : 1998Natur.392..677Z . doi : 10.1038 / 33612 . PMID 9565029 . S2CID 4380033 .
- ^ Hao GF, Wang F, Li H, Zhu XL, Yang WC, Huang LS, et al. (2012). "Descubrimiento computacional de inhibidores del sitio picomolar Q (o) del complejo citocromo bc1". J Am Chem Soc . 134 (27): 11168–76. doi : 10.1021 / ja3001908 . PMID 22690928 .
- ^ Yang XH, Trumpower BL (1986). "Purificación de un complejo de tres subunidades ubiquinol-citocromo c oxidorreductasa de Paracoccus denitrificans" . J Biol Chem . 261 (26): 12282–9. doi : 10.1016 / S0021-9258 (18) 67236-9 . PMID 3017970 .
- ^ Kramer DM, Roberts AG, Muller F, Cabo J, Bowman MK (2004). "Reacciones de bypass del ciclo Q en el sitio Qo de los complejos del citocromo bc1 (y relacionados)". Quiñones y quinona enzimas, Parte B . Meth. Enzymol . Métodos en enzimología. 382 . págs. 21–45. doi : 10.1016 / S0076-6879 (04) 82002-0 . ISBN 978-0-12-182786-1. PMID 15047094 .
- ^ Crofts AR (2004). "El complejo citocromo bc1: función en el contexto de la estructura". Annu. Rev. Physiol . 66 : 689–733. doi : 10.1146 / annurev.physiol.66.032102.150251 . PMID 14977419 .
- ^ Ferguson SJ, Nicholls D, Ferguson S (2002). Bioenergética (3ª ed.). San Diego: Académico. págs. 114-117. ISBN 978-0-12-518121-1.
- ^ Holmes, JH; Sapeika, N; Zwarenstein, H (1975). "Efecto inhibidor de fármacos contra la obesidad en NADH deshidrogenasa de homogeneizados de corazón de ratón". Comunicaciones de Investigación en Patología Química y Farmacología . 11 (4): 645–6. PMID 241101 .
- ^ Muller, FL; Lustgarten, MS; Jang, Y .; Richardson, A. y Van Remmen, H. (2007). "Tendencias en las teorías del envejecimiento oxidativo". Radic libre. Biol. Med . 43 (4): 477–503. doi : 10.1016 / j.freeradbiomed.2007.03.034 . PMID 17640558 .
- ^ Skulachev VP (mayo de 1996). "Papel de las oxidaciones desacopladas y no acopladas en el mantenimiento de niveles bajos de oxígeno de forma segura y sus reductores de un electrón". Q. Rev. Biophys . 29 (2): 169–202. doi : 10.1017 / s0033583500005795 . PMID 8870073 .
- ↑ a b Muller F (2000). "La naturaleza y el mecanismo de producción de superóxido por la cadena de transporte de electrones: su relevancia para el envejecimiento" . EDAD . 23 (4): 227-253. doi : 10.1007 / s11357-000-0022-9 . PMC 3455268 . PMID 23604868 .
- ↑ a b Muller FL, Liu Y, Van Remmen H (noviembre de 2004). "El complejo III libera superóxido a ambos lados de la membrana mitocondrial interna" . J. Biol. Chem . 279 (47): 49064–73. doi : 10.1074 / jbc.M407715200 . PMID 15317809 .
- ^ Han D, Williams E, Cadenas E (enero de 2001). "Generación dependiente de la cadena respiratoria mitocondrial del anión superóxido y su liberación en el espacio intermembrana" . Biochem. J . 353 (Parte 2): 411–6. doi : 10.1042 / 0264-6021: 3530411 . PMC 1221585 . PMID 11139407 .
- ^ DiMauro S (noviembre de 2006). "Miopatías mitocondriales" (PDF) . Curr Opin Rheumatol . 18 (6): 636–41. doi : 10.1097 / 01.bor.0000245729.17759.f2 . PMID 17053512 . S2CID 29140366 .
- ^ DiMauro S (junio de 2007). "Medicina del ADN mitocondrial". Biosci. Rep . 27 (1-3): 5-9. doi : 10.1007 / s10540-007-9032-5 . PMID 17484047 . S2CID 5849380 .
- ^ Schuelke M, Krude H, Finckh B, Mayatepek E, Janssen A, Schmelz M, Trefz F, Trijbels F, Smeitink J (marzo de 2002). "Displasia septo-óptica asociada a una nueva mutación del citocromo b mitocondrial". Ana. Neurol . 51 (3): 388–92. doi : 10.1002 / ana.10151 . PMID 11891837 . S2CID 12425236 .
- ^ Wibrand F, Ravn K, Schwartz M, Rosenberg T, Horn N, Vissing J (octubre de 2001). "Trastorno multisistémico asociado con una mutación sin sentido en el gen del citocromo b mitocondrial". Ana. Neurol . 50 (4): 540–3. doi : 10.1002 / ana.1224 . PMID 11601507 . S2CID 8944744 .
- ^ Fisher N, Castleden CK, Bourges I, Brasseur G, Dujardin G, Meunier B (marzo de 2004). "Mutaciones relacionadas con enfermedades humanas en el citocromo b estudiadas en levadura" . J. Biol. Chem . 279 (13): 12951–8. doi : 10.1074 / jbc.M313866200 . PMID 14718526 .
Otras lecturas
- Marres CM, Slater EC (1977). "Composición de polipéptidos de QH2 purificado: citocromo c oxidorreductasa de mitocondrias de corazón de res". Biochim. Biophys. Acta . 462 (3): 531–548. doi : 10.1016 / 0005-2728 (77) 90099-8 . PMID 597492 .
- Rieske JS (1976). "Composición, estructura y función del complejo III de la cadena respiratoria". Biochim. Biophys. Acta . 456 (2): 195–247. doi : 10.1016 / 0304-4173 (76) 90012-4 . PMID 788795 .
- Wikstrom M, Krab K, Saraste M (1981). "Complejos de citocromo translocadores de protones". Annu. Rev. Biochem . 50 : 623–655. doi : 10.1146 / annurev.bi.50.070181.003203 . PMID 6267990 .
enlaces externos
- Sitio complejo del citocromo bc 1 (Edward A. Berry) en Wayback Machine (archivado el 9 de octubre de 2006) en lbl.gov
- sitio del complejo del citocromo bc 1 (Antony R. Crofts) en uiuc.edu
- PROMISE Base de datos: citocromo bc 1 complejo en archive.today (archivada 27 agosto de 1999) en scripps.edu
- Modelo molecular interactivo del Complejo III en Wayback Machine (archivado el 12 de enero de 2009) (Requiere MDL Chime )
- UMich Orientación de proteínas en familias de membranas / superfamilia-3 - Posiciones calculadas de bc1 y complejos relacionados en las membranas
- Coenzima + Q-citocromo-c + reductasa en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .
- EC 7.1.1
- Enzimas de estructura conocida
- Respiración celular
- Proteínas de hierro y azufre
- Proteínas transmembrana