Los cloroplastos tienen su propio ADN , [1] [2] a menudo abreviado como cpDNA . [3] También se conoce como plastoma cuando se hace referencia a los genomas de otros plastidios . Su existencia fue probada por primera vez en 1962. [4] Las primeras secuencias completas del genoma del cloroplasto fueron publicadas en 1986, Nicotiana tabacum (tabaco) por Sugiura y colegas y Marchantia polymorpha (hepática) por Ozeki et al. [5] [6] Desde entonces, se han secuenciado cientos de ADN de cloroplasto de varias especies , pero son [¿Cuándo? ]principalmente los delas plantas terrestresylas algas verdes: losglaucófitos,las algas rojasy otros grupos de algas están extremadamente subrepresentados, lo que podría introducir algúnsesgoen las opiniones sobre la estructura y el contenido del ADN del cloroplasto "típico". [7]
Estructura molecular [ editar ]
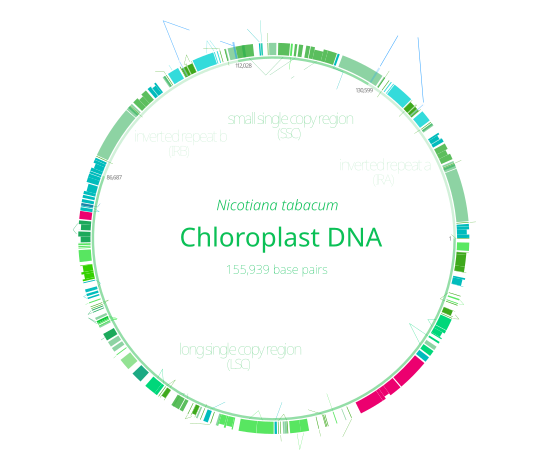
Los ADN de cloroplasto son circulares y, por lo general, tienen una longitud de 120 000 a 170 000 pares de bases . [4] [8] [9] Pueden tener una longitud de contorno de entre 30 y 60 micrómetros y una masa de entre 80 y 130 millones de dalton . [10]
La mayoría de los cloroplastos tienen todo su genoma de cloroplasto combinado en un solo anillo grande, aunque los de las algas dinofitas son una excepción notable: su genoma se divide en unos cuarenta plásmidos pequeños , cada uno de 2.000 a 10.000 pares de bases de largo. [7] Cada minicírculo contiene de uno a tres genes, [7] [11] pero también se han encontrado plásmidos en blanco, sin ADN codificante .
Repeticiones invertidas [ editar ]
Muchos ADN de cloroplasto contienen dos repeticiones invertidas , que separan una sección larga de copia única (LSC) de una sección corta de copia única (SSC). [9]
Las repeticiones invertidas varían enormemente en longitud, desde 4.000 hasta 25.000 pares de bases cada una. [7] Las repeticiones invertidas en las plantas tienden a estar en el extremo superior de este rango, cada una con una longitud de 20.000 a 25.000 pares de bases. [9] [12] Las regiones de repetición invertida generalmente contienen tres genes de ARN ribosómico y dos de ARNt , pero pueden expandirse o reducirse para contener tan solo cuatro o hasta más de 150 genes. [7] Si bien un par dado de repeticiones invertidas rara vez son completamente idénticas, siempre son muy similares entre sí, aparentemente como resultado de una evolución concertada . [7]
Las regiones de repetición invertida están muy conservadas entre las plantas terrestres y acumulan pocas mutaciones. [9] [12] Existen repeticiones invertidas similares en los genomas de las cianobacterias y los otros dos linajes de cloroplasto ( glaucophyta y rhodophyceæ ), lo que sugiere que son anteriores al cloroplasto, [7] aunque algunos ADN de cloroplasto como los de los guisantes y algunas algas rojas [7] desde entonces han perdido las repeticiones invertidas. [12] [13] Otros, como el alga roja Porphyra, voltearon una de sus repeticiones invertidas (haciéndolas repeticiones directas). [7]Es posible que las repeticiones invertidas ayuden a estabilizar el resto del genoma del cloroplasto, ya que los ADN del cloroplasto que han perdido algunos de los segmentos de repetición invertidos tienden a reorganizarse más. [13]
Estructura lineal [ editar ]
Se ha pensado durante mucho tiempo que el ADN del cloroplasto tiene una estructura circular, pero alguna evidencia sugiere que el ADN del cloroplasto toma más comúnmente una forma lineal. [14] Se ha observado que más del 95% del ADN del cloroplasto en los cloroplastos de maíz se encuentra en forma lineal ramificada en lugar de círculos individuales. [7]
Nucleoides [ editar ]
Cada cloroplasto contiene alrededor de 100 copias de su ADN en las hojas jóvenes, disminuyendo a 15-20 copias en las hojas más viejas. [15] Por lo general, se empaquetan en nucleoides que pueden contener varios anillos de ADN de cloroplasto idénticos. Se pueden encontrar muchos nucleoides en cada cloroplasto. [10]
Aunque el ADN del cloroplasto no está asociado con las histonas verdaderas , [16] en las algas rojas , se ha encontrado una proteína cloroplasto similar a histona (HC) codificada por el ADN del cloroplasto que empaqueta cada anillo de ADN del cloroplasto en un nucleoide . [17]
En las algas rojas primitivas , los nucleoides de ADN del cloroplasto se agrupan en el centro de un cloroplasto, mientras que en las plantas verdes y las algas verdes , los nucleoides se dispersan por todo el estroma . [17]
Replicación del ADN [ editar ]
Modelo líder de replicación de cpDNA [ editar ]
El mecanismo para la replicación del ADN del cloroplasto (ADNcp) no se ha determinado de manera concluyente, pero se han propuesto dos modelos principales. Los científicos han intentado observar la replicación del cloroplasto mediante microscopía electrónica desde la década de 1970. [18] [19] Los resultados de los experimentos de microscopía llevaron a la idea de que el ADN del cloroplasto se replica utilizando un bucle de doble desplazamiento (bucle D). A medida que el bucle D se mueve a través del ADN circular, adopta una forma intermedia theta, también conocida como un intermedio de replicación de Cairns, y completa la replicación con un mecanismo de círculo rodante. [18] [20] La replicación comienza en puntos de origen específicos. Varias bifurcaciones de replicaciónse abren, permitiendo que la maquinaria de replicación replique el ADN. A medida que continúa la replicación, las bifurcaciones crecen y finalmente convergen. Las nuevas estructuras de cpDNA se separan, creando cromosomas de cpDNA hijos.
Además de los primeros experimentos de microscopía, este modelo también está respaldado por las cantidades de desaminación observadas en el cpDNA. [18] La desaminación ocurre cuando se pierde un grupo amino y es una mutación que a menudo resulta en cambios de base. Cuando se desamina la adenina, se convierte en hipoxantina . La hipoxantina puede unirse a la citosina , y cuando el par de bases XC se replica, se convierte en un GC (por lo tanto, un cambio de base A → G). [21]
En el cpDNA, hay varios gradientes de desaminación A → G. El ADN se vuelve susceptible a eventos de desaminación cuando es monocatenario. Cuando se forman las bifurcaciones de replicación, la hebra que no se copia es monocatenaria y, por tanto, corre el riesgo de desaminación A → G. Por lo tanto, los gradientes en la desaminación indican que lo más probable es que las horquillas de replicación estuvieran presentes y la dirección en la que se abrieron inicialmente (el gradiente más alto probablemente sea el más cercano al sitio de inicio porque fue monocatenario durante la mayor cantidad de tiempo). [18]Este mecanismo sigue siendo la teoría principal en la actualidad; sin embargo, una segunda teoría sugiere que la mayor parte del cpDNA es realmente lineal y se replica mediante recombinación homóloga. Además, sostiene que solo una minoría del material genético se mantiene en cromosomas circulares, mientras que el resto está en estructuras ramificadas, lineales u otras estructuras complejas. [18] [20]
Modelo alternativo de replicación [ editar ]
Uno de los principales modelos que compiten con el cpDNA afirma que la mayoría del cpDNA es lineal y participa en estructuras de recombinación y replicación homólogas similares al bacteriófago T4 . [20] Se ha establecido que algunas plantas tienen ADNcp lineal, como el maíz, y que muchas más contienen estructuras complejas que los científicos aún no comprenden; [20] sin embargo, la opinión predominante en la actualidad es que la mayoría de cpDNA es circular. Cuando se realizaron los experimentos originales sobre cpDNA, los científicos notaron estructuras lineales; sin embargo, atribuyeron estas formas lineales a círculos rotos. [20]Si las estructuras ramificadas y complejas observadas en los experimentos de cpDNA son reales y no artefactos de ADN circular concatenado o círculos rotos, entonces un mecanismo de replicación de bucle D es insuficiente para explicar cómo se replicarían esas estructuras. [20] Al mismo tiempo, la recombinación homóloga no explica los múltiples gradientes A → G observados en los plastomas. [18] Esta deficiencia es una de las más importantes de la teoría de la estructura lineal.
Contenido genético y síntesis de proteínas [ editar ]
El genoma del cloroplasto incluye más comúnmente alrededor de 100 genes [8] [11] que codifican una variedad de cosas, principalmente relacionadas con la cadena de proteínas y la fotosíntesis . Al igual que en los procariotas , los genes del ADN del cloroplasto están organizados en operones . [11] Los intrones son comunes en las moléculas de ADN del cloroplasto, mientras que son raros en las moléculas de ADN procariotas ( los ADN mitocondriales de las plantas suelen tener intrones, pero no ADNmt humano). [22]
Entre las plantas terrestres, el contenido del genoma del cloroplasto es bastante similar [9]: codifican cuatro ARN ribosomales , 30-31 ARNt , 21 proteínas ribosómicas y 4 subunidades de ARN polimerasa , [23] [24] involucradas en la síntesis de proteínas. Para la fotosíntesis, el ADN del cloroplasto incluye genes para 28 proteínas tilacoides y la gran subunidad de Rubisco . [23] Además, sus genes codifican once subunidades de un complejo proteico que media las reacciones redox para reciclar electrones, [25] que es similar a la NADH deshidrogenasa.que se encuentra en las mitocondrias. [23] [26]
Reducción del genoma del cloroplasto y transferencia de genes [ editar ]
Con el tiempo, muchas partes del genoma del cloroplasto se transfirieron al genoma nuclear del huésped, [4] [8] [27] un proceso llamado transferencia génica endosimbiótica . Como resultado, el genoma del cloroplasto está muy reducido en comparación con el de las cianobacterias de vida libre. Los cloroplastos pueden contener de 60 a 100 genes, mientras que las cianobacterias suelen tener más de 1500 genes en su genoma. [28] Por el contrario, solo hay unos pocos casos conocidos en los que se han transferido genes al cloroplasto de varios donantes, incluidas bacterias. [29] [30] [31]
La transferencia de genes endosimbióticos es la forma en que conocemos los cloroplastos perdidos en muchos linajes de cromalveolados . Incluso si un cloroplasto finalmente se pierde, los genes que donó al núcleo del antiguo huésped persisten, lo que proporciona evidencia de la existencia del cloroplasto perdido. Por ejemplo, mientras que las diatomeas (una heterokontophyte ) tienen ahora un alga roja cloroplasto derivado , la presencia de muchas algas verdes genes en el núcleo de diatomeas proporcionar evidencia de que el ancestro de diatomeas (probablemente el antepasado de todos los cromalveolados también) tenía un alga verde deriva del cloroplasto en algún punto, que posteriormente fue reemplazado por el cloroplasto rojo. [32]
En las plantas terrestres, entre el 11 y el 14% del ADN de sus núcleos se remonta al cloroplasto, [33] hasta el 18% en Arabidopsis , lo que corresponde a unos 4.500 genes que codifican proteínas. [34] Ha habido algunas transferencias recientes de genes del ADN del cloroplasto al genoma nuclear en plantas terrestres. [8]
Proteínas codificadas por el cloroplasto [ editar ]
De las aproximadamente tres mil proteínas que se encuentran en los cloroplastos, alrededor del 95% están codificadas por genes nucleares. Muchos de los complejos de proteínas del cloroplasto consisten en subunidades tanto del genoma del cloroplasto como del genoma nuclear del huésped. Como resultado, la síntesis de proteínas debe coordinarse entre el cloroplasto y el núcleo. El cloroplasto está principalmente bajo control nuclear, aunque los cloroplastos también pueden emitir señales que regulan la expresión génica en el núcleo, lo que se denomina señalización retrógrada . [35]
Síntesis de proteínas [ editar ]
La síntesis de proteínas dentro de los cloroplastos se basa en una ARN polimerasa codificada por el propio genoma del cloroplasto, que está relacionada con las ARN polimerasas que se encuentran en las bacterias. Los cloroplastos también contienen una misteriosa segunda ARN polimerasa codificada por el genoma nuclear de la planta. Las dos ARN polimerasas pueden reconocer y unirse a diferentes tipos de promotores dentro del genoma del cloroplasto. [36] Los ribosomas en los cloroplastos son similares a los ribosomas bacterianos. [23]
Esta sección necesita expansión con: diferencias de tamaño de genoma entre algas y plantas terrestres, material de cloroplasto codificado por el núcleo, replicación de ADN, NADPH redox, tRNA sintetasas especiales, etc. Puede ayudar añadiéndole . ( Enero de 2013 ) |
Edición de ARN en plastidios [ editar ]
La edición de ARN es la inserción, deleción y sustitución de nucleótidos en una transcripción de ARNm antes de la traducción a proteína. El entorno altamente oxidativo dentro de los cloroplastos aumenta la tasa de mutación, por lo que se necesitan reparaciones posteriores a la transcripción para conservar las secuencias funcionales. El editosoma del cloroplasto sustituye a C -> U y U -> C en lugares muy específicos de la transcripción. Esto puede cambiar el codón de un aminoácido o restaurar un pseudogén no funcional agregando un codón de inicio AUG o eliminando un codón de terminación UAA prematuro. [37]
El editosoma reconoce y se une a la secuencia cis corriente arriba del sitio de edición. La distancia entre el sitio de unión y el sitio de edición varía según el gen y las proteínas involucradas en el editosoma. Cientos de diferentes proteínas PPR del genoma nuclear están involucradas en el proceso de edición de ARN. Estas proteínas constan de 35 aminoácidos repetidos, cuya secuencia determina el sitio de unión cis para la transcripción editada. [37]
Las plantas terrestres basales como hepáticas, musgos y helechos tienen cientos de sitios de edición diferentes, mientras que las plantas con flores suelen tener entre treinta y cuarenta. Las plantas parasitarias como Epifagus virginiana muestran una pérdida de edición de ARN que resulta en una pérdida de función para los genes de fotosíntesis. [38]
Importación y selección de proteínas [ editar ]
El movimiento de tantos genes del cloroplasto hacia el núcleo significa que muchas proteínas del cloroplasto que se suponía que debían traducirse en el cloroplasto ahora se sintetizan en el citoplasma. Esto significa que estas proteínas deben dirigirse de regreso al cloroplasto e importarse a través de al menos dos membranas de cloroplasto. [39]
Curiosamente, alrededor de la mitad de los productos proteicos de los genes transferidos ni siquiera se dirigen al cloroplasto. Muchos se convirtieron en exaptaciones , asumiendo nuevas funciones como participar en la división celular , el enrutamiento de proteínas e incluso la resistencia a enfermedades . Algunos genes de cloroplasto encontraron nuevos hogares en el genoma mitocondrial; la mayoría se convirtió en pseudogenes no funcionales , aunque algunos genes de ARNt todavía funcionan en la mitocondria . [28] Algunos productos de proteínas de ADN de cloroplasto transferidos se dirigen a la vía secretora [28] (aunque muchos plástidos secundariosestán delimitados por una membrana más externa derivada de la membrana celular del huésped y, por lo tanto, topológicamente fuera de la célula, porque para llegar al cloroplasto desde el citosol , debe atravesar la membrana celular , como si se dirigiera al espacio extracelular . En esos casos, las proteínas dirigidas al cloroplasto viajan inicialmente a lo largo de la vía secretora). [40]
Debido a que la célula que adquiere un cloroplasto ya tenía mitocondrias (y peroxisomas , y una membrana celular para la secreción), el nuevo huésped del cloroplasto tuvo que desarrollar un sistema único de selección de proteínas para evitar que las proteínas del cloroplasto se enviaran al orgánulo incorrecto . [39]
Secuencias de traducción citoplásmica y tránsito N-terminal [ editar ]
Los polipéptidos , los precursores de las proteínas , son cadenas de aminoácidos . Los dos extremos de un polipéptido se denominan N-terminal , o extremo amino , y C-terminal , o extremo carboxilo . [41] Para muchas (pero no todas) [42] proteínas de cloroplasto codificadas por genes nucleares , se agregan péptidos de tránsito escindibles a los extremos N-terminales de los polipéptidos, que se utilizan para ayudar a dirigir el polipéptido al cloroplasto para su importación [39] [43] (Los péptidos de tránsito N-terminal también se utilizan para dirigir polipéptidos a las mitocondrias de las plantas ).[44] Las secuencias de tránsito N-terminal también se denominan presecuencias [39] porque están ubicadas en el extremo "frontal" de un polipéptido; los ribosomas sintetizan polipéptidos desde el extremo N al extremo C. [41]
Los péptidos de tránsito de cloroplasto exhiben una gran variación en longitud y secuencia de aminoácidos . [43] Pueden tener entre 20 y 150 aminoácidos de longitud [39], una longitud inusualmente larga, lo que sugiere que los péptidos de tránsito son en realidad colecciones de dominios con diferentes funciones. [43] péptidos de tránsito tienden a ser cargado positivamente , [39] rica en hidroxilado amino ácidos tales como serina , treonina y prolina , y pobre en ácidos aminoácidos como ácido aspártico y ácido glutámico . [43]En una solución acuosa , la secuencia de tránsito forma una espiral aleatoria. [39]
Sin embargo, no todas las proteínas del cloroplasto incluyen un péptido de tránsito escindible N-terminal. [39] Algunos incluyen la secuencia de tránsito dentro de la parte funcional de la proteína misma. [39] En cambio, algunos tienen su secuencia de tránsito adjunta a su extremo C-terminal . [45] La mayoría de los polipéptidos que carecen de secuencias de dirección N-terminales son los que se envían a la membrana externa del cloroplasto , más al menos uno enviado a la membrana interna del cloroplasto . [39]
Fosforilación, acompañantes y transporte [ editar ]
Después de que se sintetiza un polipéptido de cloroplasto en un ribosoma en el citosol , la energía de ATP puede usarse para fosforilar o agregar un grupo fosfato a muchos (pero no a todos) de ellos en sus secuencias de tránsito. [39] La serina y la treonina (ambas muy comunes en las secuencias de tránsito del cloroplasto, que constituyen el 20-30% de la secuencia) [46] son a menudo los aminoácidos que aceptan el grupo fosfato . [44] [46] La enzima que lleva a cabo la fosforilación es específicapara polipéptidos de cloroplasto e ignora los destinados a mitocondrias o peroxisomas . [46]
La fosforilación cambia la forma del polipéptido, [46] facilitando que las proteínas 14-3-3 se unan al polipéptido. [39] [47] En las plantas, las proteínas 14-3-3 solo se unen a las preproteínas del cloroplasto. [44] También está obligado por la h comer s hock p rotein Hsp70 que mantiene el polipéptido a partir de plegado antes de tiempo. [39] Esto es importante porque evita que las proteínas del cloroplasto asuman su forma activa y lleven a cabo sus funciones de cloroplasto en el lugar equivocado: el citosol . [44] [47]Al mismo tiempo, deben mantener la forma suficiente para que puedan ser reconocidos e importados al cloroplasto. [44]
La proteína de choque térmico y las proteínas 14-3-3 juntas forman un complejo de guía citosólico que facilita la importación del polipéptido del cloroplasto al cloroplasto. [39]
Alternativamente, si el péptido de tránsito de una preproteína de cloroplasto no está fosforilado, una preproteína de cloroplasto todavía puede unirse a una proteína de choque térmico o Toc159 . Estos complejos pueden unirse al complejo TOC en la membrana exterior del cloroplasto utilizando energía GTP . [39]
El translocón en la membrana exterior del cloroplasto (TOC) [ editar ]
El complejo TOC , o t ranslocon en la o uter c hloroplast membrana , es una colección de proteínas que las importaciones preproteínas a través de la envoltura del cloroplasto exterior . Se han identificado cinco subunidades del complejo TOC: dos proteínas de unión a GTP , Toc34 y Toc159 , el túnel de importación de proteínas Toc75 , más las proteínas Toc64 [39] y Toc12 . [42]
Las primeras tres proteínas forman un complejo central que consta de un Toc159, cuatro a cinco Toc34 y cuatro Toc75 que forman cuatro agujeros en un disco de 13 nanómetros de diámetro. Todo el complejo central pesa alrededor de 500 kilodaltons . Las otras dos proteínas, Toc64 y Toc12, están asociadas con el complejo central, pero no forman parte de él. [42]
Toc34 y 33 [ editar ]
Toc34 es una proteína integral en la membrana exterior del cloroplasto que está anclada a ella por su cola hidrófoba [49] C-terminal . [39] [47] Sin embargo, la mayor parte de la proteína, incluido su gran dominio de unión al trifosfato de guanosina (GTP) , se proyecta hacia el estroma. [47]
El trabajo de Toc34 es atrapar algunas preproteínas de cloroplasto en el citosol y entregarlas al resto del complejo TOC. [39] Cuando GTP , una molécula de energía similar al ATP se une a Toc34, la proteína se vuelve mucho más capaz de unirse a muchas preproteínas de cloroplasto en el citosol . [39] La presencia de la preproteína del cloroplasto hace que Toc34 rompa el GTP en difosfato de guanosina (GDP) y fosfato inorgánico . Esta pérdida de GTP hace que la proteína Toc34 libere la preproteína del cloroplasto, entregándola a la siguiente proteína TOC. [39]Toc34 luego libera la molécula del PIB agotada, probablemente con la ayuda de un factor de intercambio del PIB desconocido . Un dominio de Toc159 podría ser el factor de cambio que lleve a cabo la remoción del PIB. La proteína Toc34 puede tomar otra molécula de GTP y comenzar el ciclo nuevamente. [39]
Toc34 se puede apagar mediante fosforilación . Una proteína quinasa que se desplaza por la membrana externa del cloroplasto puede usar ATP para agregar un grupo fosfato a la proteína Toc34, evitando que pueda recibir otra molécula de GTP , inhibiendo la actividad de la proteína. Esto podría proporcionar una forma de regular la importación de proteínas a los cloroplastos. [39] [47]
Arabidopsis thaliana tiene dosproteínas homólogas , AtToc33 y AtToc34 (La At significa A rabidopsis t haliana ), [39] [47] que son aproximadamente un 60% idénticas en secuencia de aminoácidos a Toc34 en guisantes (llamado ps Toc34). [47] AtToc33 es el más común en Arabidopsis , [47] y es el análogo funcionalde Toc34 porque puede desactivarse por fosforilación. AtToc34, por otro lado, no se puede fosforilar. [39] [47]
Toc159 [ editar ]
Toc159 es otra subunidad de TOC de unión a GTP , como Toc34 . Toc159 tiene tres dominios . En el extremo N-terminal está el dominio A, que es rico en aminoácidos ácidos y ocupa aproximadamente la mitad de la longitud de la proteína. [39] [49] El dominio A a menudo se escinde , dejando un fragmento de 86 kilodalton llamado Toc86 . [49] En el medio está su dominio de unión a GTP , que es muy similar al dominio de unión a GTP homólogo en Toc34. [39] [49] En la terminal CEl extremo es el dominio M hidrófilo , [39] que ancla la proteína a la membrana exterior del cloroplasto. [49]
Toc159 probablemente funciona de manera muy similar a Toc34, reconociendo proteínas en el citosol usando GTP . Puede ser regulada por fosforilación , pero por una proteína quinasa diferente a la que fosforila Toc34. [42] Su dominio M forma parte del túnel por el que viajan las preproteínas del cloroplasto, y parece proporcionar la fuerza que empuja a las preproteínas, utilizando la energía de GTP . [39]
Toc159 no siempre se encuentra como parte del complejo TOC; también se ha encontrado disuelto en el citosol . Esto sugiere que podría actuar como un transbordador que encuentra preproteínas de cloroplasto en el citosol y las lleva de regreso al complejo TOC. Sin embargo, no hay mucha evidencia directa de este comportamiento. [39]
Se ha encontrado una familia de proteínas Toc159, Toc159 , Toc132 , Toc120 y Toc90 en Arabidopsis thaliana . Varían en la longitud de sus dominios A, que desapareció por completo en Toc90. Toc132, Toc120 y Toc90 parecen tener funciones especializadas en la importación de cosas como preproteínas no fotosintéticas y no pueden reemplazar a Toc159. [39]
Toc75 [ editar ]
Toc75 es la proteína más abundante en la envoltura exterior del cloroplasto. Es un tubo transmembrana que forma la mayor parte del poro de TOC. Toc75 es un canal de barril β revestido por 16 láminas plisadas β . [39] El agujero que forma tiene unos 2,5 nanómetros de ancho en los extremos y se contrae a unos 1,4-1,6 nanómetros de diámetro en su punto más estrecho, lo suficientemente ancho para permitir el paso de las preproteínas de cloroplasto parcialmente plegadas. [39]
Toc75 también puede unirse a preproteínas de cloroplasto, pero es mucho peor en esto que Toc34 o Toc159. [39]
Arabidopsis thaliana tiene múltiples isoformas de Toc75 que son nombradas por lasposiciones cromosómicas de los genes que las codifican. AtToc75 III es el más abundante de estos. [39]
El translocón en la membrana interna del cloroplasto (TIC) [ editar ]
El translocon TIC , o t ranslocon en el i nner c hloroplast membrana translocon [39] es otro complejo de proteína que importa proteínas a través de la envoltura del cloroplasto interior . Las cadenas polipeptídicas de cloroplastos probablemente viajan a menudo a través de los dos complejos al mismo tiempo, pero el complejo TIC también puede recuperar preproteínas perdidas en el espacio intermembrana . [39]
Al igual que el translocón de TOC , el translocón de TIC tiene un gran complejo central rodeado por algunas proteínas periféricas poco asociadas como Tic110 , Tic40 y Tic21 . [50] El complejo central pesa alrededor de un millón de daltons y contiene Tic214 , Tic100 , Tic56 y Tic20 I , posiblemente tres de cada uno. [50]
Tic20 [ editar ]
Tic20 es una proteína integral que se cree que tiene cuatro hélices α transmembrana . [39] Se encuentra en el complejo TIC de 1 millón de dalton . [50] Debido a que es similar a bacterianas de aminoácidos transportistas y el mitocondrial proteína importación Tim17 [39] ( t ranslocase en el i nner m itochondrial m embrane ), [51] se ha propuesto para ser parte del canal de importación TIC. [39] No hay in vitroevidencia de esto sin embargo. [39] En Arabidopsis thaliana , se sabe que por cada cinco proteínas Toc75 en la membrana externa del cloroplasto, hay dos proteínas Tic20 I (la forma principal de Tic20 en Arabidopsis ) en la membrana interna del cloroplasto. [50]
A diferencia de Tic214 , Tic100 o Tic56 , Tic20 tiene parientes homólogos en cianobacterias y casi todos los linajes de cloroplasto, lo que sugiere que evolucionó antes de la primera endosimbiosis de cloroplasto. Tic214 , Tic100 y Tic56 son exclusivos de los cloroplastos de cloroplastidan, lo que sugiere que evolucionaron más tarde. [50]
Tic214 [ editar ]
Tic214 es otra proteína del complejo central de TIC, llamada así porque pesa poco menos de 214 kilodaltons . Tiene 1786 aminoácidos de longitud y se cree que tiene seis dominios transmembrana en su extremo N-terminal . Tic214 se destaca por estar codificado por el ADN del cloroplasto, más específicamente el primer marco de lectura abierto ycf1 . Tic214 y Tic20 juntos probablemente forman parte del complejo TIC de un millón de dalton que se extiende por toda la membrana . Tic20 está enterrado dentro del complejo, mientras que Tic214 está expuesto en ambos lados de la membrana interna de cloroplasto . [50]
Tic100 [ editar ]
Tic100 es una proteína codificada nuclear que tiene 871 aminoácidos de longitud. Los 871 aminoácidos pesan colectivamente un poco menos de 100 mil daltons , y dado que la proteína madura probablemente no pierde ningún aminoácido cuando se importa al cloroplasto (no tiene péptido de tránsito escindible ), se llamó Tic100. Tic100 se encuentra en los bordes del complejo de 1 millón de dalton en el lado que mira hacia el espacio intermembrana del cloroplasto . [50]
Tic56 [ editar ]
Tic56 también es una proteína codificada nuclear . La preproteína que codifica su gen tiene 527 aminoácidos de longitud, con un peso cercano a los 62 mil daltons ; la forma madura probablemente se somete a un procesamiento que la reduce a algo que pesa 56 mil daltons cuando se importa al cloroplasto. Tic56 está incrustado en gran parte dentro del complejo de 1 millón de dalton. [50]
Tic56 y Tic100 están altamente conservados entre las plantas terrestres, pero no se parecen a ninguna proteína cuya función sea conocida. Tampoco tiene dominios transmembrana . [50]
Ver también [ editar ]
- Lista de plastomas secuenciados
- ADN mitocondrial
Referencias [ editar ]
- ^ de Vries J, Archibald JM (abril de 2018). "Genomas de plastidios" . Biología actual . 28 (8): R336 – R337. doi : 10.1016 / j.cub.2018.01.027 . PMID 29689202 . S2CID 207053862 .
- ^ C.Michael Hogan. 2010. Ácido desoxirribonucleico . Enciclopedia de la Tierra. Consejo Nacional de Ciencia y Medio Ambiente. eds. S.Draggan y C.Cleveland. Washington DC
- ^ Sakamoto W, Takami T (junio de 2018). "Dinámica del ADN del cloroplasto: número de copias, control de calidad y degradación" . Fisiología vegetal y celular . 59 (6): 1120–1127. doi : 10.1093 / pcp / pcy084 . PMID 29860378 .
- ↑ a b c Dann L (2002). Biociencia — Explicación (PDF) . ADN verde: BIOSCIENCIA EXPLICADA.
- ^ Shinozaki, K .; Oh, M .; Tanaka, M .; Wakasugi, T .; Hayashida, N .; Matsubayashi, T .; Zaita, N .; Chunwongse, J .; Obokata, J .; Yamaguchi-Shinozaki, K .; Ohto, C. (1986). "La secuencia completa de nucleótidos del genoma del cloroplasto del tabaco: su organización y expresión génica" . El diario EMBO . 5 (9): 2043-2049. doi : 10.1002 / j.1460-2075.1986.tb04464.x . ISSN 0261-4189 . PMC 1167080 . PMID 16453699 .
- ^ Ohyama, Kanji; Fukuzawa, Hideya; Kohchi, Takayuki; Shirai, Hiromasa; Sano, Tohru; Sano, Satoshi; Umesono, Kazuhiko; Shiki, Yasuhiko; Takeuchi, Masayuki; Chang, Zhen; Aota, Shin-ichi (1986). "Organización del gen del cloroplasto deducida de la secuencia completa del ADN del cloroplasto de la hepática Marchantia polymorpha" . Naturaleza . 322 (6079): 572–574. Código Bibliográfico : 1986Natur.322..572O . doi : 10.1038 / 322572a0 . ISSN 1476-4687 . S2CID 4311952 .
- ↑ a b c d e f g h i j Sandelius AS (2009). El cloroplasto: interacciones con el medio ambiente . Saltador. pag. 18. ISBN 978-3-540-68696-5.
- ^ a b c d Clegg MT, Gaut BS, Learn GH, Morton BR (julio de 1994). "Tasas y patrones de evolución del ADN del cloroplasto" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (15): 6795–801. Código Bibliográfico : 1994PNAS ... 91.6795C . doi : 10.1073 / pnas.91.15.6795 . PMC 44285 . PMID 8041699 .
- ↑ a b c d e Shaw J, Lickey EB, Schilling EE, Small RL (marzo de 2007). "Comparación de secuencias del genoma de cloroplasto completo para elegir regiones no codificantes para estudios filogenéticos en angiospermas: la tortuga y la liebre III". Revista estadounidense de botánica . 94 (3): 275–88. doi : 10.3732 / ajb.94.3.275 . PMID 21636401 .
- ↑ a b Burgess J (1989). Una introducción al desarrollo de las células vegetales . Cambridge: prensa de la universidad de Cambridge. pag. 62. ISBN 978-0-521-31611-8.
- ↑ a b c McFadden GI (enero de 2001). "Origen e integración del cloroplasto" . Fisiología vegetal . 125 (1): 50–3. doi : 10.1104 / pp.125.1.50 . PMC 1539323 . PMID 11154294 .
- ↑ a b c Kolodner R, Tewari KK (enero de 1979). "Repeticiones invertidas en el ADN del cloroplasto de plantas superiores" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 76 (1): 41–5. Código Bibliográfico : 1979PNAS ... 76 ... 41K . doi : 10.1073 / pnas.76.1.41 . PMC 382872 . PMID 16592612 .
- ↑ a b Palmer JD, Thompson WF (junio de 1982). "Los reordenamientos del ADN del cloroplasto son más frecuentes cuando se pierde una gran secuencia repetida invertida". Celular . 29 (2): 537–50. doi : 10.1016 / 0092-8674 (82) 90170-2 . PMID 6288261 . S2CID 11571695 .
- ^ Bendich AJ (julio de 2004). "Cromosomas circulares del cloroplasto: la gran ilusión" . La célula vegetal . 16 (7): 1661–6. doi : 10.1105 / tpc.160771 . PMC 514151 . PMID 15235123 .
- ^ Plant Biochemistry (3ª ed.). Prensa académica. 2005. p. 517 . ISBN 9780120883912.
número de copias de ctDNA por cloroplasto.
- ^ Biología octava edición Campbell & Reece . Benjamin Cummings (Pearson). 2009. p. 516.
- ↑ a b Kobayashi T, Takahara M, Miyagishima SY, Kuroiwa H, Sasaki N, Ohta N, Matsuzaki M, Kuroiwa T (julio de 2002). "Detección y localización de una proteína similar a HU codificada por cloroplasto que organiza nucleoides de cloroplasto" . La célula vegetal . 14 (7): 1579–89. doi : 10.1105 / tpc.002717 . PMC 150708 . PMID 12119376 .
- ↑ a b c d e f Krishnan NM, Rao BJ (mayo de 2009). "Un enfoque comparativo para dilucidar la replicación del genoma del cloroplasto" . BMC Genomics . 10 (237): 237. doi : 10.1186 / 1471-2164-10-237 . PMC 2695485 . PMID 19457260 .
- ^ Heinhorst, Gordon C. Cannon, Sabine (1993). "Replicación de ADN en cloroplastos". Revista de ciencia celular . 104 : 1-9.
- ↑ a b c d e f Bendich AJ (julio de 2004). "Cromosomas circulares del cloroplasto: la gran ilusión" . La célula vegetal . 16 (7): 1661–6. doi : 10.1105 / tpc.160771 . PMC 514151 . PMID 15235123 .
- ^ "Efecto de mutágenos químicos sobre la secuencia de nucleótidos" . Biociclopedia . Consultado el 24 de octubre de 2015 .
- ^ Alberts B (2002). Biología molecular de la célula (4. ed.). Nueva York [ua]: Garland. ISBN 978-0-8153-4072-0.
- ↑ a b c d Harris EH, Boynton JE, Gillham NW (diciembre de 1994). "Ribosomas de cloroplasto y síntesis de proteínas" . Revisiones microbiológicas . 58 (4): 700–54. doi : 10.1128 / MMBR.58.4.700-754.1994 . PMC 372988 . PMID 7854253 .
- ^ Wakasugi T, Sugita M, Tsudzuki T, Sugiura M (1998). "Mapa de genes actualizado del ADN del cloroplasto del tabaco". Reportero de Biología Molecular Vegetal . 16 (3): 231–41. doi : 10.1023 / A: 1007564209282 . S2CID 40036883 .
- ^ Krause K (septiembre de 2008). "De los cloroplastos a los plástidos" crípticos ": evolución de los genomas de los plástidos en plantas parásitas". Genética actual . 54 (3): 111–21. doi : 10.1007 / s00294-008-0208-8 . PMID 18696071 . S2CID 24879257 .
- ^ Peng L, Fukao Y, Fujiwara M, Shikanai T (enero de 2012). "El ensamblaje de múltiples pasos del subcomplejo A similar a la deshidrogenasa NADH del cloroplasto requiere varias proteínas codificadas por el núcleo, incluidas CRR41 y CRR42, en Arabidopsis" . La célula vegetal . 24 (1): 202-14. doi : 10.1105 / tpc.111.090597 . PMC 3289569 . PMID 22274627 .
- ^ Huang CY, Ayliffe MA, Timmis JN (marzo de 2003). "Medición directa de la tasa de transferencia del ADN del cloroplasto al núcleo". Naturaleza . 422 (6927): 72–6. Código Bib : 2003Natur.422 ... 72H . doi : 10.1038 / nature01435 . PMID 12594458 . S2CID 4319507 .
- ^ a b c Martin W, Rujan T, Richly E, Hansen A, Cornelsen S, Lins T, Leister D, Stoebe B, Hasegawa M, Penny D (septiembre de 2002). "El análisis evolutivo de genomas de Arabidopsis, cianobacterias y cloroplasto revela filogenia de plastidios y miles de genes de cianobacterias en el núcleo" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (19): 12246–51. Código Bibliográfico : 2002PNAS ... 9912246M . doi : 10.1073 / pnas.182432999 . PMC 129430 . PMID 12218172 .
- ^ Mackiewicz P, Bodył A, Moszczyński K (julio de 2013). "El caso de la transferencia horizontal de genes de bacterias al peculiar genoma del plastidio dinoflagelado" . Elementos genéticos móviles . 3 (4): e25845. doi : 10.4161 / mge.25845 . PMC 3812789 . PMID 24195014 .
- ^ Leliaert F, Lopez-Bautista JM (marzo de 2015). "Los genomas de cloroplasto de Bryopsis plumosa y Tydemania expeditiones (Bryopsidales, Chlorophyta): genomas compactos y genes de origen bacteriano" . BMC Genomics . 16 (1): 204. doi : 10.1186 / s12864-015-1418-3 . PMC 4487195 . PMID 25879186 .
- ^ Robison, TA, Grusz AL, Wolf PG, Mower, JP, Fauskee BD, Sosa K y Schuettpelz E (octubre de 2018). "Elementos móviles dan forma a la evolución del plastomo en helechos" . Biología y evolución del genoma . 10 (10): 2669-2571. doi : 10.1093 / gbe / evy189 . PMC 6166771 . PMID 30165616 .
- ^ Moustafa A, Beszteri B, Maier UG, Bowler C, Valentin K, Bhattacharya D (junio de 2009). "Huellas genómicas de una endosimbiosis plastídica críptica en diatomeas" (PDF) . Ciencia . 324 (5935): 1724–6. Código Bibliográfico : 2009Sci ... 324.1724M . doi : 10.1126 / science.1172983 . PMID 19556510 . S2CID 11408339 .
- ^ Nowack EC, Vogel H, Groth M, Grossman AR, Melkonian M, Glöckner G (enero de 2011). "Transferencia de genes endosimbióticos y regulación transcripcional de genes transferidos en Paulinella chromatophora" . Biología Molecular y Evolución . 28 (1): 407–22. doi : 10.1093 / molbev / msq209 . PMID 20702568 .
- ^ Archibald JM (diciembre de 2006). "Genómica de las algas: explorar la huella de la endosimbiosis" . Biología actual . 16 (24): R1033-5. doi : 10.1016 / j.cub.2006.11.008 . PMID 17174910 . S2CID 17830745 .
- ^ Koussevitzky S, Nott A, Mockler TC, Hong F, Sachetto-Martins G, Surpin M, Lim J, Mittler R, Chory J (mayo de 2007). "Las señales de los cloroplastos convergen para regular la expresión de genes nucleares" . Ciencia . 316 (5825): 715–9. Código Bibliográfico : 2007Sci ... 316..715K . doi : 10.1126 / science.1140516 (inactivo 2021-01-14). PMID 17395793 . Mantenimiento de CS1: DOI inactivo a partir de enero de 2021 ( enlace )
- ^ Hedtke B, Börner T, Weihe A (agosto de 1997). "Polimerasas de ARN de tipo fago mitocondrial y cloroplasto en Arabidopsis". Ciencia . 277 (5327): 809-11. doi : 10.1126 / science.277.5327.809 . PMID 9242608 .
- ↑ a b Takenaka M, Zehrmann A, Verbitskiy D, Härtel B, Brennicke A (2013). "Edición de ARN en plantas y su evolución". Revisión anual de genética . 47 (1): 335–52. doi : 10.1146 / annurev-genet-111212-133519 . PMID 24274753 .
- ^ Tillich M, Krause K (julio de 2010). "Los entresijos de la edición y empalme de ARN plastid: lecciones de plantas parásitas". Nueva biotecnología . Número especial: Revisión anual de biotecnología 2010 Conceptos básicos de ARN y aplicaciones de biotecnología. 27 (3): 256–66. doi : 10.1016 / j.nbt.2010.02.020 . PMID 20206308 .
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak al am an Soll J, Schleiff E (marzo de 2004) . "Importación de proteínas en cloroplastos". Reseñas de la naturaleza. Biología celular molecular . 5 (3): 198-208. doi : 10.1038 / nrm1333 .PMID 14991000 . S2CID 32453554 .
- ^ Keeling PJ (marzo de 2010). "El origen endosimbiótico, diversificación y destino de los plastidios" . Transacciones filosóficas de la Royal Society de Londres. Serie B, Ciencias Biológicas . 365 (1541): 729–48. doi : 10.1098 / rstb.2009.0103 . PMC 2817223 . PMID 20124341 .
- ^ a b Biología 8ª edición: Campbell & Reece . Benjamin Cummings. 2008. p. 340. ISBN 978-0-321-54325-7.
- ↑ a b c d Wise RR, Hoober JK (2007). Estructura y función de plastidios . Berlín: Springer. págs. 53–74. ISBN 978-1-4020-6570-5.
- ^ a b c d Lee DW, Lee S, Lee GJ, Lee KH, Kim S, Cheong GW, Hwang I (febrero de 2006). "Caracterización funcional de motivos de secuencia en el péptido de tránsito de subunidad pequeña de Arabidopsis de rubisco" . Fisiología vegetal . 140 (2): 466–83. doi : 10.1104 / pp.105.074575 . PMC 1361317 . PMID 16384899 .
- ↑ a b c d e May T, Soll J (enero de 2000). "Las proteínas 14-3-3 forman un complejo guía con las proteínas precursoras del cloroplasto en las plantas" . La célula vegetal . 12 (1): 53–64. doi : 10.1105 / tpc.12.1.53 . PMC 140214 . PMID 10634907 .
- ^ Lung SC, Chuong SD (abril de 2012). "Una señal de clasificación similar a un péptido de tránsito en el extremo C dirige el receptor de preproteína de Bienertia sinuspersici Toc159 a la membrana externa del cloroplasto" . La célula vegetal . 24 (4): 1560–78. doi : 10.1105 / tpc.112.096248 . PMC 3398564 . PMID 22517318 .
- ↑ a b c d Waegemann K, Soll J (marzo de 1996). "Fosforilación de la secuencia de tránsito de proteínas precursoras de cloroplasto" . La Revista de Química Biológica . 271 (11): 6545–54. doi : 10.1074 / jbc.271.11.6545 . PMID 8626459 . S2CID 26014578 .
- ↑ a b c d e f g h i Jarvis P, Soll J (diciembre de 2001). "Importación de proteínas Toc, Tic y cloroplasto". Biochimica et Biophysica Acta (BBA) - Investigación de células moleculares . 1541 (1–2): 64–79. doi : 10.1016 / S0167-4889 (01) 00147-1 . PMID 11750663 .
- ^ Sun YJ, Forouhar F, Li Hm HM, Tu SL, Yeh YH, Kao S, Shr HL, Chou CC, Chen C, Hsiao CD (febrero de 2002). "Estructura cristalina de guisante Toc34, una nueva GTPasa del translocón de proteína de cloroplasto". Biología estructural de la naturaleza . 9 (2): 95–100. doi : 10.1038 / nsb744 . PMID 11753431 . S2CID 21855733 .
- ↑ a b c d e Agne B, Andrès C, Montandon C, Christ B, Ertan A, Jung F, Infanger S, Bischof S, Baginsky S, Kessler F (julio de 2010). "El dominio A ácido de Arabidopsis TOC159 se produce como una proteína hiperfosforilada" . Fisiología vegetal . 153 (3): 1016–30. doi : 10.1104 / pp.110.158048 . PMC 2899928 . PMID 20457805 .
- ^ a b c d e f g h i Kikuchi S, Bédard J, Hirano M, Hirabayashi Y, Oishi M, Imai M, Takase M, Ide T, Nakai M (febrero de 2013). "Descubriendo la proteína translocon en la membrana de la envoltura interna del cloroplasto". Ciencia . 339 (6119): 571–4. Código bibliográfico : 2013Sci ... 339..571K . doi : 10.1126 / science.1229262 . PMID 23372012 . S2CID 5062593 .
- ^ Curran SP, Koehler CM (2004). Función mitocondrial y biogénesis . Saltador. pag. 59. ISBN 9783540214892.