Coactivador (genética)
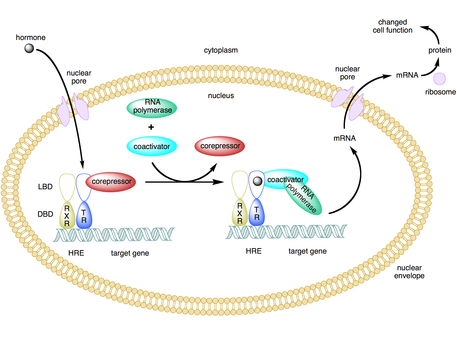
Un coactivador es un tipo de corregulador transcripcional que se une a un activador (un factor de transcripción ) para aumentar la tasa de transcripción de un gen o conjunto de genes. [1] El activador contiene un dominio de unión de ADN que se une a un sitio promotor de ADN o una secuencia reguladora de ADN específica llamada potenciador . [2] [3] La unión del complejo activador-coactivador aumenta la velocidad de transcripción al reclutar maquinaria de transcripción general para el promotor, lo que aumenta la expresión génica.. [3] [4] [5] El uso de activadores y coactivadores permite la expresión altamente específica de ciertos genes según el tipo de célula y la etapa de desarrollo. [2]
Algunos coactivadores también tienen actividad histona acetiltransferasa (HAT). Los HAT forman grandes complejos multiproteicos que debilitan la asociación de histonas con el ADN al acetilar la cola de histonas N-terminal . Esto proporciona más espacio para que la maquinaria de transcripción se una al promotor, aumentando así la expresión génica. [1] [4]
Los activadores se encuentran en todos los organismos vivos , pero las proteínas coactivadoras generalmente solo se encuentran en eucariotas porque son más complejas y requieren un mecanismo más intrincado para la regulación genética. [1] [4] En eucariotas, los coactivadores suelen ser proteínas que se localizan en el núcleo. [1] [6]
Mecanismo

Algunos coactivadores regulan indirectamente la expresión génica uniéndose a un activador e induciendo un cambio conformacional que luego permite que el activador se una a la secuencia promotora o potenciadora del ADN. [2] [7] [8] Una vez que el complejo activador-coactivador se une al potenciador, la ARN polimerasa II y otra maquinaria de transcripción general se incorporan al ADN y comienza la transcripción. [9]
Histona acetiltransferasa
El ADN nuclear normalmente se envuelve firmemente alrededor de las histonas, lo que dificulta o imposibilita que la maquinaria de transcripción acceda al ADN. Esta asociación se debe principalmente a la atracción electrostática entre el ADN y las histonas, ya que la cadena principal de fosfato del ADN está cargada negativamente y las histonas son ricas en residuos de lisina, que están cargados positivamente. [10] La estrecha asociación ADN-histona evita la transcripción del ADN en ARN.
Muchos coactivadores tienen actividad histona acetiltransferasa (HAT), lo que significa que pueden acetilar residuos de lisina específicos en las colas N-terminales de las histonas. [4] [7] [11] En este método, un activador se une a un sitio potenciador y recluta un complejo HAT que luego acetila las histonas unidas al promotor nucleosómico neutralizando los residuos de lisina cargados positivamente. [7] [11] Esta neutralización de carga hace que las histonas tengan un enlace más débil con el ADN cargado negativamente, lo que relaja la estructura de la cromatina, permitiendo que otros factores de transcripción o maquinaria de transcripción se unan al promotor (inicio de la transcripción). [4] [11]La acetilación por complejos HAT también puede ayudar a mantener la cromatina abierta durante todo el proceso de elongación, aumentando la velocidad de transcripción. [4]
La acetilación de la cola de histona N-terminal es una de las modificaciones de proteínas más comunes que se encuentran en eucariotas, con aproximadamente el 85% de todas las proteínas humanas acetiladas. [12] La acetilación es crucial para la síntesis, estabilidad, función, regulación y localización de proteínas y transcripciones de ARN. [11] [12]
Los HAT funcionan de manera similar a las acetiltransferasas N-terminales (NAT) pero su acetilación es reversible a diferencia de los NAT. [13] La acetilación de histonas mediada por HAT se invierte utilizando histona deactetilasa (HDAC), que cataliza la hidrólisis de residuos de lisina, eliminando el grupo acetilo de las histonas. [4] [7] [11] Esto hace que la cromatina vuelva a cerrarse desde su estado relajado, lo que dificulta que la maquinaria de transcripción se una al promotor, reprimiendo así la expresión génica. [4] [7]
Ejemplos de coactivadores que muestran actividad HAT incluyen CARM1 , CBP y EP300 . [14] [15]
Corepresión
Muchos coactivadores también funcionan como correpresores en determinadas circunstancias. [5] [9] Los cofactores como TAF1 y BTAF1 pueden iniciar la transcripción en presencia de un activador (actuar como coactivador) y reprimir la transcripción basal en ausencia de un activador (actuar como correpresor). [9]
Significado
Importancia biológica
La regulación transcripcional es una de las formas más comunes que tiene un organismo de alterar la expresión génica. [16] El uso de activación y coactivación permite un mayor control sobre cuándo, dónde y cuánto se produce una proteína. [1] [7] [16] Esto permite que cada célula pueda responder rápidamente a los cambios ambientales o fisiológicos y ayuda a mitigar cualquier daño que pueda ocurrir si no estuviera regulada. [1] [7]
Trastornos asociados
Las mutaciones en genes coactivadores que conducen a la pérdida o ganancia de la función proteica se han relacionado con enfermedades y trastornos como defectos de nacimiento , cáncer (especialmente cánceres dependientes de hormonas), trastornos del desarrollo neurológico y discapacidad intelectual (DI), entre muchos otros. [17] [5] La desregulación que conduce a la expresión excesiva o insuficiente de los coactivadores puede interactuar de forma perjudicial con muchos fármacos (especialmente fármacos anti-hormonales ) y se ha relacionado con el cáncer, problemas de fertilidad y trastornos del neurodesarrollo y neuropsiquiátricos . [5] Para un ejemplo específico, la desregulación deLa proteína de unión a CREB (CBP), que actúa como coactivador de numerosos factores de transcripción dentro del sistema nervioso central (SNC), el sistema reproductivo, el timo y los riñones, se ha relacionado con la enfermedad de Huntington , leucemia , síndrome de Rubinstein-Taybi , trastornos del desarrollo neurológico y déficits del sistema inmunológico , hematopoyesis y función del músculo esquelético . [14] [18]
Como objetivos de drogas
Los coactivadores son objetivos prometedores para las terapias con medicamentos en el tratamiento del cáncer, trastornos metabólicos , enfermedades cardiovasculares y diabetes tipo 2 , junto con muchos otros trastornos. [5] [19] Por ejemplo, el coactivador del receptor de esteroides (SCR) NCOA3 a menudo se sobreexpresa en el cáncer de mama , por lo que el desarrollo de una molécula inhibidora que se dirija a este coactivador y disminuya su expresión podría usarse como un tratamiento potencial para el cáncer de mama. [15] [20]
Debido a que los factores de transcripción controlan muchos procesos biológicos diferentes, son objetivos ideales para la terapia con medicamentos. [14] [21] Los coactivadores que los regulan pueden reemplazarse fácilmente con un ligando sintético que permite controlar el aumento o la disminución de la expresión génica. [14]
Los avances tecnológicos adicionales proporcionarán nuevos conocimientos sobre la función y la regulación de los coactivadores a nivel de todo el organismo y aclararán su papel en las enfermedades humanas, lo que con suerte proporcionará mejores objetivos para futuras terapias con medicamentos. [14] [15]
Coactivadores conocidos
Hasta la fecha hay más de 300 correguladores conocidos. [15] Algunos ejemplos de estos coactivadores incluyen: [22]
- ARA54 se dirige a los receptores de andrógenos
- ATXN7L3 se dirige a varios miembros de la superfamilia de receptores nucleares
- BCL3 se dirige al receptor del ácido 9-cis retinoico (RXR)
- CBP se dirige a muchos factores de transcripción
- CDC25B se dirige a los receptores de esteroides
- COPS5 se dirige a varios receptores nucleares
- DDC se dirige a los receptores de andrógenos
- EP300 se dirige a muchos factores de transcripción
- KAT5 se dirige a muchos receptores nucleares
- KDM1A se dirige a los receptores de andrógenos
- Familia de coactivadores de receptores de esteroides (SRC)
- NCOA1 se dirige a varios miembros de la superfamilia de receptores nucleares
- NCOA2 se dirige a varios miembros de la superfamilia de receptores nucleares
- NCOA3 se dirige a varios receptores nucleares y factores de transcripción
Ver también
- Represor
- Regulación de la expresión génica
- Corregulador de transcripción
- Traducción
- TcoF-DB
Referencias
- ↑ a b c d e f Courey AJ (2008). Mecanismos de regulación transcripcional . Malden, MA: Blackwell. ISBN 978-1-4051-0370-1. OCLC 173367793 .
- ^ a b c "Factor de transcripción general / factor de transcripción" . Aprenda ciencias en Scitable . Consultado el 29 de noviembre de 2017 .
- ↑ a b Pennacchio LA, Bickmore W, Dean A, Nobrega MA, Bejerano G (abril de 2013). "Potenciadores: cinco preguntas esenciales" . Nature Reviews Genética . 14 (4): 288–95. doi : 10.1038 / nrg3458 . PMC 4445073 . PMID 23503198 .
- ^ a b c d e f g h Brown CE, Lechner T, Howe L, Workman JL (enero de 2000). "Los muchos HAT de los coactivadores de transcripción". Tendencias en Ciencias Bioquímicas . 25 (1): 15–9. doi : 10.1016 / S0968-0004 (99) 01516-9 . PMID 10637607 .
- ↑ a b c d e Kumar R, O'Malley BW (2008). Correguladores de NR y enfermedades humanas . Hackensack, Nueva Jersey: World Scientific. ISBN 978-981-270-536-5. OCLC 261137374 .
- ^ Vosnakis N, Koch M, Scheer E, Kessler P, Mély Y, Didier P, Tora L (septiembre de 2017). "Los coactivadores y los factores de transcripción generales tienen dos poblaciones dinámicas distintas que dependen de la transcripción" . El diario EMBO . 36 (18): 2710–2725. doi : 10.15252 / embj.201696035 . PMC 5599802 . PMID 28724529 .
- ↑ a b c d e f g Spiegelman BM, Heinrich R (octubre de 2004). "Control biológico mediante coactivadores transcripcionales regulados" . Celular . 119 (2): 157–67. doi : 10.1016 / j.cell.2004.09.037 . PMID 15479634 . S2CID 14668705 .
- ^ Scholes NS, Weinzierl RO (mayo de 2016). "Dinámica molecular de las interacciones activador-coactivador transcripcional" difuso "" . PLOS Biología Computacional . 12 (5): e1004935. Código bibliográfico : 2016PLSCB..12E4935S . doi : 10.1371 / journal.pcbi.1004935 . PMC 4866707 . PMID 27175900 .
- ^ a b c Thomas MC, Chiang CM (mayo de 2006). "La maquinaria de transcripción general y cofactores generales". Revisiones críticas en bioquímica y biología molecular . 41 (3): 105–78. CiteSeerX 10.1.1.376.5724 . doi : 10.1080 / 10409230600648736 . PMID 16858867 . S2CID 13073440 .
- ↑ Decher, Gero (29 de agosto de 1997). "Nanoensamblajes difusos: Hacia multicompuestos poliméricos estratificados". Ciencia . 277 (5330): 1232–1237. doi : 10.1126 / science.277.5330.1232 . ISSN 0036-8075 .
- ↑ a b c d e Hermanson O, Glass CK, Rosenfeld MG (2002). "Correguladores de receptores nucleares: múltiples modos de modificación". Tendencias en endocrinología y metabolismo . 13 (2): 55–60. doi : 10.1016 / s1043-2760 (01) 00527-6 . PMID 11854019 . S2CID 38649132 .
- ^ a b Van Damme P, Hole K, Pimenta-Marques A, Helsens K, Vandekerckhove J, Martinho RG, Gevaert K, Arnesen T (julio de 2011). "NatF contribuye a un cambio evolutivo en la acetilación N-terminal de la proteína y es importante para la segregación cromosómica normal" . PLOS Genetics . 7 (7): e1002169. doi : 10.1371 / journal.pgen.1002169 . PMC 3131286 . PMID 21750686 .
- ^ Starheim KK, Gevaert K, Arnesen T (abril de 2012). "Proteínas acetiltransferasas N-terminales: cuando el comienzo importa". Tendencias en Ciencias Bioquímicas . 37 (4): 152–61. doi : 10.1016 / j.tibs.2012.02.003 . PMID 22405572 .
- ↑ a b c d e Lonard DM, O'Malley BW (octubre de 2012). "Correguladores de receptores nucleares: moduladores de patología y dianas terapéuticas" . Reseñas de la naturaleza. Endocrinología . 8 (10): 598–604. doi : 10.1038 / nrendo.2012.100 . PMC 3564250 . PMID 22733267 .
- ^ a b c d Hsia EY, Goodson ML, Zou JX, Privalsky ML, Chen HW (octubre de 2010). "Correguladores de receptores nucleares como un nuevo paradigma para la focalización terapéutica" . Revisiones avanzadas de entrega de medicamentos . 62 (13): 1227–37. doi : 10.1016 / j.addr.2010.09.016 . PMC 5004779 . PMID 20933027 .
- ^ a b "Potenciador" . Aprenda ciencias en Scitable . Consultado el 29 de noviembre de 2017 .
- ^ Berk A, Zipursky SL, Matsudaira PT, Baltimore D, Darnell J (2000). Lodish HF (ed.). Biología celular molecular (4ª ed.). Nueva York: WH Freeman. ISBN 978-0-7167-3136-8. OCLC 41266312 .
- ^ Becnel LB, Darlington YF, Orechsner S, Easton-Marks J, Watkins CA, McOwiti A, Kankanamge WH, Dehart M, Silva CM. "CBP". Moléculas NURSA . doi : 10.1621 / 8egsudafco .
- ^ "Receptores nucleares" . cursos.washington.edu . Consultado el 29 de noviembre de 2017 .
- ^ Tien JC, Xu J (noviembre de 2012). "Coactivador-3 del receptor de esteroides como potencial objetivo molecular para la terapia del cáncer" . Opinión de expertos sobre objetivos terapéuticos . 16 (11): 1085–96. doi : 10.1517 / 14728222.2012.718330 . PMC 3640986 . PMID 22924430 .
- ^ Sladek FM (octubre de 2003). "Receptores nucleares como dianas de fármacos: nuevos desarrollos en correguladores, receptores huérfanos y grandes áreas terapéuticas". Opinión de expertos sobre objetivos terapéuticos . 7 (5): 679–84. doi : 10.1517 / 14728222.7.5.679 . PMID 14498828 . S2CID 16891519 .
- ^ "NURSA - Moléculas" . nursa.org . Consultado el 30 de noviembre de 2017 .
enlaces externos
- Atlas de señalización de receptores nucleares (base de datos y consorcio de investigación financiado por los NIH; incluye una revista indexada en PubMed de acceso abierto, Señalización de receptores nucleares )
- TcoF: base de datos Dragon de cofactores de transcripción y proteínas que interactúan con el factor de transcripción
- La expresion genica
- Genética molecular
- Proteínas
- Correguladores de transcripción