La sección principal de este artículo puede ser demasiado corta para resumir adecuadamente sus puntos clave . ( Octubre de 2018 ) |
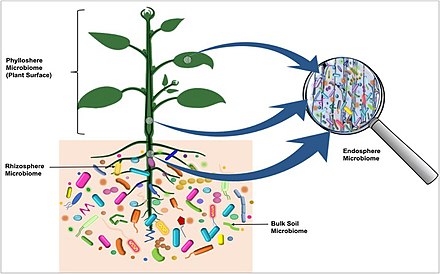
La microbiota son "comunidades ecológicas de microorganismos comensales , simbióticos y patógenos " [2] [3] que se encuentran en y sobre todos los organismos multicelulares estudiados hasta la fecha, desde plantas hasta animales. La microbiota incluye bacterias, arqueas , protistas, hongos y virus [ cita requerida ] . Se ha descubierto que la microbiota es crucial para la homeostasis inmunológica, hormonal y metabólica de su huésped. El término microbioma describe los genomas colectivos de los microorganismos que residen en un nicho ambiental o los propios microorganismos. [4][5] [6]
El microbioma y el huésped surgieron durante la evolución como una unidad sinérgica de la epigenética y las características genéticas, a las que a veces se hace referencia colectivamente como holobionte . [7] [8]
Introducción [ editar ]
Todas las plantas y animales, desde formas de vida simples hasta humanos, viven en estrecha asociación con organismos microbianos. [9] Varios avances han impulsado la percepción de los microbiomas, que incluyen:
- la capacidad de realizar análisis genómicos y de expresión génica de células individuales y de comunidades microbianas completas en las disciplinas de metagenómica y metatranscriptómica [10]
- bases de datos accesibles para investigadores de múltiples disciplinas [10]
- métodos de análisis matemático adecuados para conjuntos de datos complejos [10]
Los biólogos han llegado a apreciar que los microbios constituyen una parte importante del fenotipo de un organismo , mucho más allá del estudio de caso simbiótico ocasional. [10]
Tipos de relaciones microbio-huésped [ editar ]
El comensalismo , un concepto desarrollado por Pierre-Joseph van Beneden (1809-1894), profesor belga de la Universidad de Lovaina durante el siglo XIX [11], es fundamental para el microbioma, donde la microbiota coloniza un huésped en una coexistencia no dañina. La relación con su anfitrión se llama mutualista cuando los organismos realizan tareas que se sabe que son útiles para el anfitrión, [12] : 700 [13] parasitarias , cuando son desventajosas para el anfitrión. Otros autores definen una situación como mutualista donde ambos se benefician, y comensal, donde el anfitrión no afectado beneficia al simbionte. [14]Un intercambio de nutrientes puede ser bidireccional o unidireccional, puede depender del contexto y puede ocurrir de diversas formas. [14] La microbiota que se espera que esté presente y que, en circunstancias normales, no causa enfermedad, se considera flora normal o microbiota normal ; [12] la flora normal no solo puede ser inofensiva, sino que también puede proteger al huésped. [15]
Adquisición y cambio [ editar ]
La adquisición inicial de microbiota en animales, desde mamíferos hasta esponjas marinas, se produce al nacer e incluso puede ocurrir a través de la línea de células germinales. En las plantas, el proceso de colonización puede iniciarse bajo tierra en la zona de la raíz , alrededor de la semilla en germinación, la espermosfera , o puede originarse en las partes aéreas , la filosfera y la zona de la flor o antosfera. [16] La estabilidad de la microbiota de la rizosfera a lo largo de generaciones depende del tipo de planta, pero aún más de la composición del suelo, es decir, el entorno vivo y no vivo. [17]Clínicamente, se puede adquirir nueva microbiota a través del trasplante de microbiota fecal para tratar infecciones como la infección crónica por C. difficile . [18]
Microbiota por anfitrión [ editar ]
Humanos [ editar ]
La microbiota humana incluye bacterias , hongos , arqueas y virus. Se excluyen los microanimales que viven en el cuerpo humano. El microbioma humano se refiere a sus genomas . [12]
Los humanos están colonizados por muchos microorganismos; la estimación tradicional era que los seres humanos viven con diez veces más células no humanas que células humanas; estimaciones más recientes lo han reducido a 3: 1 e incluso a aproximadamente 1: 1. [19] [20] [21] [22]
El Proyecto del Microbioma Humano secuenció el genoma de la microbiota humana, centrándose particularmente en la microbiota que normalmente habita la piel, la boca, la nariz, el tracto digestivo y la vagina. [12] Alcanzó un hito en 2012 cuando publicó los resultados iniciales. [23]
Animales no humanos [ editar ]
- Los anfibios tienen microbiota en la piel. [24] Algunas especies son capaces de portar un hongo llamado Batrachochytrium dendrobatidis , que en otras puede causar una infección mortal Chytridiomycosis dependiendo de su microbioma, resistiendo la colonización de patógenos o inhibiendo su crecimiento con péptidos antimicrobianos de la piel. [25]
- En los mamíferos, los herbívoros, como el ganado, dependen de su microbioma ruminal para convertir la celulosa en proteínas, ácidos grasos de cadena corta y gases. Los métodos de cultivo no pueden proporcionar información sobre todos los microorganismos presentes. Los estudios metagenómicos comparativos arrojaron el sorprendente resultado de que el ganado individual posee estructuras comunitarias marcadamente diferentes, fenotipo predicho y potenciales metabólicos, [26] a pesar de que fueron alimentados con dietas idénticas, fueron alojados juntos y aparentemente fueron funcionalmente idénticos en su utilización de la pared celular vegetal. recursos.
- Los ratones se han convertido en los mamíferos más estudiados en cuanto a sus microbiomas. La microbiota intestinal se ha estudiado en relación con la enfermedad alérgica de las vías respiratorias, la obesidad, las enfermedades gastrointestinales y la diabetes. El cambio perinatal de la microbiota a través de dosis bajas de antibióticos puede tener efectos duraderos sobre la susceptibilidad futura a la enfermedad alérgica de las vías respiratorias. La frecuencia de ciertos subconjuntos de microbios se ha relacionado con la gravedad de la enfermedad. La presencia de microbios específicos en las primeras etapas de la vida posnatal instruye las respuestas inmunitarias futuras. [27] [28] En ratones gnotobióticos, se encontró que ciertas bacterias intestinales transmitían un fenotipo particular a los ratones receptores libres de gérmenes, lo que promovía la acumulación de células T reguladoras del colon y cepas que modulaban la adiposidad del ratón y las concentraciones de metabolitos cecales.[29] Este enfoque combinatorio permite una comprensión a nivel de sistemas de las contribuciones microbianas a la biología humana. [30] Pero también se han estudiado otros tejidos mucoides como el pulmón y la vagina en relación con enfermedades como el asma, la alergia y la vaginosis. [31]
- Los insectos tienen sus propios microbiomas. Por ejemplo, las hormigas cortadoras de hojas forman enormes colonias subterráneas que cosechan cientos de kilogramos de hojas cada año y no pueden digerir la celulosa de las hojas directamente. Mantienen los jardines de hongos como la principal fuente de alimento de la colonia. Si bien el hongo en sí no digiere la celulosa, una comunidad microbiana que contiene una diversidad de bacterias lo está haciendo. El análisis del genoma de la población microbiana reveló muchos genes con un papel en la digestión de la celulosa. El perfil de enzimas degradantes de carbohidratos pronosticado de este microbioma es similar al del rumen bovino, pero la composición de especies es casi completamente diferente. [32] Microbiota intestinal de la mosca de la fruta.puede afectar la apariencia de su intestino, al afectar la tasa de renovación epitelial, el espaciamiento celular y la composición de diferentes tipos de células en el epitelio. [33] Cuando la polilla Spodoptera exigua se infecta con baculovirus , los genes relacionados con la inmunidad se regulan negativamente y aumenta la cantidad de su microbiota intestinal. [34] En el intestino dípteros, las células enteroendocrinas detectan los metabolitos derivados de la microbiota intestinal y coordinan las ramas antibacterianas, mecánicas y metabólicas de la respuesta inmune innata intestinal del huésped a la microbiota comensal. [35]
- Los peces tienen sus propios microbiomas, incluida la especie de vida corta Nothobranchius furzeri ( killis turquesa). La transferencia de la microbiota intestinal de los peces muertos jóvenes a los peces muertos de mediana edad extiende significativamente la esperanza de vida de los peces muertos de mediana edad. [36]
Plantas [ editar ]
Recientemente se descubrió que el microbioma del plan se origina a partir de la semilla. [38] Los microorganismos que se transmiten a través de la semilla migran a la plántula en desarrollo en una ruta específica en la que ciertas comunidades se mueven hacia las hojas y otras hacia las raíces. [38] En el diagrama de la derecha, la microbiota que coloniza la rizosfera , ingresa a las raíces y coloniza la siguiente generación de tubérculos a través de los estolones , se visualiza con un color rojo. Las bacterias presentes en el tubérculo madre , que pasan a través de los estolones y migran a la planta, así como a la próxima generación de tubérculos, se muestran en azul. [37]
- El suelo es el principal reservorio de bacterias que colonizan los tubérculos de papa.
- Las bacterias se reclutan del suelo de manera más o menos independiente de la variedad de papa.
- Las bacterias pueden colonizar los tubérculos principalmente desde el interior de las plantas a través del estolón.
- La microbiota bacteriana de los tubérculos de papa consiste en bacterias transmitidas de una generación de tubérculos a la siguiente y las bacterias reclutadas del suelo colonizan las plantas de papa a través de la raíz. [37]
Las plantas son huéspedes atractivos para los microorganismos, ya que proporcionan una variedad de nutrientes. Los microorganismos en las plantas pueden ser epífitas (que se encuentran en las plantas) o endófitas (que se encuentran dentro del tejido vegetal). [39] [40] Los oomicetos y los hongos , a través de la evolución convergente, han desarrollado una morfología similar y ocupan nichos ecológicos similares. Desarrollan hifas , estructuras filiformes que penetran en la célula huésped. En situaciones mutualistas , la planta a menudo intercambia azúcares de hexosa por fosfato inorgánico del simbionte fúngico. Se especula que asociaciones tan antiguas han ayudado a las plantas cuando colonizaron la tierra por primera vez. [14] [41] Las bacterias promotoras del crecimiento de las plantas (PGPB) brindan a la planta servicios esenciales como la fijación de nitrógeno , la solubilización de minerales como el fósforo, la síntesis de hormonas vegetales , la mejora directa de la absorción de minerales y la protección contra patógenos. [42] [43] Las PGPB pueden proteger a las plantas de los patógenos compitiendo con el patógeno por un nicho ecológico o un sustrato, produciendo aleloquímicos inhibidores o induciendo resistencia sistémica en las plantas hospedadoras al patógeno [16]
Investigación [ editar ]
La relación simbiótica entre un huésped y su microbiota está bajo investigación de laboratorio para determinar cómo puede moldear el sistema inmunológico de los mamíferos. [44] [45] En muchos animales, el sistema inmunológico y la microbiota pueden entablar una "conversación cruzada" mediante el intercambio de señales químicas, lo que puede permitir que la microbiota influya en la reactividad inmunológica y la focalización. [46] Las bacterias se pueden transferir de madre a hijo a través del contacto directo y después del nacimiento . [47] A medida que se establece el microbioma infantil, las bacterias comensales pueblan rápidamente el intestino, provocando una variedad de respuestas inmunes y "programando" el sistema inmunológico con efectos duraderos. [46]Las bacterias pueden estimular el tejido linfoide asociado con la mucosa intestinal, lo que permite que el tejido produzca anticuerpos para los patógenos que pueden ingresar al intestino. [46]
El microbioma humano puede desempeñar un papel en la activación de receptores de tipo toll en los intestinos, un tipo de receptor de reconocimiento de patrones que las células huésped utilizan para reconocer peligros y reparar daños. Los patógenos pueden influir en esta coexistencia conduciendo a una desregulación inmunológica que incluye y susceptibilidad a enfermedades, mecanismos de inflamación , tolerancia inmunológica y enfermedades autoinmunes . [48] [49]
Co-evolución de la microbiota [ editar ]
Los organismos evolucionan dentro de los ecosistemas de modo que el cambio de un organismo afecta el cambio de otros. La teoría de la evolución del hologenoma propone que un objeto de la selección natural no es el organismo individual, sino el organismo junto con sus organismos asociados, incluidas sus comunidades microbianas.
Arrecifes de coral . La teoría del hologenoma se originó en estudios sobre arrecifes de coral. [50] Los arrecifes de coral son las estructuras más grandes creadas por organismos vivos y contienen comunidades microbianas abundantes y muy complejas. Durante las últimas décadas, se han producido importantes descensos en las poblaciones de coral. El cambio climático , la contaminación del agua y la pesca excesiva son tres factores de estrés que, según se ha descrito, conducen a la susceptibilidad a las enfermedades. Se han descrito más de veinte enfermedades diferentes de los corales, pero de ellas, solo un puñado de ellos ha tenido sus agentes causantes aislados y caracterizados. El blanqueamiento de los corales es la más grave de estas enfermedades. En el mar Mediterráneo, el blanqueamiento de Oculina patagonicase describió por primera vez en 1994 y en breve se determinó que se debía a una infección por Vibrio shiloi . De 1994 a 2002, el blanqueamiento bacteriano de O. patagonica se produjo todos los veranos en el Mediterráneo oriental. Sin embargo, sorprendentemente, después de 2003, O. patagonica en el Mediterráneo oriental ha sido resistente a la infección por V. shiloi , aunque otras enfermedades todavía causan blanqueamiento. La sorpresa proviene del conocimiento de que los corales tienen una vida larga, con una esperanza de vida del orden de décadas, [51] y no tienen un sistema inmunológico adaptativo . [ cita requerida ] Su sistema inmunológico innatono producen anticuerpos, y aparentemente no deberían ser capaces de responder a nuevos desafíos excepto en escalas de tiempo evolutivas. [ cita requerida ]
El enigma de cómo los corales lograron adquirir resistencia a un patógeno específico llevó a una propuesta de 2007, que existe una relación dinámica entre los corales y sus comunidades microbianas simbióticas. Se cree que al alterar su composición, el holobionte puede adaptarse a las condiciones ambientales cambiantes mucho más rápidamente que por mutación genética y selección solamente. La extrapolación de esta hipótesis a otros organismos, incluidas plantas y animales superiores, llevó a la propuesta de la teoría de la evolución del hologenoma. [50]
En 2007, [actualizar]la teoría del hologenoma todavía se estaba debatiendo. [52] Una crítica importante ha sido la afirmación de que V. shiloi fue identificado erróneamente como el agente causante del blanqueamiento de los corales, y que su presencia en O. patagonica blanqueada era simplemente una colonización oportunista. [53] Si esto es cierto, la observación básica que conduce a la teoría sería inválida. La teoría ha ganado una popularidad significativa como una forma de explicar los cambios rápidos en la adaptación que de otra manera no podrían explicarse por los mecanismos tradicionales de selección natural. Dentro de la teoría del hologenoma, el holobionte no solo se ha convertido en la unidad principal de la selección natural, sino también en el resultado de otro paso de integración que también se observa en la célula (simbiogénesis , endosimbiosis ) y niveles genómicos. [7]
Métodos de investigación [ editar ]
Secuenciación de amplicones dirigida [ editar ]
La secuenciación de amplicones dirigida se basa en tener algunas expectativas sobre la composición de la comunidad que se está estudiando. En la secuenciación de amplicones diana, un marcador filogenéticamente informativo se dirige a la secuenciación. Este marcador debería estar presente idealmente en todos los organismos esperados. También debería evolucionar de tal manera que se conserve lo suficiente como para que los cebadores puedan apuntar a genes de una amplia gama de organismos mientras evolucionan lo suficientemente rápido como para permitir una resolución más fina a nivel taxonómico. Un marcador común para los estudios del microbioma humano es el gen del ARNr 16S bacteriano ( es decir, "ADNr 16S", la secuencia de ADN que codifica la molécula de ARN ribosómico). [54]Dado que los ribosomas están presentes en todos los organismos vivos, el uso de ADNr 16S permite amplificar el ADN de muchos más organismos que si se usara otro marcador. El gen del rDNA 16S contiene tanto regiones de evolución lenta como regiones de rápida evolución; el primero se puede utilizar para diseñar cebadores amplios, mientras que el segundo permite una distinción taxonómica más fina. Sin embargo, la resolución a nivel de especie no suele ser posible utilizando el rDNA 16S. La selección del cebador es un paso importante, ya que todo lo que no pueda ser el objetivo del cebador no se amplificará y, por lo tanto, no se detectará. Se ha demostrado que diferentes conjuntos de cebadores amplifican diferentes grupos taxonómicos debido a la variación de secuencia.
Los estudios dirigidos a comunidades eucariotas y virales son limitados [55] y están sujetos al desafío de excluir el ADN del huésped de la amplificación y la biomasa eucariota y viral reducida en el microbioma humano. [56]
Una vez secuenciados los amplicones, se utilizan métodos filogenéticos moleculares para inferir la composición de la comunidad microbiana. Esto se hace agrupando los amplicones en unidades taxonómicas operativas (OTU) e infiriendo relaciones filogenéticas entre las secuencias. Debido a la complejidad de los datos, las medidas de distancia, como las distancias UniFrac , generalmente se definen entre muestras de microbioma, y los métodos multivariados posteriores se llevan a cabo en las matrices de distancia. Un punto importante es que la escala de datos es extensa y deben adoptarse enfoques adicionales para identificar patrones a partir de la información disponible. Las herramientas utilizadas para analizar los datos incluyen VAMPS, [57] QIIME [58] y mothur. [59]
Secuenciación metagenómica [ editar ]
La metagenómica también se usa ampliamente para estudiar comunidades microbianas. [60] [61] [62] En la secuenciación metagenómica, el ADN se recupera directamente de muestras ambientales de una manera no dirigida con el objetivo de obtener una muestra imparcial de todos los genes de todos los miembros de la comunidad. Estudios recientes utilizan secuenciación de escopeta Sanger o pirosecuenciación para recuperar las secuencias de las lecturas. [63] Las lecturas se pueden ensamblar en contigs . Para determinar la identidad filogenética de una secuencia, se compara con las secuencias del genoma completo disponibles utilizando métodos como BLAST. Un inconveniente de este enfoque es que muchos miembros de las comunidades microbianas no tienen un genoma secuenciado representativo, pero esto se aplica también a la secuenciación de amplicones de ARNr 16S y es un problema fundamental. [54] Con la secuenciación de escopeta, se puede resolver teniendo una alta cobertura (50-100x) del genoma desconocido, realizando efectivamente un ensamblaje genómico de novo . Tan pronto como se disponga de un genoma completo de un organismo desconocido, se puede comparar filogenéticamente y colocar el organismo en su lugar en el árbol de la vida mediante la creación de nuevos taxones . Un enfoque emergente es combinar la secuenciación de escopeta con datos de ligadura de proximidad ( Hi-C ) para ensamblar genomas microbianos completos sin cultivar. [64]
A pesar de que la metagenómica está limitada por la disponibilidad de secuencias de referencia, una ventaja significativa de la metagenómica sobre la secuenciación de amplicones dirigida es que los datos de la metagenómica pueden dilucidar el potencial funcional del ADN de la comunidad. [65] [66] Los estudios de genes dirigidos no pueden hacer esto, ya que solo revelan la relación filogenética entre el mismo gen de diferentes organismos. El análisis funcional se realiza comparando las secuencias recuperadas con bases de datos de anotaciones metagenómicas como KEGG . Las vías metabólicas que estos genes están implicados en la continuación, se puede predecir con herramientas como MG-RAST, [67] CÁMARA [68] y IMG / M . [69]
Enfoques basados en ARN y proteínas [ editar ]
Se han realizado estudios de metatranscriptómica para estudiar la expresión génica de comunidades microbianas mediante métodos como la pirosecuenciación del ARN extraído. [70] Los estudios basados en la estructura también han identificado ARN no codificantes (ncRNA) como las ribozimas de la microbiota. [71] La metaproteómica es un enfoque que estudia las proteínas expresadas por la microbiota, dando una idea de su potencial funcional. [72]
Proyectos [ editar ]
El Proyecto de Microbioma Humano lanzado en 2008 fue una iniciativa de los Institutos Nacionales de Salud de los Estados Unidos para identificar y caracterizar los microorganismos que se encuentran tanto en humanos sanos como enfermos. [73] El proyecto de cinco años, mejor caracterizado como un estudio de viabilidad con un presupuesto de $ 115 millones, probó cómo los cambios en el microbioma humano están asociados con la salud o la enfermedad humana. [73]
El Earth Microbiome Project (EMP) es una iniciativa para recolectar muestras naturales y analizar la comunidad microbiana de todo el mundo. Los microbios son muy abundantes, diversos y tienen un papel importante en el sistema ecológico. Sin embargo, a partir de 2010 [actualizar], se estimó que el esfuerzo de secuenciación del ADN ambiental global total había producido menos del 1 por ciento del ADN total encontrado en un litro de agua de mar o un gramo de suelo, [74] y las interacciones específicas entre microbios son en gran parte desconocidas. . El EMP tiene como objetivo procesar hasta 200,000 muestras en diferentes biomas, generando una base de datos completa de microbios en la tierra para caracterizar ambientes y ecosistemas por composición e interacción microbiana. Con estos datos, se pueden proponer y probar nuevas teorías ecológicas y evolutivas.[75]
Microbiota intestinal y diabetes tipo 2 [ editar ]
La microbiota intestinal es muy importante para la salud del huésped porque juega un papel en la degradación de polisacáridos no digeribles (fermentación de almidón resistente, oligosacáridos, inulina) fortaleciendo la integridad intestinal o dando forma al epitelio intestinal, recolectando energía, protegiendo contra patógenos y regulando al huésped. inmunidad. [76] [77]
Varios estudios mostraron que la composición bacteriana intestinal en pacientes diabéticos se alteró con niveles elevados de Lactobacillus gasseri , Streptococcus mutans y miembros de Clostridiales, con disminución de bacterias productoras de butirato como Roseburia intestinalis y Faecalibacterium prausnitzii [78] [79] . esta alteración se debe a muchos factores como el abuso de antibióticos, la dieta y la edad .
La disminución en la producción de butirato se asocia con un defecto en la permeabilidad intestinal, este defecto conduce al caso de la endotoxemia, que es el aumento del nivel de lipopolisacáridos circulantes de la pared de las células bacterianas gramnegativas. Se encuentra que la endotoxemia se asocia con el desarrollo de resistencia a la insulina. [78]
Además, la producción de butirato afecta el nivel de serotonina. [78] El nivel elevado de serotonina contribuye a la obesidad, que se sabe que es un factor de riesgo para el desarrollo de diabetes.
La microbiota se puede trasplantar en el cuerpo humano con fines médicos. [80]
Desarrollo de la microbiota intestinal y antibióticos [ editar ]
La colonización de la microbiota intestinal humana puede comenzar ya antes del nacimiento. [81] Hay múltiples factores en el medio ambiente que afectan el desarrollo de la microbiota, siendo el modo de nacimiento uno de los más impactantes. [82]
Otro factor que se ha observado que causa grandes cambios en la microbiota intestinal, particularmente en los niños, es el uso de antibióticos, que se asocia con problemas de salud como un IMC más alto, [83] [84] y un mayor riesgo de padecer enfermedades metabólicas como obesidad. [85] En los lactantes se observó que la amoxicilina y los macrólidos causan cambios significativos en la microbiota intestinal caracterizados por un cambio en las clases bacterianas Bifidobacteria, Enterobacteria y Clostridia. [86] Un solo ciclo de antibióticos en adultos provoca cambios en la microbiota bacteriana y fúngica, con cambios aún más persistentes en las comunidades fúngicas. [87]Las bacterias y los hongos viven juntos en el intestino y es muy probable que exista una competencia por las fuentes de nutrientes presentes. [88] [89] Seelbinder y col . encontraron que las bacterias comensales en el intestino regulan el crecimiento y patogenicidad de Candida albicans por sus metabolitos, particularmente por propionato, ácido acético y 5-dodecenoato. [90] Candida se ha asociado previamente con EII [91] y, además, se ha observado que aumenta en personas que no responden a un fármaco biológico, infliximab, administrado a pacientes con EII que padecen EII grave. [92]El propionato y el ácido acético son ácidos grasos de cadena corta (AGCC) que se ha observado que son beneficiosos para la salud de la microbiota intestinal. [93] [94] [95] Cuando los antibióticos afectan el crecimiento de bacterias en el intestino, puede haber un crecimiento excesivo de ciertos hongos, que pueden ser patógenos cuando no están regulados. [96]
Problemas de privacidad [ editar ]
El ADN microbiano que habita el cuerpo humano de una persona puede identificar de forma única a la persona. La privacidad de una persona puede verse comprometida si la persona dona de forma anónima datos de ADN de microbios. Su condición médica e identidad podrían revelarse. [97] [98] [99]
Ver también [ editar ]
- Anagénesis
- Bioma
- Viroma humano
- Lista de microbiota bacteriana de la vaginosis
- Microbiota marina
- Microbiota del tracto reproductivo inferior de la mujer.
- Fitobioma
- Probiótico
- Psicobiótico
- Flora cutánea
- Flora vaginal
- Microbiota vaginal en el embarazo
Referencias [ editar ]
- ^ Dastogeer, KM, Tumpa, FH, Sultana, A., Akter, MA y Chakraborty, A. (2020) "Microbioma vegetal: una descripción de los factores que dan forma a la composición y diversidad de la comunidad". Biología vegetal actual , 23 : 100161. doi : 10.1016 / j.cpb.2020.100161 . El material se copió de esta fuente, que está disponible bajo una licencia internacional Creative Commons Attribution 4.0 .
- ^ Lederberg, J; McCray, AT (2001). " ' Ome Sweet' Omics - un tesoro genealógico de palabras" . Científico . 15 : 8.
- ^ Grupo de trabajo de NIH HMP; Peterson, J; Garges, S; et al. (2009). "El Proyecto del Microbioma Humano de los NIH" . Genome Res . 19 (12): 2317–2323. doi : 10.1101 / gr.096651.109 . PMC 2792171 . PMID 19819907 .
- ^ Backhed, F .; Ley, RE; Sonnenburg, JL; Peterson, DA; Gordon, JI (2005). "Mutualismo huésped-bacteriano en el intestino humano". Ciencia . 307 (5717): 1915–1920. Código Bibliográfico : 2005Sci ... 307.1915B . doi : 10.1126 / science.1104816 . PMID 15790844 . S2CID 6332272 .
- ^ Turnbaugh, PJ; Ley, RE; Hamady, M .; Fraser-Liggett, CM; Knight, R .; Gordon, JI (2007). "El Proyecto del Microbioma Humano" . Naturaleza . 449 (7164): 804–810. Código Bibliográfico : 2007Natur.449..804T . doi : 10.1038 / nature06244 . PMC 3709439 . PMID 17943116 .
- ^ Ley, RE; Peterson, DA; Gordon, JI (2006). "Fuerzas ecológicas y evolutivas que dan forma a la diversidad microbiana en el intestino humano". Celular . 124 (4): 837–848. doi : 10.1016 / j.cell.2006.02.017 . PMID 16497592 . S2CID 17203181 .
- ↑ a b Salvucci, E. (2016). "Microbioma, holobionte y la red de la vida". Revisiones críticas en microbiología . 42 (3): 485–494. doi : 10.3109 / 1040841X.2014.962478 . PMID 25430522 . S2CID 30677140 .
- ↑ Guerrero, R .; Margulis, Lynn ; Berlanga, M. (2013). "Simbiogénesis: El holobionte como unidad de evolución". Microbiología internacional . 16 (3): 133–43. doi : 10.2436 / 20.1501.01.188 . PMID 24568029 .
- ↑ Mendes, R .; Raaijmakers, JM (2015). "Similitudes entre reinos en las funciones del microbioma" . El diario ISME . 9 (9): 1905-1907. doi : 10.1038 / ismej.2015.7 . PMC 4542044 . PMID 25647346 .
- ^ a b c d Bosch, TCG; McFall-Ngai, MJ (2011). "Metaorganismos como la nueva frontera" . Zoología . 114 (4): 185-190. doi : 10.1016 / j.zool.2011.04.001 . PMC 3992624 . PMID 21737250 .
- ^ Poreau B., Biologie et complexité: histoire et modèles du commensalisme . Tesis de Doctorado, Universidad de Lyon, Francia, 2014.
- ^ a b c d Sherwood, Linda; Willey, Joanne; Woolverton, Christopher (2013). Microbiología de Prescott (9ª ed.). Nueva York: McGraw Hill. págs. 713–721. ISBN 9780073402406. OCLC 886600661 .
- ^ Quigley, EM (septiembre de 2013). "Las bacterias intestinales en la salud y la enfermedad" . Gastroenterol Hepatol (NY) . 9 (9): 560–9. PMC 3983973 . PMID 24729765 .
- ↑ a b c Remy W, Taylor TN, Hass H, Kerp H (1994). "Micorrizas vesiculares arbusculares de cuatrocientos millones de años" . Proc. Natl. Acad. Sci. USA . 91 (25): 11841–3. Código Bibliográfico : 1994PNAS ... 9111841R . doi : 10.1073 / pnas.91.25.11841 . PMC 45331 . PMID 11607500 .
- ^ Copeland, CS (septiembre-octubre de 2017). "El mundo dentro de nosotros" . Revista de salud de Nueva Orleans .
- ↑ a b Compant S, Duffy B, Nowak J, Clément C, Barka EA (2005). "Uso de bacterias promotoras del crecimiento vegetal para el control biológico de enfermedades de las plantas: principios, mecanismos de acción y perspectivas futuras" . Appl Environ Microbiol . 71 (9): 4951–9. doi : 10.1128 / AEM.71.9.4951-4959.2005 . PMC 1214602 . PMID 16151072 .
- ^ Tkacz, Andrzej; Cheema, Jitender; Chandra, Govind; Grant, Alastair; Poole, Philip S. (noviembre de 2015). "La estabilidad y sucesión de la microbiota de la rizosfera depende del tipo de planta y la composición del suelo" . ISME J . 9 (11): 2349–2359. doi : 10.1038 / ismej.2015.41 . PMC 4611498 . PMID 25909975 .
- ^ Copeland, CS (19 de abril de 2019). "¿Qué es el Clostridium difficile?" . Vitalacy .
- ^ Preguntas frecuentes de la Academia Estadounidense de Microbiología : Microbioma humano Archivado el 31 de diciembre de 2016en Wayback Machine, enero de 2014
- ^ Judah L. Rosner para Microbe Magazine, febrero de 2014. ¿ Diez veces más células microbianas que células corporales en los seres humanos?
- ^ Alison Abbott para Nature News. 8 de enero de 2016 Los científicos rompen el mito de que nuestros cuerpos tienen más bacterias que células humanas
- ^ Remitente, R; Fuchs, S; Milo, R (enero de 2016). "¿Realmente nos superan en número? Revisando la proporción de bacterias a las células huésped en los seres humanos". Celular . 164 (3): 337–40. doi : 10.1016 / j.cell.2016.01.013 . PMID 26824647 . S2CID 1790146 .
- ^ "Proyecto de microbioma humano de NIH define la composición bacteriana normal del cuerpo" . NIH News. 13 de junio de 2012.
- ^ Bataille, A; Lee-Cruz, L; Tripathi, B; Kim, H; Waldman, B (enero de 2016). "Variación del microbioma a través de regiones de piel de anfibios: implicaciones para los esfuerzos de mitigación de la quitridiomicosis". Microb. Ecol . 71 (1): 221–32. doi : 10.1007 / s00248-015-0653-0 . PMID 26271741 . S2CID 12951957 .
- ^ Woodhams DC, Rollins-Smith LA, Alford RA, Simon MA, Harris RN (2007). "Defensas inmunes innatas de la piel de anfibios: péptidos antimicrobianos y más". Conservación animal . 10 (4): 425–8. doi : 10.1111 / j.1469-1795.2007.00150.x .
- ^ Brulc JM; Antonopoulos DA; Miller MEB; et al. (2009). "La metagenómica centrada en el gen del microbioma del rumen bovino adherente a la fibra revela hidrolasas de glucósidos específicos del forraje" . Proc. Natl. Acad. Sci. USA . 106 (6): 1948–53. Código bibliográfico : 2009PNAS..106.1948B . doi : 10.1073 / pnas.0806191105 . PMC 2633212 . PMID 19181843 .
- ^ Russell SL, Gold MJ; et al. (Mayo de 2012). "Los cambios en la microbiota impulsados por antibióticos en la vida temprana mejoran la susceptibilidad al asma alérgica" . Rep . EMBO 13 (5): 440–7. doi : 10.1038 / embor.2012.32 . PMC 3343350 . PMID 22422004 .
- ^ Russell SL, Gold MJ, et al. (Agosto de 2014). "Los cambios inducidos por antibióticos perinatales en la microbiota intestinal tienen efectos diferenciales sobre las enfermedades pulmonares inflamatorias". J Allergy Clin Immunol . 135 (1): 100–9. doi : 10.1016 / j.jaci.2014.06.027 . PMID 25145536 .
- ^ Turnbaugh PJ, et al. (Diciembre de 2006). "Un microbioma intestinal asociado a la obesidad con mayor capacidad de recolección de energía". Naturaleza . 444 (7122): 1027–31. Código Bib : 2006Natur.444.1027T . doi : 10.1038 / nature05414 . PMID 17183312 . S2CID 4400297 .
- ^ Faith JJ, Ahern PP, Ridaura VK, et al. (Enero de 2014). "Identificación de las relaciones del fenotipo del huésped-microbio intestinal utilizando comunidades combinatorias en ratones gnotobióticos" . Sci. Transl. Med . 6 (220): 220. doi : 10.1126 / scitranslmed.3008051 . PMC 3973144 . PMID 24452263 .
- ^ Barfod, KK; Roggenbuck, M; Hansen, LH; Schjørring, S; Larsen, ST; Sørensen, SJ; Krogfelt, KA (2013). "El microbioma pulmonar murino en relación con las comunidades bacterianas intestinales y vaginales" . BMC Microbiol . 13 : 303. doi : 10.1186 / 1471-2180-13-303 . PMC 3878784 . PMID 24373613 .
- ^ Suen; Scott JJ; Aylward FO; et al. (2010). Sonnenburg, Justin (ed.). "Un microbioma de insectos herbívoros con alta capacidad de degradación de biomasa vegetal" . PLOS Genet . 6 (9): e1001129. doi : 10.1371 / journal.pgen.1001129 . PMC 2944797 . PMID 20885794 .
- ^ Broderick, Nichole A .; Buchon, Nicolas; Lemaitre, Bruno (2014). "Cambios inducidos por la microbiota en la expresión del gen huésped de Drosophila melanogaster y la morfología intestinal" . mBio . 5 (3): e01117–14. doi : 10.1128 / mBio.01117-14 . PMC 4045073 . PMID 24865556 .
- ↑ Jakubowska, Agata K .; Vogel, Heiko; Herrero, Salvador (mayo de 2013). "Aumento de la microbiota intestinal después de la supresión inmune en larvas infectadas por baculovirus" . PLOS Pathog . 9 (5): e1003379. doi : 10.1371 / journal.ppat.1003379 . PMC 3662647 . PMID 23717206 .
- ^ Watnick, Paula I .; Jugder, Bat-Erdene (1 de febrero de 2020). "Control microbiano de la homeostasis intestinal a través de la señalización inmune innata de células enteroendocrinas" . Tendencias en microbiología . 28 (2): 141-149. doi : 10.1016 / j.tim.2019.09.005 . ISSN 0966-842X . PMC 6980660 . PMID 31699645 .
- ^ Tibbs TN, Lopez LR, Arthur JC (2019). "La influencia de la microbiota en el desarrollo inmunológico, la inflamación crónica y el cáncer en el contexto del envejecimiento" . Célula microbiana . 6 (8): 324–334. doi : 10.15698 / mic2019.08.685 . PMC 6685047 . PMID 31403049 .
- ^ a b c Buchholz, F., Antonielli, L., Kostić, T., Sessitsch, A. y Mitter, B. (2019) "La comunidad bacteriana en la papa se recluta del suelo y se hereda parcialmente a través de generaciones". PLOS One , 14 (11): e0223691. doi : 10.1371 / journal.pone.0223691 . El material se copió de esta fuente, que está disponible bajo una licencia internacional Creative Commons Attribution 4.0 .
- ^ a b Abdelfattah, Ahmed; Wisniewski, Michael; Schena, Leonardo; Tack, Ayco JM "Evidencia experimental de herencia microbiana en plantas y rutas de transmisión de semilla a filosfera y raíz" . Microbiología ambiental . n / a (n / a). doi : 10.1111 / 1462-2920.15392 . ISSN 1462-2920 .
- ↑ Berlec, Aleš (1 de septiembre de 2012). "Nuevas técnicas y hallazgos en el estudio de la microbiota vegetal: búsqueda de probióticos vegetales". Ciencia de las plantas . 193-194: 96-102. doi : 10.1016 / j.plantsci.2012.05.010 . PMID 22794922 .
- ^ Látigos, Jm; Hand, P .; Pink, D .; Doblando, Di-s (2008-12-01). "Microbiología de la filosfera con especial referencia a la diversidad y el genotipo vegetal" (PDF) . Revista de microbiología aplicada . 105 (6): 1744-1755. doi : 10.1111 / j.1365-2672.2008.03906.x . ISSN 1365-2672 . PMID 19120625 . S2CID 35055151 .
- ^ Chibucos MC, Tyler BM (2009). "Temas comunes en la adquisición de nutrientes por microbios simbióticos de plantas, descritos por el Gene Ontology" . Microbiología BMC . 9 (Supl. 1): S6. doi : 10.1186 / 1471-2180-9-S1-S6 . PMC 2654666 . PMID 19278554 .
- ^ Kloepper, J. W (1993). "Rizobacterias promotoras del crecimiento vegetal como agentes de control biológico". En Metting, FB Jr (ed.). Ecología microbiana del suelo: aplicaciones en la gestión agrícola y medioambiental . Nueva York: Marcel Dekker Inc. págs. 255–274. ISBN 978-0-8247-8737-0.
- ^ Bloemberg, GV; Lugtenberg, BJJ (2001). "Bases moleculares de la promoción del crecimiento vegetal y el biocontrol por rizobacterias". Opinión actual en biología vegetal . 4 (4): 343–350. doi : 10.1016 / S1369-5266 (00) 00183-7 . PMID 11418345 .
- ^ Palm, Noah W .; de Zoete, Marcel R .; Flavell, Richard A. (30 de junio de 2015). "Interacciones inmuno-microbiota en salud y enfermedad" . Inmunología clínica . 159 (2): 122-127. doi : 10.1016 / j.clim.2015.05.014 . ISSN 1521-6616 . PMC 4943041 . PMID 26141651 .
- ^ Ronda, junio L .; O'Connell, Ryan M .; Mazmanian, Sarkis K. (2010). "Coordinación de respuestas inmunes tolerogénicas por la microbiota comensal" . Revista de autoinmunidad . 34 (3): J220 – J225. doi : 10.1016 / j.jaut.2009.11.007 . PMC 3155383 . PMID 19963349 .
- ^ a b c Cahenzli, Julia; Balmer, Maria L .; McCoy, Kathy D. (2012). "Intercomunicación microbiana-inmune y regulación del sistema inmunológico" . Inmunologia . 138 (1): 12-22. doi : 10.1111 / j.1365-2567.2012.03624.x . PMC 3533697 . PMID 22804726 .
- ^ Rosenberg, Eugene; Zilber-Rosenberg, Ilana (2016). "Los microbios impulsan la evolución de animales y plantas: el concepto de hologenoma" . mBio . 7 (2): e01395–15. doi : 10.1128 / mbio.01395-15 . PMC 4817260 . PMID 27034283 .
- ^ Blander, J Magarian; Longman, Randy S; Iliev, Iliyan D; Sonnenberg, Gregory F; Artis, David (19 de julio de 2017). "Regulación de la inflamación por interacciones de la microbiota con el huésped" . Inmunología de la naturaleza . 18 (8): 851–860. doi : 10.1038 / ni.3780 . ISSN 1529-2908 . PMC 5800875 . PMID 28722709 .
- ^ Nikoopour, E; Singh, B (2014). "Reciprocidad en las interacciones del microbioma y el sistema inmunológico y sus implicaciones en la enfermedad y la salud" . Blancos de medicamentos para la alergia a Inflamm . 13 (2): 94-104. doi : 10.2174 / 1871528113666140330201056 . PMID 24678760 .
- ↑ a b Rosenberg E, Koren O, Reshef L, Efrony R, Zilber-Rosenberg I (2007). "El papel de los microorganismos en la salud, enfermedad y evolución de los corales". Nature Reviews Microbiología . 5 (5): 355–362. doi : 10.1038 / nrmicro1635 . PMID 17384666 . S2CID 2967190 .
- ^ Baird AH, Bhagooli R, Ralph PJ, Takahashi S (2009). "Blanqueamiento de corales: el papel del anfitrión" (PDF) . Tendencias en ecología y evolución . 24 (1): 16-20. doi : 10.1016 / j.tree.2008.09.005 . PMID 19022522 .
- ^ Leggat W, Ainsworth T, Bythell J, Dove S, Gates R, Hoegh-Guldberg O, Iglesias-Prieto R, Yellowlees D (2007). "La teoría del hologenoma ignora el holobionte de coral". Nature Reviews Microbiología . 5 (10): Correspondencia en línea. doi : 10.1038 / nrmicro1635-c1 . S2CID 9031305 .
- ↑ Ainsworth TD, Fine M, Roff G, Hoegh-Guldberg O (2008). "Las bacterias no son la principal causa de blanqueamiento en el coral mediterráneo Oculina patagonica ". El diario ISME . 2 (1): 67–73. doi : 10.1038 / ismej.2007.88 . PMID 18059488 . S2CID 1032896 .
- ↑ a b Kuczynski, J .; Lauber, CL; Walters, WA; Parfrey, LW; Clemente, JC; Gevers, D .; Caballero, R. (2011). "Herramientas experimentales y analíticas para el estudio del microbioma humano" . Nature Reviews Genética . 13 (1): 47–58. doi : 10.1038 / nrg3129 . PMC 5119550 . PMID 22179717 .
- ^ Marchesi, JR (2010). "Diversidad procariota y eucariota del intestino humano". Avances en microbiología aplicada Volumen 72 . Avances en microbiología aplicada. 72 . págs. 43–62. doi : 10.1016 / S0065-2164 (10) 72002-5 . ISBN 9780123809896. PMID 20602987 .
- ↑ Vestheim, H .; Jarman, SN (2008). "Bloqueo de cebadores para mejorar la amplificación por PCR de secuencias raras en muestras mixtas: un estudio de caso sobre ADN de presa en estómagos de krill antártico" . Fronteras en Zoología . 5 : 12. doi : 10.1186 / 1742-9994-5-12 . PMC 2517594 . PMID 18638418 .
- ^ "VAMPS: la visualización y análisis de estructuras de población microbiana" . Bay Paul Center, MBL, Woods Hole . Consultado el 11 de marzo de 2012 .
- ^ Caporaso, JG; Kuczynski, J .; Stombaugh, J .; Bittinger, K .; Bushman, FD; Costello, EK; Fierer, N .; Peña, AG; Goodrich, JK; Gordon, JI; Huttley, GA; Kelley, ST; Caballeros, D .; Koenig, JE; Ley, RE; Lozupone, CA; McDonald, D .; Muegge, BD; Pirrung, M .; Reeder, J .; Sevinsky, JR; Turnbaugh, PJ; Walters, WA; Widmann, J .; Yatsunenko, T .; Zaneveld, J .; Caballero, R. (2010). "QIIME permite el análisis de datos de secuenciación de la comunidad de alto rendimiento" . Métodos de la naturaleza . 7 (5): 335–336. doi : 10.1038 / nmeth.f.303 . PMC 3156573 . PMID 20383131 .
- ^ Schloss, PD; Westcott, SL; Ryabin, T .; Hall, JR; Hartmann, M .; Hollister, EB; Lesniewski, RA; Oakley, BB; Parks, DH; Robinson, CJ; Sahl, JW; Stres, B .; Thallinger, GG; Van Horn, DJ; Weber, CF (2009). "Presentación de mothur: software de código abierto, independiente de la plataforma, apoyado por la comunidad para describir y comparar comunidades microbianas" . Microbiología aplicada y ambiental . 75 (23): 7537–7541. doi : 10.1128 / AEM.01541-09 . PMC 2786419 . PMID 19801464 .
- ^ Turnbaugh, PJ; Hamady, M .; Yatsunenko, T .; Cantarel, BL; Duncan, A .; Ley, RE; Sogin, ML; Jones, WJ; Roe, BA; Affourtit, JP; Egholm, M .; Henrissat, B .; Heath, AC; Knight, R .; Gordon, JI (2008). "Un microbioma intestinal central en gemelos obesos y delgados" . Naturaleza . 457 (7228): 480–484. Código Bibliográfico : 2009Natur.457..480T . doi : 10.1038 / nature07540 . PMC 2677729 . PMID 19043404 .
- ^ Qin, J .; Li, R .; Raes, J .; Arumugam, M .; Burgdorf, KS; Manichanh, C .; Nielsen, T .; Pons, N .; Levenez, F .; Yamada, T .; Mende, DR; Li, J .; Xu, J .; Li, S .; Li, D .; Cao, J .; Wang, B .; Liang, H .; Zheng, H .; Xie, Y .; Tap, J .; Lepage, P .; Bertalan, M .; Batto, JM; Hansen, T .; Le Paslier, D .; Linneberg, A .; Nielsen, HBR; Pelletier, E .; Renault, P. (2010). "Un catálogo de genes microbianos del intestino humano establecido por secuenciación metagenómica" . Naturaleza . 464 (7285): 59–65. Código Bibliográfico : 2010Natur.464 ... 59. . doi : 10.1038 / nature08821 . PMC 3779803 . PMID 20203603 .
- ^ Tringe, SG; Von Mering, C .; Kobayashi, A .; Salamov, AA; Chen, K .; Chang, HW; Podar, M .; Corto, JM; Mathur, EJ; Detter, JC; Bork, P .; Hugenholtz, P .; Rubin, EM (2005). "Metagenómica comparativa de comunidades microbianas". Ciencia . 308 (5721): 554–557. Código Bibliográfico : 2005Sci ... 308..554T . CiteSeerX 10.1.1.377.2288 . doi : 10.1126 / science.1107851 . PMID 15845853 . S2CID 161283 .
- ^ Wooley, JC; Godzik, A .; Friedberg, I. (2010). Bourne, Philip E. (ed.). "Una introducción a la metagenómica" . PLOS Biología Computacional . 6 (2): e1000667. Código bibliográfico : 2010PLSCB ... 6E0667W . doi : 10.1371 / journal.pcbi.1000667 . PMC 2829047 . PMID 20195499 .
- ^ Watson, Mick; Roehe, Rainer; Walker, Alan W .; Dewhurst, Richard J .; Snelling, Timothy J .; Ivan Liachko; Langford, Kyle W .; Prensa, Maximilian O .; Más sabio, Andrew H. (28 de febrero de 2018). "Ensamblaje de 913 genomas microbianos a partir de la secuenciación metagenómica del rumen de la vaca" . Comunicaciones de la naturaleza . 9 (1): 870. Bibcode : 2018NatCo ... 9..870S . doi : 10.1038 / s41467-018-03317-6 . ISSN 2041-1723 . PMC 5830445 . PMID 29491419 .
- ^ Muller, J .; Szklarczyk, D .; Julien, P .; Letunic, I .; Roth, A .; Kuhn, M .; Powell, S .; Von Mering, C .; Doerks, T .; Jensen, LJ; Bork, P. (2009). "EggNOG v2.0: ampliar la genealogía evolutiva de genes con grupos ortólogos no supervisados mejorados, especies y anotaciones funcionales" . Investigación de ácidos nucleicos . 38 (Problema de la base de datos): D190 – D195. doi : 10.1093 / nar / gkp951 . PMC 2808932 . PMID 19900971 .
- ^ Kanehisa, M .; Ve a S.; Furumichi, M .; Tanabe, M .; Hirakawa, M. (2009). "KEGG para la representación y análisis de redes moleculares que involucran enfermedades y medicamentos" . Investigación de ácidos nucleicos . 38 (Problema de la base de datos): D355 – D360. doi : 10.1093 / nar / gkp896 . PMC 2808910 . PMID 19880382 .
- ^ Meyer, F .; Paarmann, D .; d'Souza, M .; Olson, R .; Vidrio, EM; Kubal, M .; Paczian, T .; Rodríguez, A .; Stevens, R .; Wilke, A .; Wilkening, J .; Edwards, RA (2008). "El servidor RAST de metagenómica - un recurso público para el análisis filogenético y funcional automático de metagenomas" . BMC Bioinformática . 9 : 386. doi : 10.1186 / 1471-2105-9-386 . PMC 2563014 . PMID 18803844 .
- ^ Sol, S .; Chen, J .; Li, W .; Altintas, I .; Lin, A .; Peltier, S .; Acciones, K .; Allen, EE; Ellisman, M .; Grethe, J .; Wooley, J. (2010). "Ciberinfraestructura comunitaria para la investigación y el análisis de ecología microbiana avanzada: el recurso CAMERA" . Investigación de ácidos nucleicos . 39 (Problema de la base de datos): D546 – D551. doi : 10.1093 / nar / gkq1102 . PMC 3013694 . PMID 21045053 .
- ^ Markowitz, VM; Ivanova, NN; Szeto, E .; Palaniappan, K .; Chu, K .; Dalevi, D .; Chen, IMA; Grechkin, Y .; Dubchak, I .; Anderson, I .; Lykidis, A .; Mavromatis, K .; Hugenholtz, P .; Kyrpides, Carolina del Norte (2007). "IMG / M: un sistema de análisis y gestión de datos para metagenomas" . Investigación de ácidos nucleicos . 36 (Problema de la base de datos): D534 – D538. doi : 10.1093 / nar / gkm869 . PMC 2238950 . PMID 17932063 .
- ^ Shi, Y .; Tyson, GW; Delong, EF (2009). "La metatranscriptómica revela pequeños ARN microbianos únicos en la columna de agua del océano". Naturaleza . 459 (7244): 266–269. Código Bibliográfico : 2009Natur.459..266S . doi : 10.1038 / nature08055 . PMID 19444216 . S2CID 4340144 .
- ^ Jiménez, RM; Delwart, E .; Luptak, A (2011). "Búsqueda basada en estructura revela ribozimas martillo en el microbioma humano" . Revista de Química Biológica . 286 (10): 7737–7743. doi : 10.1074 / jbc.C110.209288 . PMC 3048661 . PMID 21257745 .
- ^ Maron, PA; Ranjard, L .; Mougel, C .; Lemanceau, P. (2007). "Metaproteómica: un nuevo enfoque para estudiar la ecología microbiana funcional". Ecología microbiana . 53 (3): 486–493. doi : 10.1007 / s00248-006-9196-8 . PMID 17431707 . S2CID 26953155 .
- ^ a b "Proyecto de microbioma humano NIH" . Institutos Nacionales de Salud de EE. UU., Departamento de Salud y Servicios Humanos, Gobierno de EE. UU. 2016. Archivado desde el original el 11 de junio de 2016 . Consultado el 14 de junio de 2016 .
- ^ Gilbert, JA; Meyer, F .; Antonopoulos, D .; et al. (2010). "Informe de la reunión: el taller de metagenómica de Terabase y la visión de un proyecto de microbioma terrestre" . Estándares en Ciencias Genómicas . 3 (3): 243–248. doi : 10.4056 / sigs.1433550 . PMC 3035311 . PMID 21304727 .
- ^ Gilbert, JA; O'Dor, R .; King, N .; Vogel, TM (2011). "La importancia de los estudios metagenómicos para la ecología microbiana: o por qué Darwin habría sido un científico metagenómico" . Informática y Experimentación Microbiana . 1 (1): 5. doi : 10.1186 / 2042-5783-1-5 . PMC 3348666 . PMID 22587826 .
- ↑ Ibrahim, Nesma (1 de julio de 2018). "Microbiota intestinal y diabetes mellitus tipo 2: ¿Cuál es el vínculo?" . Revista Afro-Egipcia de Enfermedades Infecciosas y Endémicas . 6 (2): 112-119. doi : 10.21608 / aeji.2018.9950 . ISSN 2090-7184 .
- ^ Jueves, Isabel; Juge, Nathalie (1 de junio de 2017). "Introducción a la microbiota intestinal humana" . Revista bioquímica . 474 (11): 1823–1836. doi : 10.1042 / BCJ20160510 . ISSN 0264-6021 . PMC 5433529 . PMID 28512250 .
- ^ a b c Muñoz-Garach, Araceli; Díaz-Perdigones, Cristina; Tinahones, Francisco J. (diciembre de 2016). "Microbiota y diabetes mellitus tipo 2". Endocrinología y Nutrición (en español). 63 (10): 560–568. doi : 10.1016 / j.endonu.2016.07.008 . PMID 27633134 .
- ^ Blandino, G .; Inturri, R .; Lazzara, F .; Di Rosa, M .; Malaguarnera, L. (1 de noviembre de 2016). "Impacto de la microbiota intestinal en la diabetes mellitus" . Diabetes y metabolismo . 42 (5): 303–315. doi : 10.1016 / j.diabet.2016.04.004 . ISSN 1262-3636 . PMID 27179626 .
- ^ https://microbioma.it . Cite journal requiere
|journal=( ayuda );Falta o vacío|title=( ayuda ) - ^ Vandenplas, Y., Carnielli, VP, Ksiazyk, J., Luna, MS, Migacheva, N., Mosselmans, JM, ... y Wabitsch, M. (2020), Factores que afectan el desarrollo de la microbiota intestinal en la vida temprana. Nutrición, 78, 110812.
- ^ Korpela K, Helve O, Kolho KL, Saisto T, Skogberg K, Dikareva E, Stefanovic V, Salonen A, Andersson S, de Vos WM. El trasplante de microbiota fecal materna en bebés nacidos por cesárea restaura rápidamente el desarrollo microbiano intestinal normal: un estudio de prueba de concepto. Célula. 15 de octubre de 2020; 183 (2): 324-334.e5. doi: 10.1016 / j.cell.2020.08.047. Publicación electrónica 1 de octubre de 2020 PMID: 33007265.
- ^ Korpela, K., Salonen, A., Saxen, H., Nikkonen, A., Peltola, V., Jaakkola, T., ... y Kolho, KL (2020). Los antibióticos en la vida temprana se asocian con firmas específicas de la microbiota intestinal en una cohorte infantil longitudinal prospectiva. Investigación pediátrica, 1-6
- ^ Schei, K., Simpson, MR, Avershina, E., Rudi, K., Øien, T., Júlíusson, PB, ... y Ødegård, RA (2020). Microbiota bacteriana y micótica intestinal temprana y crecimiento infantil. Fronteras en pediatría, 8, 658
- ^ Korpela, K., Salonen, A., Virta, LJ, Kekkonen, RA, Forslund, K., Bork, P. y De Vos, WM (2016). El microbioma intestinal está relacionado con el uso de antibióticos de por vida en niños en edad preescolar finlandeses. Comunicaciones sobre la naturaleza, 7, 10410
- ^ Korpela, K., Salonen, A., Saxen, H., Nikkonen, A., Peltola, V., Jaakkola, T., ... y Kolho, KL (2020). Los antibióticos en la vida temprana se asocian con firmas específicas de la microbiota intestinal en una cohorte infantil longitudinal prospectiva. Investigación pediátrica, 1-6.
- ^ Seelbinder, B., Chen, J., Brunke, S., Vazquez-Uribe, R., Santhaman, R., Meyer, AC, ... y Panagiotou, G. (2020). Los antibióticos crean un cambio del mutualismo a la competencia en las comunidades intestinales humanas con un impacto más duradero en los hongos que en las bacterias. Microbioma, 8 (1), 1-20
- ^ Cabral, DJ, Penumutchu, S., Norris, C., Morones-Ramirez, JR y Belenky, P. (2018). La competencia microbiana entre Escherichia coli y Candida albicans revela un factor fungicida soluble. Célula microbiana, 5 (5), 249
- ^ Peleg, AY, Hogan, DA y Mylonakis, E. (2010). Interacciones bacteriano-fúngicas de importancia médica. Nature Reviews Microbiology, 8 (5), 340-349
- ^ Seelbinder, B., Chen, J., Brunke, S., Vazquez-Uribe, R., Santhaman, R., Meyer, AC, ... y Panagiotou, G. (2020). Los antibióticos crean un cambio del mutualismo a la competencia en las comunidades intestinales humanas con un impacto más duradero en los hongos que en las bacterias. Microbioma, 8 (1), 1-20
- ^ Sokol H, Leducq V, Aschard H, Pham HP, Jegou S, Landman C, Cohen D, Liguori G, Bourrier A, Nion-Larmurier I, Cosnes J, Seksik P, Langella P, Skurnik D, Richard ML, Beaugerie L Disbiosis de la microbiota fúngica en la EII. Gut 2017; 66: 1039–1048. doi: 10.1136 / gutjnl-2015-310746
- ^ Rebecka Ventin-Holmberg, Anja Eberl, Schahzad Saqib, Katri Korpela, Seppo Virtanen, Taina Sipponen, Anne Salonen, Päivi Saavalainen, Eija Nissilä, Perfiles bacterianos y fúngicos como marcadores de la respuesta al fármaco Infliximab en la enfermedad inflamatoria intestinal y la colitis de Crohn, Journal of , 2020 ;, jjaa252, https://doi.org/10.1093/ecco-jcc/jjaa252
- ^ El Hage, R., Hernandez-Sanabria, E., Calatayud Arroyo, M., Props, R. y Van de Wiele, T. (2019). El consorcio productor de propionato restaura la disbiosis inducida por antibióticos en un modelo dinámico in vitro del ecosistema microbiano intestinal humano. Fronteras en microbiología, 10, 1206.
- ^ Tian, X., Hellman, J., Horswill, AR, Crosby, HA, Francis, KP y Prakash, A. (2019). Los niveles elevados de propionato derivado del microbioma intestinal se asocian con una reducción de la inflamación pulmonar estéril y la inmunidad bacteriana en ratones. Fronteras en microbiología, 10, 159.
- ^ Li, Y., Faden, HS y Zhu, L. (2020). La respuesta de la microbiota intestinal a los cambios dietéticos en los dos primeros años de vida. Fronteras en farmacología, 11, 334.
- ^ Seelbinder, B., Chen, J., Brunke, S., Vazquez-Uribe, R., Santhaman, R., Meyer, AC, ... y Panagiotou, G. (2020). Los antibióticos crean un cambio del mutualismo a la competencia en las comunidades intestinales humanas con un impacto más duradero en los hongos que en las bacterias. Microbioma, 8 (1), 1-20
- ^ revista, Ewen. "El ADN microbiano en el cuerpo humano se puede utilizar para identificar individuos" . Consultado el 17 de mayo de 2015 .
- ^ Callaway, Ewen (2015). "Los microbiomas plantean problemas de privacidad". Naturaleza . 521 (7551): 136. Código Bibliográfico : 2015Natur.521..136C . doi : 10.1038 / 521136a . PMID 25971486 . S2CID 4393347 .
- ↑ Yong, Ed (11 de mayo de 2015). "¿Pueden usarse los microbios que dejas atrás para identificarte?" . National Geographic . Consultado el 17 de mayo de 2015 .
| Busque microbiota en Wiktionary, el diccionario gratuito. |
| Scholia tiene un perfil de tema para el microbioma . |