Acoplamiento (molecular)
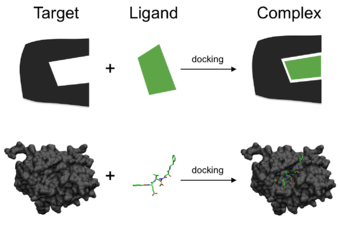
En el campo del modelado molecular , el acoplamiento es un método que predice la orientación preferida de una molécula a una segunda cuando un lingando y un objetivo se unen entre sí para formar un complejo estable . [1] El conocimiento de la orientación preferida, a su vez, puede usarse para predecir la fuerza de asociación o la afinidad de unión entre dos moléculas usando, por ejemplo, funciones de puntuación .
Las asociaciones entre moléculas biológicamente relevantes como proteínas , péptidos , ácidos nucleicos , carbohidratos y lípidos juegan un papel central en la transducción de señales . Además, la orientación relativa de los dos socios que interactúan puede afectar el tipo de señal producida (p. ej., agonismo frente a antagonismo ). Por lo tanto, el acoplamiento es útil para predecir tanto la fuerza como el tipo de señal producida.
El acoplamiento molecular es uno de los métodos más utilizados en el diseño de fármacos basado en la estructura , debido a su capacidad para predecir la conformación de unión de ligandos de moléculas pequeñas al sitio de unión objetivo adecuado . La caracterización del comportamiento de unión juega un papel importante en el diseño racional de fármacos , así como para dilucidar los procesos bioquímicos fundamentales. [2] [3]
Uno puede pensar en el acoplamiento molecular como un problema de "cerradura y llave" , en el que uno quiere encontrar la orientación relativa correcta de la "llave" que abrirá la "cerradura" (donde en la superficie de la cerradura está el orificio de la llave, en qué dirección girar la llave después de insertarla, etc.). Aquí, la proteína puede considerarse como el "candado" y el ligando como una "llave". El acoplamiento molecular se puede definir como un problema de optimización, que describiría la orientación de "mejor ajuste" de un ligando que se une a una proteína de interés particular. Sin embargo, dado que tanto el ligando como la proteína son flexibles, una analogía de "mano a mano" es más apropiada que "cerradura y llave" . [4] Durante el curso del proceso de acoplamiento, el ligando y la proteína ajustan su conformación para lograr un "mejor ajuste" general y este tipo de ajuste conformacional que da como resultado la unión general se denomina "ajuste inducido" . [5]
La investigación de acoplamiento molecular se centra en la simulación computacional del proceso de reconocimiento molecular . Su objetivo es lograr una conformación optimizada tanto para la proteína como para el ligando y una orientación relativa entre la proteína y el ligando de modo que se minimice la energía libre del sistema global.
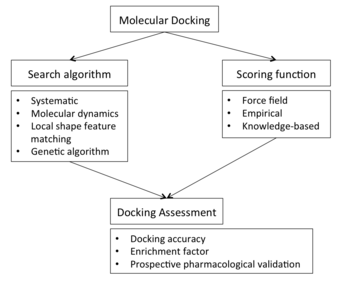
Dos enfoques son particularmente populares dentro de la comunidad de acoplamiento molecular. Un enfoque utiliza una técnica de emparejamiento que describe la proteína y el ligando como superficies complementarias. [6] [7] [8] El segundo enfoque simula el proceso de acoplamiento real en el que se calculan las energías de interacción ligando-proteína por pares. [9] Ambos enfoques tienen ventajas significativas, así como algunas limitaciones. Estos se describen a continuación.