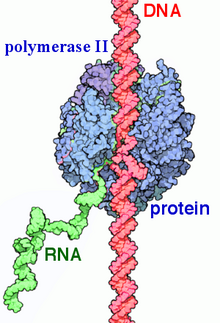
La ARN polimerasa II ( RNAP II y Pol II ) es un complejo multiproteico que transcribe el ADN en precursores del ARN mensajero (ARNm) y la mayoría del ARN nuclear pequeño (snRNA) y microARN . [1] [2] Es una de las tres enzimas RNAP que se encuentran en el núcleo de las células eucariotas . [3] Un complejo de 550 kDa de 12 subunidades, RNAP II es el tipo de ARN polimerasa más estudiado . Se requiere una amplia gama de factores de transcripción para que se una al gen corriente arribapromotores y comenzar la transcripción.
Descubrimiento [ editar ]
Los primeros estudios sugirieron un mínimo de dos RNAP: uno que sintetizaba ARNr en el nucleolo y otro que sintetizaba otro ARN en el nucleoplasma , parte del núcleo pero fuera del nucleolo. [5] En 1969, los experimentadores científicos Robert Roeder y William Rutter descubrieron definitivamente un RNAP adicional que era responsable de la transcripción de algún tipo de ARN en el nucleoplasma. El hallazgo se obtuvo mediante el uso de cromatografía de intercambio iónico a través de Sephadex recubierto con DEAE.rosario. La técnica separó las enzimas por el orden de las eluciones correspondientes, Ι, ΙΙ, ΙΙΙ, aumentando la concentración de sulfato de amonio. Las enzimas se nombraron según el orden de las eluciones, RNAP I , RNAP II, RNAP IΙI . [3] Este descubrimiento demostró que había una enzima adicional presente en el nucleoplasma, que permitió la diferenciación entre RNAP II y RNAP III.
Subunidades [ editar ]
La ARN polimerasa II del núcleo eucariota se purificó primero usando ensayos de transcripción. [7] La enzima purificada tiene típicamente de 10 a 12 subunidades (12 en humanos y levaduras) y es incapaz de reconocer un promotor específico. [8] Se conocen muchas interacciones subunidad-subunidad. [9]
- Subunidad RPB1 de la ARN polimerasa II dirigida por ADN : una enzima que en los seres humanos está codificada por el gen POLR2A y en la levadura está codificada por RPO21. RPB1 es la subunidad más grande de la ARN polimerasa II. Contiene un dominio carboxi terminal (CTD) compuesto por hasta 52 repeticiones de heptapéptidos (YSPTSPS) que son esenciales para la actividad de la polimerasa. [10] El CTD fue descubierto por primera vez en el laboratorio de CJ Ingles en la Universidad de Toronto y por JL Corden en la Universidad Johns Hopkins . En combinación con varias otras subunidades de la polimerasa, la subunidad RPB1 forma el dominio de unión al ADN de la polimerasa, un surco en el que la plantilla de ADN se transcribe en ARN. [11]Interactúa fuertemente con RPB8. [9]
- RPB2 ( POLR2B ): la segunda subunidad más grande que, en combinación con al menos otras dos subunidades de polimerasa, forma una estructura dentro de la polimerasa que mantiene contacto en el sitio activo de la enzima entre la plantilla de ADN y el ARN recién sintetizado. [12]
- RPB3 ( POLR2C ): la tercera subunidad más grande. Existe como un heterodímero con otra subunidad de polimerasa, POLR2J , formando un subconjunto central. RPB3 interactúa fuertemente con RPB1-5, 7, 10-12. [9]
- La subunidad B4 de la ARN polimerasa II (RPB4) , codificada por el gen POLR2D [13], es la cuarta subunidad más grande y puede tener una función protectora del estrés.
- RPB5 : en los seres humanos, está codificado por el gen POLR2E . Dos moléculas de esta subunidad están presentes en cada ARN polimerasa II. [14] RPB5 interactúa fuertemente con RPB1, RPB3 y RPB6. [9]
- RPB6 ( POLR2F ) : forma una estructura con al menos otras dos subunidades que estabilizan la polimerasa de transcripción en la plantilla de ADN. [15]
- RPB7 : codificado por POLR2G y puede desempeñar un papel en la regulación de la función de la polimerasa. [16] RPB7 interactúa fuertemente con RPB1 y RPB5. [9]
- RPB8 ( POLR2H ) : interactúa con las subunidades RPB1-3, 5 y 7. [9]
- RPB9 : el surco en el que se transcribe la plantilla de ADN en ARN está compuesto por RPB9 ( POLR2I ) y RPB1.
- RPB10 : el producto del gen POLR2L . Interactúa con RPB1-3 y 5, y fuertemente con RPB3. [9]
- RPB11 : la subunidad RPB11 se compone de tres subunidades en los seres humanos: POLR2J (RPB11-a), POLR2J2 (RPB11-b) y POLR2J3 [17] (RPB11-c).
- RPB12 - También interactúa con RPB3 RPB12 ( POLR2K ). [9]
Montaje [ editar ]
RPB3 participa en el ensamblaje de la ARN polimerasa II. [18] Un subcomplejo de RPB2 y RPB3 aparece poco después de la síntesis de subunidades. [18] Este complejo posteriormente interactúa con RPB1. [18] RPB3, RPB5 y RPB7 interactúan entre sí para formar homodímeros, y RPB3 y RPB5 juntos pueden ponerse en contacto con todas las demás subunidades de RPB, excepto RPB9. [9] Solo RPB1 se une fuertemente a RPB5. [9] La subunidad RPB1 también contacta con RPB7, RPB10 y de manera más débil pero más eficiente con RPB8. [9] Una vez que RPB1 ingresa al complejo, pueden ingresar otras subunidades como RPB5 y RPB7, donde RPB5 se une a RPB6 y RPB8 y RPB3 trae RPB10, RPB 11 y RPB12. [9]RPB4 y RPB9 pueden ingresar una vez que la mayor parte del complejo esté ensamblado. RPB4 forma un complejo con RPB7. [9]
Cinética [ editar ]
Las enzimas pueden catalizar hasta varios millones de reacciones por segundo. Las tasas de enzimas dependen de las condiciones de la solución y la concentración del sustrato. Como otras enzimas, POLR2 tiene una curva de saturación y una velocidad máxima ( V max ). Tiene una K m (concentración de sustrato requerida para la mitad de V máx ) y una k cat (el número de moléculas de sustrato manejadas por un sitio activo por segundo). La constante de especificidad viene dada por k cat / K m . El máximo teórico para la constante de especificidad es el límite de difusión de aproximadamente 10 8 a 10 9 ( M −1s −1 ), donde cada colisión de la enzima con su sustrato resulta en catálisis. En la levadura, la mutación en el dominio Trigger-Loop de la subunidad más grande puede cambiar la cinética de la enzima. [19]
La ARN polimerasa bacteriana, un pariente de la ARN polimerasa II, cambia entre los estados inactivado y activado mediante la translocación de ida y vuelta a lo largo del ADN. [20] Las concentraciones de [NTP] eq = 10 μM GTP, 10 μM UTP, 5 μM ATP y 2.5 μM CTP, producen una tasa de elongación media, número de recambio, de ~ 1 pb (NTP) -1 para RNAP bacteriano, un relativo de la ARN polimerasa II. [20]
La ARN polimerasa II sufre una extensa pausa cotranscripcional durante el alargamiento de la transcripción. [21] [22] Esta pausa es especialmente pronunciada en los nucleosomas y surge en parte debido a que la polimerasa entra en un estado de retroceso transcripcionalmente incompetente. [21] La duración de estas pausas varía de segundos a minutos o más, y la salida de las pausas de larga duración puede ser promovida por factores de alargamiento como TFIIS. [23] A su vez, la tasa de transcripción influye en si las histonas de los nucleosomas transcritos se expulsan de la cromatina o se reinsertan detrás de la polimerasa de transcripción. [24]
Alfa-Amanitina [ editar ]
La ARN polimerasa II es inhibida por la α-Amanitina [25] y otras amatoxinas . La α-amanitina es una sustancia muy venenosa que se encuentra en muchos hongos. [5] El veneno de hongos tiene diferentes efectos en cada una de las ARN polimerasas: I, II, III. RNAP I no responde por completo a la sustancia y funcionará normalmente, mientras que RNAP III tiene una sensibilidad moderada. Sin embargo, la toxina inhibe completamente el RNAP II. La alfa-amanitina inhibe la RNAP II mediante interacciones fuertes en las regiones "embudo", "hendidura" y " hélice α del puente" de la enzima de la subunidad RPB-1. [26]
Holoenzima [ editar ]
ARN polimerasa II holoenzima es una forma de eucariota ARN polimerasa II que se reclutó a los promotores de proteína de genes -coding en células vivas. [8] Consiste en ARN polimerasa II, un subconjunto de factores de transcripción generales y proteínas reguladoras conocidas como proteínas SRB.
Parte del ensamblaje de la holoenzima se denomina complejo de preiniciación , porque su ensamblaje tiene lugar en el promotor del gen antes del inicio de la transcripción . El complejo mediador actúa como puente entre la ARN polimerasa II y los factores de transcripción.
Control por estructura de cromatina [ editar ]
Este es un esquema de un mecanismo de ejemplo de las células de levadura por el cual la estructura de la cromatina y la modificación postraduccional de la histona ayudan a regular y registrar la transcripción de genes por la ARN polimerasa II.
Esta vía da ejemplos de regulación en estos puntos de transcripción:
- Preiniciación (promoción por Bre1, modificación de histonas)
- Iniciación (promoción por TFIIH, modificación de Pol II Y promoción por COMPASS, modificación de histonas)
- Alargamiento (promoción por Set2, modificación de histonas)
Esto se refiere a varias etapas del proceso como pasos regulatorios. No se ha comprobado que se utilicen para la regulación, pero es muy probable que lo sean.
Los promotores de elongación de ARN Pol II se pueden resumir en 3 clases.
- Factores afectados por la detención dependientes del fármaco / secuencia (diversas proteínas que interfieren)
- Factores orientados a la estructura de la cromatina (modificadores postranscripcionales de histonas, p. Ej., Histonas metiltransferasas)
- Factores de mejora de la catálisis de ARN Pol II (varias proteínas interferentes y cofactores Pol II; ver ARN polimerasa II).
Mecanismos de transcripción [ editar ]
- Orientada estructura de la cromatina factores:
(HMTs ( H iStone M acetato de T ransferases)):
COMPASS§ † - ( COM plex de P roteins AS sociated con S et1) - Metila la lisina 4 de la histona H3: Es responsable de la represión / silenciamiento de la transcripción. Una parte normal del crecimiento celular y la regulación de la transcripción dentro de RNAP II. [27] - Set2 - Metila la lisina 36 de la histona H3: Set2 participa en la regulación del alargamiento de la transcripción a través de su contacto directo con el CTD. [28]
(ejemplo interesante e irrelevante: Dot1 * ‡ - Metila la lisina 79 de la histona H3). - Bre1 - Ubiquinates (agrega ubiquitina a) lisina 123 de la histona H2B. Asociado con la preiniciación y que permite la unión de RNA Pol II.
CTD de la ARN polimerasa [ editar ]
El extremo C-terminal de RPB1 se adjunta para formar el dominio C-terminal (CTD). El dominio carboxi-terminal de la ARN polimerasa II consiste típicamente en hasta 52 repeticiones de la secuencia Tyr-Ser-Pro-Thr-Ser-Pro-Ser. [29] El dominio se extiende desde el núcleo de la enzima RNAPII hasta el canal de salida, esta ubicación es efectiva debido a sus inducciones de "reacciones de procesamiento de ARN, a través de interacciones directas o indirectas con componentes de la maquinaria de procesamiento de ARN". [30] El dominio CTD no existe en la ARN polimerasa I o la ARN polimerasa III. [3]La ARN polimerasa CTD se descubrió primero en el laboratorio de CJIngles en la Universidad de Toronto y también en el laboratorio de J Corden en la Universidad Johns Hopkins durante los procesos de secuenciación del ADN que codifica la subunidad RPB1 de la ARN polimerasa de levaduras y ratones, respectivamente. Otras proteínas a menudo se unen al dominio C-terminal de la ARN polimerasa para activar la actividad de la polimerasa. Es el dominio de la proteína que está involucrado en el inicio de la transcripción, la protección de la transcripción de ARN y la unión al espliceosoma para el empalme del ARN . [10]
Fosforilación del dominio CTD [ editar ]
La ARN polimerasa II existe en dos formas no fosforiladas y fosforiladas, IIA y IIO respectivamente. [5] [3] La transición entre las dos formas facilita diferentes funciones para la transcripción. La fosforilación de CTD es catalizada por uno de los seis factores de transcripción generales , TFIIH . TFIIH tiene dos propósitos: uno es desenrollar el ADN en el sitio de inicio de la transcripción y el otro es fosforilar. La forma polimerasa IIA se une al complejo de preiniciación, esto se sugiere porque IIA se une con mayor afinidad a la TBP ( proteína de unión a caja TATA ), la subunidad del factor de transcripción general TFIID., que la forma de polimerasa IIO. La forma polimerasa IIO facilita el alargamiento de la cadena de ARN. [5] El método para el inicio del alargamiento se realiza mediante la fosforilación de la serina en la posición 5 (Ser5), a través de TFIIH. El Ser5 recién fosforilado recluta enzimas para tapar el extremo 5 'del ARN recién sintetizado y los "factores de procesamiento 3' en los sitios poli (A) ". [30] Una vez que se fosforila la segunda serina, Ser2, se activa el alargamiento. Para terminar con el alargamiento, debe producirse la desfosforilación. Una vez que el dominio está completamente desfosforilado, la enzima RNAP II se "recicla" y cataliza el mismo proceso con otro sitio de iniciación. [30]
Reparación recombinacional acoplada a la transcripción [ editar ]
El daño oxidativo del ADN puede bloquear la transcripción de la ARN polimerasa II y causar roturas de la hebra. Se ha descrito un proceso de recombinación asociado a la transcripción con plantilla de ARN que puede proteger contra el daño del ADN. [31] Durante las etapas G1 / G0 del ciclo celular , las células exhiben ensamblaje de factores de recombinación homólogos en rupturas de doble cadena dentro de regiones transcritas activamente. Parece que la transcripción está acoplada a la reparación de roturas de doble hebra del ADN mediante recombinación homóloga con plantilla de ARN. Este proceso de reparación vuelve a unir de forma eficaz y precisa las roturas de doble hebra en genes que la ARN polimerasa II transcribe activamente.
Ver también [ editar ]
- Transcripción eucariota
- Modificación postranscripcional
- ARN polimerasa I
- Holoenzima ARN polimerasa II
- ARN polimerasa III
- Transcripción (genética)
Referencias [ editar ]
- ^ Kornberg RD (diciembre de 1999). "Control transcripcional eucariota". Tendencias en biología celular . 9 (12): M46–9. doi : 10.1016 / S0962-8924 (99) 01679-7 . PMID 10611681 .
- ^ Sims RJ, Mandal SS, Reinberg D (junio de 2004). "Aspectos destacados recientes de la transcripción mediada por ARN-polimerasa-II". Opinión actual en biología celular . 16 (3): 263–71. doi : 10.1016 / j.ceb.2004.04.004 . PMID 15145350 .
- ↑ a b c d Young, Richard A. (28 de noviembre de 2003). "ARN polimerasa II". Revisión anual de bioquímica . 60 (1): 689–715. doi : 10.1146 / annurev.bi.60.070191.003353 . PMID 1883205 .
- ^ Meyer PA, Ye P, Zhang M, Suh MH, Fu J (junio de 2006). "Phasing RNA polimerasa II utilizando átomos de Zn intrínsecamente unidos: un modelo estructural actualizado" . Estructura . 14 (6): 973–82. doi : 10.1016 / j.str.2006.04.003 . PMID 16765890 .
- ↑ a b c d 1942-, Weaver, Robert Franklin (1 de enero de 2012). Biología molecular . McGraw-Hill. ISBN 9780073525327. OCLC 789601172 .CS1 maint: numeric names: authors list (link)
- ^ Armache, Karim-Jean; Mitterweger, Simone; Meinhart, Anton; Cramer, Patrick (2019). "Estructuras de la ARN polimerasa II completa y su subcomplejo, Rpb4 / 7" (PDF) . Revista de Química Biológica . 280 (8): 7131–1734. doi : 10.2210 / pdb1wcm / pdb . PMID 15591044 .
- ^ Sawadogo M, Sentenac A (1990). "ARN polimerasa B (II) y factores de transcripción generales". Revisión anual de bioquímica . 59 : 711–54. doi : 10.1146 / annurev.bi.59.070190.003431 . PMID 2197989 .
- ↑ a b Myer VE, Young RA (octubre de 1998). "Holoenzimas y subcomplejos de ARN polimerasa II" . La Revista de Química Biológica . 273 (43): 27757–60. doi : 10.1074 / jbc.273.43.27757 . PMID 9774381 .
- ^ a b c d e f g h i j k l m Acker J, de Graaff M, Cheynel I, Khazak V, Kedinger C, Vigneron M (julio de 1997). "Interacciones entre las subunidades de la ARN polimerasa II humana" . La Revista de Química Biológica . 272 (27): 16815–21. doi : 10.1074 / jbc.272.27.16815 . PMID 9201987 .
- ↑ a b Brickey WJ, Greenleaf AL (junio de 1995). "Estudios funcionales del dominio de repetición carboxi-terminal de la ARN polimerasa II de Drosophila in vivo" . Genética . 140 (2): 599–613. PMC 1206638 . PMID 7498740 .
- ^ "Entrez Gen: POLR2A polimerasa (ARN) II (ADN dirigido) polipéptido A, 220kDa" .
- ^ "Gen Entrez: POLR2B polimerasa (ARN) II (ADN dirigido) polipéptido B, 140 kDa" .
- ^ Khazak V, Estojak J, Cho H, Majors J, Sonoda G, Testa JR, Golemis EA (abril de 1998). "Análisis de la interacción de la nueva ARN polimerasa II (pol II) subunidad hsRPB4 con su socio hsRPB7 y con pol II" . Biología Molecular y Celular . 18 (4): 1935–45. doi : 10.1128 / mcb.18.4.1935 . PMC 121423 . PMID 9528765 .
- ^ "Entrez Gen: POLR2E polimerasa (ARN) II (ADN dirigido) polipéptido E, 25kDa" .
- ^ "Entrez Gen: POLR2F polimerasa (ARN) II (ADN dirigido) polipéptido F" .
- ^ "Entrez Gen: POLR2G polimerasa (ARN) II (ADN dirigido) polipéptido G" .
- ^ "POLR2J3 polimerasa (ARN) II (ADN dirigido) polipéptido J3" .
- ↑ a b c Kolodziej PA, Young RA (septiembre de 1991). "Mutaciones en las tres subunidades más grandes de la ARN polimerasa II de levadura que afectan el ensamblaje de la enzima" . Biología Molecular y Celular . 11 (9): 4669–78. doi : 10.1128 / mcb.11.9.4669 . PMC 361357 . PMID 1715023 .
- ^ Kaplan CD, Jin H, Zhang IL, Belyanin A (12 de abril de 2012). "Disección de la función de bucle de activación de Pol II y control dependiente de la actividad de Pol II de la selección del sitio de inicio in vivo" . PLOS Genetics . 8 (4): e1002627. doi : 10.1371 / journal.pgen.1002627 . PMC 3325174 . PMID 22511879 .
- ↑ a b Abbondanzieri EA, Greenleaf WJ, Shaevitz JW, Landick R, Block SM (noviembre de 2005). "Observación directa del paso de pares de bases por ARN polimerasa" . Naturaleza . 438 (7067): 460–5. Código Bib : 2005Natur.438..460A . doi : 10.1038 / nature04268 . PMC 1356566 . PMID 16284617 .
- ^ a b Hodges, Courtney; Bintu, Lacramioara; Lubkowska, Lucyna; Kashlev, Mikhail; Bustamante, Carlos (31 de julio de 2009). "Las fluctuaciones nucleosomales gobiernan la dinámica de transcripción de la ARN polimerasa II" . Ciencia . 325 (5940): 626–628. Código Bibliográfico : 2009Sci ... 325..626H . doi : 10.1126 / science.1172926 . ISSN 1095-9203 . PMC 2775800 . PMID 19644123 .
- ^ Churchman, L. Stirling; Weissman, Jonathan S. (20 de enero de 2011). "La secuenciación de transcripción naciente visualiza la transcripción en resolución de nucleótidos" . Naturaleza . 469 (7330): 368–373. Código Bib : 2011Natur.469..368C . doi : 10.1038 / nature09652 . ISSN 1476-4687 . PMC 3880149 . PMID 21248844 .
- ^ Galburt, Eric A .; Grill, Stephan W .; Wiedmann, Anna; Lubkowska, Lucyna; Choy, Jason; Nogales, Eva; Kashlev, Mikhail; Bustamante, Carlos (12 de abril de 2007). "El retroceso determina la sensibilidad a la fuerza de RNAP II de una manera dependiente del factor". Naturaleza . 446 (7137): 820–823. Código Bibliográfico : 2007Natur.446..820G . doi : 10.1038 / nature05701 . ISSN 1476-4687 . PMID 17361130 . S2CID 4310108 .
- ^ Bintu, Lacramioara; Kopaczynska, Marta; Hodges, Courtney; Lubkowska, Lucyna; Kashlev, Mikhail; Bustamante, Carlos (13 de noviembre de 2011). "La tasa de elongación de la ARN polimerasa determina el destino de los nucleosomas transcritos" . Naturaleza Biología Molecular y Estructural . 18 (12): 1394-1399. doi : 10.1038 / nsmb.2164 . ISSN 1545-9985 . PMC 3279329 . PMID 22081017 .
- ^ CD de Kaplan, Larsson KM, Kornberg RD (junio de 2008). "El bucle de activación de la ARN polimerasa II funciona en la selección del sustrato y está directamente dirigido por alfa-amanitina" . Célula molecular . 30 (5): 547–56. doi : 10.1016 / j.molcel.2008.04.023 . PMC 2475549 . PMID 18538653 .
- ^ Gong, Xue Q .; Nedialkov, Yuri A .; Burton, Zachary F. (25 de junio de 2004). "La α-amanitina bloquea la translocación por la ARN polimerasa II humana" . Revista de Química Biológica . 279 (26): 27422–27427. doi : 10.1074 / jbc.M402163200 . ISSN 0021-9258 . PMID 15096519 .
- ^ Briggs, Scott D .; Bryk, Mary; Strahl, Brian D .; Cheung, Wang L .; Davie, Judith K .; Dent, Sharon YR; Winston, Fred; Allis, C. David (15 de diciembre de 2001). "La metilación de la lisina 4 de la histona H3 está mediada por Set1 y es necesaria para el crecimiento celular y el silenciamiento del ADNr en Saccharomyces cerevisiae" . Genes y desarrollo . 15 (24): 3286–3295. doi : 10.1101 / gad.940201 . ISSN 0890-9369 . PMC 312847 . PMID 11751634 .
- ^ Li, Bing; Howe, LeAnn; Anderson, Scott; Yates, John R .; Workman, Jerry L. (14 de marzo de 2003). "Las funciones de la histona metiltransferasa de Set2 a través del dominio carboxilo terminal fosforilado de la ARN polimerasa II" . Revista de Química Biológica . 278 (11): 8897–8903. doi : 10.1074 / jbc.M212134200 . ISSN 0021-9258 . PMID 12511561 .
- ^ Meinhart A, Cramer P (julio de 2004). "Reconocimiento del dominio carboxi-terminal de la ARN polimerasa II por factores de procesamiento 3'-ARN" . Naturaleza . 430 (6996): 223–6. Código Bibliográfico : 2004Natur.430..223M . doi : 10.1038 / nature02679 . PMID 15241417 . S2CID 4418258 .
- ^ a b c Egloff, Sylvain; Murphy, Shona (2008). "Rompiendo el código CTD de la ARN polimerasa II". Tendencias en Genética . 24 (6): 280–288. doi : 10.1016 / j.tig.2008.03.008 . PMID 18457900 .
- ^ Wei L, Levine AS, Lan L (2016). "Recombinación homóloga acoplada a la transcripción después del daño oxidativo". Reparación de ADN (Amst.) . 44 : 76–80. doi : 10.1016 / j.dnarep.2016.05.009 . PMID 27233112 .
Enlaces externos [ editar ]
- Más información en Berkeley National Lab
( Copia de Wayback Machine )
- ARN + polimerasa + II en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .