| Síndrome respiratorio agudo severo coronavirus 2 | |
|---|---|
 | |
| Micrografía electrónica de transmisión de viriones del SARS-CoV-2 con coronas visibles | |
 | |
| Modelo atómico científicamente preciso de la estructura externa del virión SARS-CoV-2. Cada "bola" es un átomo . [1] ● Azul: envoltura ● Turquesa: glicoproteína de pico (S) ● Rojo: proteínas de envoltura (E) ● Verde: proteínas de membrana (M) ● Naranja: glucosa | |
| Clasificación de virus | |
| (no clasificado): | Virus |
| Reino : | Riboviria |
| Reino: | Orthornavirae |
| Filo: | Pisuviricota |
| Clase: | Pisoniviricetes |
| Orden: | Nidovirales |
| Familia: | Coronaviridae |
| Género: | Betacoronavirus |
| Subgénero: | Sarbecovirus |
| Especies: | |
| Virus: | Síndrome respiratorio agudo severo coronavirus 2 |
| Variantes notables | |
| Sinónimos | |
| |
| Parte de una serie sobre el |
| Pandemia de COVID-19 |
|---|
 |
|
El síndrome respiratorio agudo severo coronavirus 2 ( SARS-CoV-2 ) [2] es el virus que causa COVID-19 (enfermedad por coronavirus 2019), la enfermedad respiratoria responsable de la pandemia de COVID-19 . [3] También conocido coloquialmente simplemente como el coronavirus , anteriormente se le conocía por su nombre provisional , nuevo coronavirus 2019 ( 2019-nCoV ), [4] [5] [6] [7] y también se le ha llamado coronavirus humano 2019. ( HCoV-19 o hCoV-19). [8] [9] [10] [11] Identificado por primera vez en la ciudad de Wuhan , Hubei , China, la Organización Mundial de la Salud declaró el brote una Emergencia de Salud Pública de Importancia Internacional el 30 de enero de 2020 y una pandemia el 11 de marzo de 2020 . [12] [13] SARS-CoV-2 es un virus de ARN monocatenario de sentido positivo [14] que es contagiosa en humanos. [15] Según lo descrito por los Institutos Nacionales de Salud de EE. UU . , Es el sucesor del SARS-CoV-1 , el virus que causó laBrote de SARS 2002-2004 . [dieciséis]
El SARS-CoV-2 es un virus de la especie coronavirus relacionado con el síndrome respiratorio agudo severo (SARSr-CoV). [2] Se cree que tiene orígenes zoonóticos y tiene una gran similitud genética con los coronavirus de los murciélagos, lo que sugiere que surgió de un virus transmitido por murciélagos . [9] [17] Se está investigando si el SARS-CoV-2 provino directamente de los murciélagos o indirectamente a través de hospedadores intermedios. [18] El virus muestra poca diversidad genética , lo que indica que es probable que el evento de propagación que introduce el SRAS-CoV-2 en los seres humanos haya ocurrido a finales de 2019. [19]
Los estudios epidemiológicos estiman que cada infección da como resultado un promedio de 2,39 a 3,44 nuevas cuando ningún miembro de la comunidad es inmune y no se toman medidas preventivas . [20] El virus se propaga principalmente entre las personas a través del contacto cercano y a través de aerosoles y gotitas respiratorias que se exhalan al hablar, respirar o exhalar, así como las que se producen al toser o estornudar. [21] [22] Entra principalmente en las células humanas uniéndose a una proteína de membrana que regula el sistema renina-angiotensina ( enzima convertidora de angiotensina 2 (ACE2)). [23] [24]
Terminología
Durante el brote inicial en Wuhan , China, se utilizaron varios nombres para el virus; algunos nombres utilizados por diferentes fuentes incluyen el "coronavirus" o "coronavirus de Wuhan". [25] [26] En enero de 2020, la Organización Mundial de la Salud recomendó "2019 nuevo coronavirus" (2019-nCov) [5] [27] como el nombre provisional del virus. Esto estaba de acuerdo con la guía de la OMS de 2015 [28] contra el uso de ubicaciones geográficas, especies animales o grupos de personas en los nombres de enfermedades y virus. [29] [30]
El 11 de febrero de 2020, el Comité Internacional de Taxonomía de Virus adoptó el nombre oficial de "coronavirus 2 del síndrome respiratorio agudo severo" (SARS-CoV-2). [31] Para evitar confusión con la enfermedad del SARS , la OMS a veces se refiere al SARS-CoV-2 como "el virus COVID-19" en las comunicaciones de salud pública [32] [33] y el nombre HCoV-19 se incluyó en algunas investigaciones artículos. [8] [9] [10]
Infección y transmisión
La transmisión de persona a persona del SARS-CoV-2 se confirmó el 20 de enero de 2020, durante la pandemia de COVID-19 . [15] [34] [35] [36] Inicialmente se asumió que la transmisión ocurría principalmente a través de gotitas respiratorias al toser y estornudar dentro de un rango de aproximadamente 1.8 metros (6 pies). [37] [38] Los experimentos de dispersión de luz láser sugieren que hablar es un modo adicional de transmisión [39] [40] y uno de gran alcance [41] y poco investigado [42] , en interiores, con poco flujo de aire. [43] [44]Otros estudios han sugerido que el virus también puede estar en el aire , y que los aerosoles pueden potencialmente transmitir el virus. [45] [46] [47] Durante la transmisión de persona a persona, se cree que entre 200 y 800 viriones infecciosos del SARS-CoV-2 inician una nueva infección. [48] [49] [50] Si se confirma, la transmisión por aerosoles tiene implicaciones de bioseguridad porque una de las principales preocupaciones asociadas con el riesgo de trabajar con virus emergentes en el laboratorio es la generación de aerosoles de diversas actividades de laboratorio que no son reconocibles de inmediato y pueden afectar a otros miembros del personal científico. [51]
El contacto indirecto a través de superficies contaminadas es otra posible causa de infección. [52] La investigación preliminar indica que el virus puede permanecer viable en plástico ( polipropileno ) y acero inoxidable ( AISI 304 ) hasta por tres días, pero no sobrevive en cartón por más de un día o en cobre por más de cuatro horas; [10] el virus es inactivado por el jabón, que desestabiliza su bicapa lipídica . [53] [54] También se ha encontrado ARN viral en muestras de heces y semen de personas infectadas. [55] [56]
El grado en que el virus es infeccioso durante el período de incubación es incierto, pero las investigaciones han indicado que la faringe alcanza la carga viral máxima aproximadamente cuatro días después de la infección [57] [58] o la primera semana de síntomas, y disminuye después. [59] La duración de la eliminación del ARN del SARS-CoV-2 es generalmente entre 3 y 46 días después del inicio de los síntomas. [60]
Un estudio realizado por un equipo de investigadores de la Universidad de Carolina del Norte encontró que la cavidad nasal es aparentemente el sitio inicial dominante para la infección con la subsiguiente siembra de virus mediada por aspiración en los pulmones en la patogénesis del SARS-CoV-2. [61] Encontraron que había un gradiente de infección desde alto en los cultivos de epitelio pulmonar proximal hacia bajo en el distal, con una infección focal en las células ciliadas y neumocitos tipo 2 en las vías respiratorias y las regiones alveolares, respectivamente. [61]
Los estudios han identificado una variedad de animales, como gatos, hurones, hámsteres, primates no humanos, visones, musarañas arborícolas, perros mapaches, murciélagos frugívoros y conejos, que son susceptibles y permisivos a la infección por SARS-CoV-2 [62]. [63] [64] Algunas instituciones han aconsejado a las personas infectadas con el SARS-CoV-2 que restrinjan el contacto con los animales. [65] [66]
Transmisión asintomática
El 1 de febrero de 2020, la Organización Mundial de la Salud (OMS) indicó que "la transmisión de casos asintomáticos probablemente no sea un factor importante de transmisión". [67] Un metanálisis encontró que el 17% de las infecciones son asintomáticas y que las personas asintomáticas tenían un 42% menos de probabilidades de transmitir el virus. [68]
Sin embargo, un modelo epidemiológico del comienzo del brote en China sugirió que "la diseminación presintomática puede ser típica entre las infecciones documentadas" y que las infecciones subclínicas pueden haber sido la fuente de la mayoría de las infecciones. [69] Eso puede explicar cómo de 217 a bordo de un crucero que atracó en Montevideo , solo 24 de 128 que dieron positivo por ARN viral mostraron síntomas. [70]De manera similar, un estudio de noventa y cuatro pacientes hospitalizados en enero y febrero de 2020 estimó que los pacientes arrojaron la mayor cantidad de virus dos o tres días antes de que aparezcan los síntomas y que "una proporción sustancial de transmisión probablemente ocurrió antes de los primeros síntomas en el caso índice ". [71]
Reinfección
Existe incertidumbre sobre la reinfección y la inmunidad a largo plazo. [72] No se sabe qué tan común es la reinfección, pero los informes han indicado que ocurre con una gravedad variable. [72]
El primer caso notificado de reinfección fue el de un hombre de 33 años de Hong Kong que dio positivo por primera vez el 26 de marzo de 2020, fue dado de alta el 15 de abril de 2020 después de dos pruebas negativas y volvió a dar positivo el 15 de agosto de 2020 (142 días después). , que fue confirmado por la secuenciación del genoma completo que muestra que los genomas virales entre los episodios pertenecen a diferentes clados . [73] Los hallazgos tenían las implicaciones de que la inmunidad colectiva puede no eliminar el virus si la reinfección no es una ocurrencia infrecuente y que las vacunas pueden no ser capaces de brindar protección de por vida contra el virus. [73]
Otro estudio de caso describió a un hombre de 25 años de Nevada que dio positivo por SARS-CoV-2 el 18 de abril de 2020 y el 5 de junio de 2020 (separados por dos pruebas negativas). Dado que los análisis genómicos mostraron diferencias genéticas significativas entre la variante del SARS-CoV-2 muestreada en esas dos fechas, los autores del estudio de caso determinaron que se trataba de una reinfección. [74] La segunda infección del hombre fue sintomáticamente más grave que la primera, pero se desconocen los mecanismos que podrían explicar esto. [74]
Embalse y origen

Las primeras infecciones conocidas por SARS-CoV-2 se descubrieron en Wuhan, China. [17] La fuente original de transmisión viral a los humanos sigue sin estar clara, al igual que si el virus se volvió patógeno antes o después del evento de desbordamiento . [19] [75] [9] Debido a que muchos de los primeros infectados eran trabajadores del mercado de mariscos de Huanan , [76] [77] se ha sugerido que el virus podría haberse originado en el mercado. [9] [78] Sin embargo, otra investigación indica que los visitantes pueden haber introducido el virus en el mercado, lo que luego facilitó la rápida expansión de las infecciones. [19] [79] Un informe convocado por la OMS en marzo de 2021 declaró que el derrame humano a través de un huésped animal intermedio era la explicación más probable, y que el derrame directo de los murciélagos era lo más probable. La introducción a través de la cadena de suministro de alimentos y el mercado de mariscos de Huanan se consideró otra explicación posible, pero menos probable. [80]
Para un virus adquirido recientemente a través de una transmisión entre especies, se espera una evolución rápida. [81] La tasa de mutación estimada de los primeros casos de SARS-CoV-2 fue de6,54 × 10 −4 por sitio por año. [80] Los coronavirus en general tienen una alta plasticidad genética , [82] pero la evolución viral del SARS-CoV-2 se ve frenada por la capacidad de corrección de pruebas de ARN de su maquinaria de replicación. [83]
La investigación sobre el reservorio natural del virus que causó el brote de SARS de 2002-2004 ha dado como resultado el descubrimiento de muchos coronavirus de murciélago similares al SARS , la mayoría originados en murciélagos de herradura . El análisis filogenético indica que las muestras tomadas de Rhinolophus sinicus muestran una semejanza del 80% con el SARS-CoV-2. [84] [85] [86] El análisis filogenético también indica que un virus de Rhinolophus affinis , recolectado en la provincia de Yunnan y designado como RaTG13 , tiene una semejanza del 96% con el SARS-CoV-2. [17] [87]La secuencia del virus RaTG13 es la secuencia conocida más cercana al SARS-CoV-2, [80] pero no es su ancestro directo. [88] También se identificaron otras secuencias estrechamente relacionadas en muestras de poblaciones locales de murciélagos. [89]

Los murciélagos se consideran el reservorio natural más probable del SARS-CoV-2. [80] [90] Las diferencias entre el coronavirus de murciélago y el SARS-CoV-2 sugieren que los seres humanos pueden haber sido infectados a través de un huésped intermedio; [78] aunque se desconoce la fuente de introducción en humanos. [91]
Aunque inicialmente se postuló el papel de los pangolines como hospedadores intermedios (un estudio publicado en julio de 2020 sugirió que los pangolines son hospedadores intermedios de coronavirus similares al SARS-CoV-2 [92] [93] ), los estudios posteriores no han corroborado su contribución al derrame. [80] Las pruebas en contra de esta hipótesis incluyen el hecho de que las muestras del virus del pangolín están demasiado distantes del SRAS-CoV-2: los aislamientos obtenidos de los pangolines incautados en Guangdong eran solo un 92% idénticos en secuencia al genoma del SRAS-CoV-2. Además, a pesar de las similitudes en algunos aminoácidos críticos, [94] las muestras de virus del pangolín exhiben una unión deficiente al receptor ACE2 humano. [95]
Filogenética y taxonomía
 Organización genómica del aislamiento Wuhan-Hu-1, la primera muestra secuenciada de SARS-CoV-2 | |
| Identificación del genoma NCBI | 86693 |
|---|---|
| Tamaño del genoma | 29,903 bases |
| Año de finalización | 2020 |
| Navegador del genoma ( UCSC ) | |
El SARS-CoV-2 pertenece a la amplia familia de virus conocidos como coronavirus . [26] Es un virus de ARN monocatenario de sentido positivo (+ ssRNA), con un solo segmento de ARN lineal. Los coronavirus infectan a los seres humanos, otros mamíferos y especies de aves, incluidos el ganado y los animales de compañía. [96] Los coronavirus humanos son capaces de causar enfermedades que van desde el resfriado común hasta enfermedades más graves como el síndrome respiratorio de Oriente Medio (MERS, tasa de mortalidad ~ 34%). El SARS-CoV-2 es el séptimo coronavirus conocido que infecta a las personas, después de 229E , NL63 , OC43 , HKU1 , MERS-CoVy el SARS-CoV original . [97]
Al igual que el coronavirus relacionado con el SARS implicado en el brote de SARS de 2003, el SARS-CoV-2 es miembro del subgénero Sarbecovirus ( linaje beta-CoV B). [98] [99] Los coronavirus también experimentan recombinación frecuente. [100] Su secuencia de ARN tiene aproximadamente 30.000 bases de longitud, [101] relativamente larga para un coronavirus (que a su vez lleva los genomas más grandes entre todas las familias de ARN) [102] Su genoma consiste casi en su totalidad en secuencias que codifican proteínas, un rasgo compartido con otros coronavirus. [100]
Una característica distintiva de SARS-CoV-2 es su incorporación de un polibásico sitio escindido por la furina , [94] , que parece ser un elemento importante mejora de su virulencia. [103] La furina proteasa reconoce la secuencia de péptidos canónicos R X [ R / K ] R ↓ X donde el sitio de escisión está indicado por una flecha hacia abajo y X es cualquier aminoácido . [104] [105] En el SARS-CoV-2, el sitio de reconocimiento está formado por la secuencia de nucleótidos de 12 codones incorporada CCT CGG CGG GCA que corresponde a la secuencia de aminoácidosP RR A . [106] Esta secuencia está aguas arriba de una arginina y serina que forma el sitio de escisión S1 / S2 ( P RR A R ↓ S ) de la proteína de pico. [107] Aunque estos sitios son una característica común de origen natural de otros virus dentro de la subfamilia Orthocoronavirinae, [106] aparece en algunos otros virus del género Beta-CoV , [108] y es único entre los miembros de su subgénero para tal sitio. [94] El SARS-CoV-1 es un ejemplo de un familiar conocido del SARS-CoV-2 que carece de FCS.
Los datos de la secuencia genética viral pueden proporcionar información crítica sobre si es probable que los virus separados por tiempo y espacio estén vinculados epidemiológicamente. [109] Con un número suficiente de genomas secuenciados , es posible reconstruir un árbol filogenético del historial de mutaciones de una familia de virus. Para el 12 de enero de 2020, cinco genomas de SARS-CoV-2 habían sido aislados en Wuhan y reportados por el Centro Chino para el Control y la Prevención de Enfermedades (CCDC) y otras instituciones; [101] [110] el número de genomas aumentó a 42 para el 30 de enero de 2020. [111] Un análisis filogenético de esas muestras mostró que estaban "altamente relacionados con un máximo de siete mutaciones en relación con unancestro común ", lo que implica que la primera infección humana ocurrió en noviembre o diciembre de 2019. [111] El examen de la topología del árbol filogenético al comienzo de la pandemia también encontró grandes similitudes entre los aislamientos humanos. [112] Al 7 de mayo de 2020 , [update]4.690 genomas del SRAS-CoV-2 muestreados en seis continentes estaban a disposición del público. [113] [se necesita aclaración ].
El 11 de febrero de 2020, el Comité Internacional de Taxonomía de Virus anunció que, de acuerdo con las reglas existentes que calculan las relaciones jerárquicas entre los coronavirus basándose en cinco secuencias conservadas de ácidos nucleicos, las diferencias entre lo que entonces se llamaba 2019-nCoV y el virus del SARS de 2003 brote fueron insuficientes para separar las especies virales . Por lo tanto, identificaron el 2019-nCoV como un virus del coronavirus relacionado con el síndrome respiratorio agudo severo . [114]
En julio de 2020, los científicos informaron que una variante de SARS-CoV-2 más infecciosa con la variante de proteína de pico G614 ha reemplazado a D614 como la forma dominante en la pandemia. [115] [116]
Los genomas y subgenomas de coronavirus codifican seis marcos de lectura abiertos (ORF). [117] En octubre de 2020, los investigadores descubrieron un posible gen superpuesto llamado ORF3d , en el genoma del SARS-CoV-2 . Se desconoce si la proteína producida por ORF3d tiene alguna función, pero provoca una fuerte respuesta inmune. ORF3d se ha identificado antes, en una variante de coronavirus que infecta a los pangolines . [118] [119]
Árbol filogenético
Un árbol filogenético basado en secuencias de genoma completo de SARS-CoV-2 y coronavirus relacionados es: [120] [121] [122]
| Coronavirus relacionado con el SARS-CoV-2 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
SARS-CoV-1 , 79% a SARS-COV-2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Variantes
Hay muchos miles de variantes de SARS-CoV-2, que se pueden agrupar en clados mucho más grandes . [128] Se han propuesto varias nomenclaturas de clados diferentes . Nextstrain divide las variantes en cinco clados (19A, 19B, 20A, 20B y 20C), mientras que GISAID las divide en siete (L, O, V, S, G, GH y GR). [129]
Varias variantes notables de SARS-CoV-2 surgieron a fines de 2020. La Organización Mundial de la Salud ha declarado actualmente cuatro variantes de preocupación , que son las siguientes: [130]
- Alpha : Lineage B.1.1.7 surgió en el Reino Unido en septiembre de 2020, con evidencia de una mayor transmisibilidad y virulencia. Las mutaciones notables incluyen N501Y y P681H .
- Se ha observado una mutación E484K en algunos viriones del linaje B.1.1.7 y varias agencias de salud pública también la siguen .
- Beta : El linaje B.1.351 surgió en Sudáfrica en mayo de 2020, con evidencia de una mayor transmisibilidad y cambios en la antigenicidad, y algunos funcionarios de salud pública alertaron sobre su impacto en la eficacia de algunas vacunas. Las mutaciones notables incluyen K417N , E484K y N501Y.
- Gamma : Lineage P.1 surgió en Brasil en noviembre de 2020, también con evidencia de mayor transmisibilidad y virulencia, junto con cambios en la antigenicidad. Se han planteado preocupaciones similares sobre la eficacia de la vacuna. Las mutaciones notables también incluyen K417N, E484K y N501Y.
- Delta : Lineage B.1.617.2 surgió en India en octubre de 2020. También hay evidencia de una mayor transmisibilidad y cambios en la antigenicidad.
Otras variantes notables incluyen otras 6 variantes designadas por la OMS que se encuentran bajo investigación y el Grupo 5 , que surgió entre los visones en Dinamarca y resultó en una campaña de eutanasia de visones que los dejó prácticamente extintos. [131]
Virología
Estructura

Cada virión del SARS-CoV-2 tiene un diámetro de 50 a 200 nanómetros (2,0 × 10 −6 a 7,9 × 10 −6 pulgadas ). [77] Al igual que otros coronavirus, el SARS-CoV-2 tiene cuatro proteínas estructurales, conocidas como proteínas S ( pico ), E ( envoltura ), M ( membrana ) y N ( nucleocápside ); la proteína N contiene el genoma del ARN, y las proteínas S, E y M juntas crean la envoltura viral . [132] Las proteínas del coronavirus S son glicoproteínas que se dividen en dos partes funcionales (S1 y S2). [96]En el SARS-CoV-2, la proteína de pico, que se ha obtenido a nivel atómico mediante microscopía electrónica criogénica , [133] [134] es la proteína responsable de permitir que el virus se adhiera y se fusione con la membrana de una célula huésped. ; [132] específicamente, su subunidad S1 cataliza la unión, la fusión de la subunidad S2. [135]
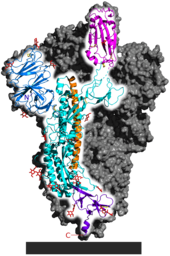
Genoma
El SARS-CoV-2 tiene un genoma de ARN monocatenario lineal, de sentido positivo , de aproximadamente 30.000 bases de largo. [96] Su genoma tiene un sesgo contra los nucleótidos de citosina (C) y guanina (G) como otros coronavirus. [136] El genoma tiene la composición más alta de U (32,2%), seguido de A (29,9%), y una composición similar de G (19,6%) y C (18,3%). [137] El sesgo de nucleótidos surge de la mutación de guaninas y citosinas en adenosinas y uracilos , respectivamente. [138] La mutación de dinucleótidos CGse cree que surge para evitar el mecanismo de defensa de las células relacionado con la proteína antiviral del dedo de zinc , [139] y para disminuir la energía para desvincular el genoma durante la replicación y traducción ( par de bases de adenosina y uracilo a través de dos enlaces de hidrógeno , citosina y guanina a través de tres) . [138] El agotamiento de dinucleótidos CG en su genoma ha llevado al virus a tener un sesgo notable en el uso de codones . Por ejemplo, los seis codones diferentes de la arginina tienen un uso de codón sinónimo relativo de AGA (2.67), CGU (1.46), AGG (.81), CGC (.58), CGA (.29) y CGG (.19). [137]Se observa una tendencia de sesgo de uso de codones similar en otros coronavirus relacionados con el SARS. [140]
Ciclo de replicación
Las infecciones por virus comienzan cuando las partículas virales se unen a los receptores celulares de la superficie del huésped. [141] Los experimentos de modelado de proteínas en la proteína de pico del virus pronto sugirieron que el SARS-CoV-2 tiene suficiente afinidad por el receptor de la enzima convertidora de angiotensina 2 (ACE2) en las células humanas para utilizarlas como mecanismo de entrada celular . [142] Para el 22 de enero de 2020, un grupo de China que trabajaba con el genoma completo del virus y un grupo de los Estados Unidos que utilizaba métodos de genética inversa demostraron de forma independiente y experimental que el ACE2 podía actuar como receptor del SARS-CoV-2. [17] [143] [144] [145]Los estudios han demostrado que el SARS-CoV-2 tiene una mayor afinidad con el ACE2 humano que el virus del SARS original. [133] [146] El SARS-CoV-2 también puede utilizar basigin para ayudar en la entrada de la celda. [147]
El cebado inicial de la proteína de pico por la proteasa transmembrana, serina 2 (TMPRSS2) es esencial para la entrada de SARS-CoV-2. [23] La proteína del huésped neuropilina 1 (NRP1) puede ayudar al virus en la entrada de la célula huésped utilizando ACE2. [148] Después de que un virión del SARS-CoV-2 se adhiere a una célula diana, el TMPRSS2 de la célula abre la proteína espiga del virus, exponiendo un péptido de fusión en la subunidad S2 y el receptor ACE2 del huésped. [135] Después de la fusión, se forma un endosoma alrededor del virión, que lo separa del resto de la célula huésped. El virión se escapa cuando el pH del endosoma desciende o cuando la catepsina , una cisteína del huéspedproteasa, lo escinde. [135] El virión luego libera ARN en la célula y obliga a la célula a producir y diseminar copias del virus , que infectan más células. [149]
El SARS-CoV-2 produce al menos tres factores de virulencia que promueven la eliminación de nuevos viriones de las células huésped e inhiben la respuesta inmunitaria . [132] Si incluyen la regulación a la baja de ACE2, como se ve en coronavirus similares, sigue bajo investigación (hasta mayo de 2020). [150]
Epidemiología
Sobre la base de la baja variabilidad exhibida entre las secuencias genómicas conocidas del SARS-CoV-2 , las autoridades sanitarias probablemente detectaron el virus a las pocas semanas de su aparición entre la población humana a fines de 2019. [19] [151] Del grupo inicial de casos, dos- Se descubrió que tercios tenían un vínculo con el mercado húmedo, pero el primer caso conocido con síntomas es de principios de diciembre de 2019 y carece de conexiones visibles con el grupo posterior. [80] Ha habido informes no confirmados en los periódicos de casos anteriores. [152] [153] El análisis del reloj molecular sugiere que es probable que el caso índice se haya infectado con el virus entre mediados de octubre y mediados de noviembre de 2019. [153] [154]Posteriormente, el virus se propagó a todas las provincias de China y a más de 150 países de todo el mundo. [155] Se ha confirmado la transmisión del virus de persona a persona en todas estas regiones. [156] El 30 de enero de 2020, el SARS-CoV-2 fue designado Emergencia de Salud Pública de Importancia Internacional por la OMS, [157] [12] y el 11 de marzo de 2020 la OMS lo declaró pandemia . [13] [158]
Las pruebas retrospectivas recopiladas dentro del sistema de vigilancia chino no revelaron indicios claros de una circulación sustancial no reconocida de SARS-CoV-2 en Wuhan durante la última parte de 2019. [80]
Un metaanálisis de noviembre de 2020 estimó el número de reproducción básico () del virus entre 2,39 y 3,44. [20] Esto significa que se espera que cada infección por el virus resulte en 2,39 a 3,44 nuevas infecciones cuando ningún miembro de la comunidad es inmune y no se toman medidas preventivas . El número de reproducción puede ser mayor en condiciones densamente pobladas como las que se encuentran en los cruceros . [159] Se pueden emplear muchas formas de esfuerzos preventivos en circunstancias específicas para reducir la propagación del virus. [117]
Ha habido alrededor de 96.000 casos confirmados de infección en China continental. [155] Si bien la proporción de infecciones que resultan en casos confirmados o progresan a una enfermedad diagnosticable sigue sin estar clara, [160] un modelo matemático estimó que 75,815 personas se infectaron el 25 de enero de 2020 solo en Wuhan, en un momento en que el número de casos confirmados en todo el mundo fue de solo 2.015. [161] Antes del 24 de febrero de 2020, más del 95% de todas las muertes por COVID-19 en todo el mundo habían ocurrido en la provincia de Hubei , donde se encuentra Wuhan. [162] [163] Al 1 de agosto de 2021, el porcentaje había disminuido al 0,076%. [155]
Al 1 de agosto de 2021, había un total de 198.182.570 casos confirmados de infección por SARS-CoV-2 en la pandemia en curso. [155] El número total de muertes atribuidas al virus es 4.222.338. [155]
Referencias
- ↑ Solodovnikov, Alexey; Arkhipova, Valeria (29 de julio de 2021). "Достоверно красиво: как мы сделали 3D-модель SARS-CoV-2" [Realmente hermoso: cómo hicimos el modelo 3D del SARS-CoV-2] (en ruso). N + 1 . Archivado desde el original el 30 de julio de 2021 . Consultado el 30 de julio de 2021 .
- ^ Zimmer C (26 de febrero de 2021). "La vida secreta de un coronavirus: una burbuja de genes aceitosa de 100 nanómetros de ancho ha matado a más de dos millones de personas y ha remodelado el mundo. Los científicos no saben muy bien qué hacer con ella" . Archivado desde el original el 27 de febrero de 2021 . Consultado el 28 de febrero de 2021 .
- ^ Definiciones de casos de vigilancia para la infección humana con el nuevo coronavirus (nCoV): guía provisional v1, enero de 2020 (Informe). Organización Mundial de la Salud. Enero de 2020. hdl : 10665/330376 . WHO / 2019-nCoV / Surveillance / v2020.1.
- ^ a b "Profesionales de la salud: respuestas y preguntas frecuentes" . Centros de Estados Unidos para el Control y la Prevención de Enfermedades (CDC) . 11 de febrero de 2020. Archivado desde el original el 14 de febrero de 2020 . Consultado el 15 de febrero de 2020 .
- ^ "Acerca del nuevo coronavirus (2019-nCoV)" . Centros de Estados Unidos para el Control y la Prevención de Enfermedades (CDC) . 11 de febrero de 2020. Archivado desde el original el 11 de febrero de 2020 . Consultado el 25 de febrero de 2020 .
- ^ Harmon A (4 de marzo de 2020). "Hablamos con seis estadounidenses con coronavirus" . The New York Times . Archivado desde el original el 13 de marzo de 2020 . Consultado el 16 de marzo de 2020 .
- ^ a b Wong G, Bi YH, Wang QH, Chen XW, Zhang ZG, Yao YG (mayo de 2020). "Orígenes zoonóticos del coronavirus humano 2019 (HCoV-19 / SARS-CoV-2): ¿por qué es importante este trabajo?" . Investigación Zoológica . 41 (3): 213–219. doi : 10.24272 / j.issn.2095-8137.2020.031 . PMC 7231470 . PMID 32314559 .
- ^ a b c d e Andersen KG, Rambaut A, Lipkin WI, Holmes EC, Garry RF (abril de 2020). "El origen proximal del SARS-CoV-2" . Medicina de la naturaleza . 26 (4): 450–452. doi : 10.1038 / s41591-020-0820-9 . PMC 7095063 . PMID 32284615 .
- ^ a b c van Doremalen N, Bushmaker T, Morris DH, Holbrook MG, Gamble A, Williamson BN, et al. (Abril de 2020). "Estabilidad de aerosol y superficie de SARS-CoV-2 en comparación con SARS-CoV-1" . La Revista de Medicina de Nueva Inglaterra . 382 (16): 1564-1567. doi : 10.1056 / NEJMc2004973 . PMC 7121658 . PMID 32182409 .
- ^ "Base de datos hCoV-19" . China National GeneBank. Archivado desde el original el 17 de junio de 2020 . Consultado el 2 de junio de 2020 .
- ^ a b "Declaración sobre la segunda reunión del Comité de Emergencia del Reglamento Sanitario Internacional (2005) sobre el brote del nuevo coronavirus (2019-nCoV)" . Organización Mundial de la Salud (OMS) (Comunicado de prensa). 30 de enero de 2020. Archivado desde el original el 31 de enero de 2020 . Consultado el 30 de enero de 2020 .
- ^ a b "Palabras de apertura del Director General de la OMS en la rueda de prensa sobre COVID-19 - 11 de marzo de 2020" . Organización Mundial de la Salud (OMS) (Comunicado de prensa). 11 de marzo de 2020. Archivado desde el original el 11 de marzo de 2020 . Consultado el 12 de marzo de 2020 .
- ^ Machhi J, Herskovitz J, Senan AM, Dutta D, Nath B, Oleynikov MD, et al. (Septiembre de 2020). "La historia natural, la patobiología y las manifestaciones clínicas de las infecciones por SARS-CoV-2" . Revista de farmacología neuroinmune . 15 (3): 359–386. doi : 10.1007 / s11481-020-09944-5 . PMC 7373339 . PMID 32696264 .
- ^ a b Chan JF, Yuan S, Kok KH, To KK, Chu H, Yang J, et al. (Febrero de 2020). "Un grupo familiar de neumonía asociado con el nuevo coronavirus de 2019 que indica la transmisión de persona a persona: un estudio de un grupo familiar" . Lancet . 395 (10223): 514–523. doi : 10.1016 / S0140-6736 (20) 30154-9 . PMC 7159286 . PMID 31986261 .
- ^ "Nuevo coronavirus estable durante horas en superficies" . Institutos Nacionales de Salud (NIH) . NIH.gov. 17 de marzo de 2020. Archivado desde el original el 23 de marzo de 2020 . Consultado el 4 de mayo de 2020 .
- ^ a b c d Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, et al. (Marzo de 2020). "Un brote de neumonía asociado a un nuevo coronavirus de probable origen murciélago" . Naturaleza . 579 (7798): 270–273. Código Bib : 2020Natur.579..270Z . doi : 10.1038 / s41586-020-2012-7 . PMC 7095418 . PMID 32015507 .
- ^ Nuevo coronavirus (2019-nCoV): informe de situación, 22 (Informe). Organización Mundial de la Salud . 11 de febrero de 2020. hdl : 10665/330991 .
- ^ a b c d Cohen J (enero de 2020). "Es posible que el mercado de productos del mar de Wuhan no sea una fuente de propagación del nuevo virus a nivel mundial". Ciencia . doi : 10.1126 / science.abb0611 .
- ^ a b Billah MA, Miah MM, Khan MN (11 de noviembre de 2020). "Número reproductivo de coronavirus: una revisión sistemática y un metanálisis basado en evidencia de nivel global" . PLOS ONE . 15 (11): e0242128. Código bibliográfico : 2020PLoSO..1542128B . doi : 10.1371 / journal.pone.0242128 . PMC 7657547 . PMID 33175914 .
- ^ " Cómo se propaga el coronavirus, archivado el 3 de abril de 2020 en la Wayback Machine ", Centros para el Control y la Prevención de Enfermedades, consultado el 14 de mayo de 2021.
- ^ " Enfermedad por coronavirus (COVID-19): ¿Cómo se transmite? Archivado el 15 de octubre de 2020 en Wayback Machine ", Organización Mundial de la Salud
- ^ a b Hoffmann M, Kleine-Weber H, Schroeder S, Krüger N, Herrler T, Erichsen S, et al. (Abril de 2020). "La entrada de células SARS-CoV-2 depende de ACE2 y TMPRSS2 y está bloqueada por un inhibidor de proteasa clínicamente probado" . Celular . 181 (2): 271–280.e8. doi : 10.1016 / j.cell.2020.02.052 . PMC 7102627 . PMID 32142651 .
- ^ Zhao, Peng; Praissman, Jeremy L .; Grant, Oliver C .; Cai, Yongfei; Xiao, Tianshu; Rosenbalm, Katelyn E .; Aoki, Kazuhiro; Kellman, Benjamin P .; Bridger, Robert; Barouch, Dan H .; Brindley, Melinda A .; Lewis, Nathan E .; Tiemeyer, Michael; Chen, Bing; Woods, Robert J .; Wells, Lance (octubre de 2020). "Interacciones de receptor de virus de pico de SARS-CoV-2 glicosilado y receptor ACE2 humano" . Anfitrión celular y microbio . 28 (4): 586–601.e6. doi : 10.1016 / j.chom.2020.08.004 . PMC 7443692 . PMID 32841605 .
- ^ Huang P (22 de enero de 2020). "¿Cómo se compara el coronavirus de Wuhan con el MERS, el SARS y el resfriado común?" . NPR . Archivado desde el original el 2 de febrero de 2020 . Consultado el 3 de febrero de 2020 .
- ^ a b Fox D (enero de 2020). "Lo que necesita saber sobre el nuevo coronavirus". Naturaleza . doi : 10.1038 / d41586-020-00209-y . PMID 33483684 .
- ^ Organización Mundial de la Salud (30 de enero de 2020). Nuevo coronavirus (2019-nCoV): informe de situación, 10 (Informe). Organización Mundial de la Salud . hdl : 10665/330775 .
- ^ "Mejores prácticas de la Organización Mundial de la salud para la denominación de nuevas enfermedades infecciosas humanas" (PDF) . QUIEN . Mayo de 2015. Archivado (PDF) desde el original el 12 de febrero de 2020.
- ^ "Nuevo coronavirus llamado 'Covid-19': QUIÉN" . HOYen línea. Archivado desde el original el 21 de marzo de 2020 . Consultado el 11 de febrero de 2020 .
- ^ "El coronavirus propaga el racismo contra y entre los chinos étnicos" . The Economist . 17 de febrero de 2020. Archivado desde el original el 17 de febrero de 2020 . Consultado el 17 de febrero de 2020 .
- ^ "Nombrar la enfermedad por coronavirus (COVID-2019) y el virus que la causa" . Organización Mundial de la Salud. Archivado desde el original el 28 de febrero de 2020 . Consultado el 14 de diciembre de 2020 .
El 11 de febrero de 2020, ICTV anunció “coronavirus 2 del síndrome respiratorio agudo severo (SARS-CoV-2)” como el nombre del nuevo virus. Se eligió este nombre porque el virus está genéticamente relacionado con el coronavirus responsable del brote de SARS de 2003. Si bien están relacionados, los dos virus son diferentes.
- ^ Hui M (18 de marzo de 2020). "¿Por qué la OMS no llama al coronavirus por su nombre, SARS-CoV-2?" . Cuarzo . Archivado desde el original el 25 de marzo de 2020 . Consultado el 26 de marzo de 2020 .
- ^ "Nombrar la enfermedad por coronavirus (COVID-2019) y el virus que la causa" . Organización Mundial de la Salud. Archivado desde el original el 28 de febrero de 2020 . Consultado el 14 de diciembre de 2020 .
Desde una perspectiva de comunicación de riesgos, el uso del nombre SARS puede tener consecuencias no deseadas en términos de crear un miedo innecesario para algunas poblaciones.
... Por esa razón y otras, la OMS ha comenzado a referirse al virus como "el virus responsable de COVID-19" o "el virus COVID-19" al comunicarse con el público. Ninguna de estas designaciones [sic] pretende reemplazar el nombre oficial del virus según lo acordado por ICTV.
- ^ Li JY, You Z, Wang Q, Zhou ZJ, Qiu Y, Luo R, Ge XY (marzo de 2020). "La epidemia de neumonía de 2019-nuevo-coronavirus (2019-nCoV) y conocimientos para las enfermedades infecciosas emergentes en el futuro" . Microbios e infección . 22 (2): 80–85. doi : 10.1016 / j.micinf.2020.02.002 . PMC 7079563 . PMID 32087334 .
- ^ Kessler G (17 de abril de 2020). "La falsa afirmación de Trump de que la OMS dijo que el coronavirus 'no era transmisible ' " . The Washington Post . Archivado desde el original el 17 de abril de 2020 . Consultado el 17 de abril de 2020 .
- ^ Kuo L (21 de enero de 2020). "China confirma la transmisión de coronavirus de persona a persona" . The Guardian . Archivado desde el original el 22 de marzo de 2020 . Consultado el 18 de abril de 2020 .
- ^ "Cómo se propaga COVID-19" . Centros para el Control y la Prevención de Enfermedades de EE. UU . (CDC) . 27 de enero de 2020. Archivado desde el original el 28 de enero de 2020 . Consultado el 29 de enero de 2020 .
- ^ Edwards E (25 de enero de 2020). "¿Cómo se propaga el coronavirus?" . NBC News . Archivado desde el original el 28 de enero de 2020 . Consultado el 13 de marzo de 2020 .
- ^ Anfinrud P, Stadnytskyi V, Bax CE, Bax A (mayo de 2020). "Visualización de gotas de fluido oral generadas por el habla con dispersión de luz láser" . La Revista de Medicina de Nueva Inglaterra . 382 (21): 2061-2063. doi : 10.1056 / NEJMc2007800 . PMC 7179962 . PMID 32294341 .
- ^ Stadnytskyi V, Bax CE, Bax A, Anfinrud P (junio de 2020). "La vida en el aire de las pequeñas gotas de voz y su importancia potencial en la transmisión del SARS-CoV-2" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 117 (22): 11875-11877. doi : 10.1073 / pnas.2006874117 . PMC 7275719 . PMID 32404416 .
- ^ Klompas M, Baker MA, Rhee C (agosto de 2020). "Transmisión aérea de SARS-CoV-2: consideraciones teóricas y evidencia disponible" . JAMA . 324 (5): 441–442. doi : 10.1001 / jama.2020.12458 . PMID 32749495 . S2CID 220500293 .
Los investigadores han demostrado que hablar y toser producen una mezcla de gotitas y aerosoles en una variedad de tamaños, que estas secreciones pueden viajar juntas hasta 27 pies, que es factible que el SARS-CoV-2 permanezca suspendido en el aire y viable durante horas, que el ARN del SARS-CoV-2 se puede recuperar de las muestras de aire en los hospitales y que la mala ventilación prolonga la cantidad de tiempo que los aerosoles permanecen en el aire.
- ^ Asadi S, Bouvier N, Wexler AS, Ristenpart WD (2 de junio de 2020). "La pandemia del coronavirus y los aerosoles: ¿COVID-19 se transmite a través de partículas espiratorias?" . Ciencia y tecnología de aerosoles . 54 (6): 635–638. Código bibliográfico : 2020AerST..54..635A . doi : 10.1080 / 02786826.2020.1749229 . PMC 7157964 . PMID 32308568 .
No está claro cuál de estos mecanismos juega un papel clave en la transmisión de COVID-19. Gran parte de la investigación sobre enfermedades transmitidas por el aire antes de la pandemia actual se ha centrado en eventos espiratorios 'violentos' como estornudos y tos.
- ^ Rettner R (21 de enero de 2021). "Hablar es peor que toser para esparcir COVID-19 en interiores" . livescience.com . Consultado el 23 de enero de 2021 .
En un escenario modelado, los investigadores encontraron que después de una tos breve, la cantidad de partículas infecciosas en el aire caería rápidamente después de 1 a 7 minutos; por el contrario, después de hablar durante 30 segundos, solo después de 30 minutos el número de partículas infecciosas descendería a niveles similares; y una gran cantidad de partículas todavía se suspendieron después de una hora. En otras palabras, una dosis de partículas de virus capaces de causar una infección permanecería en el aire mucho más tiempo después del habla que una tos. (En este escenario modelado, se admitió la misma cantidad de gotas durante una tos de 0,5 segundos que durante el transcurso de 30 segundos de habla).
- ↑ de Oliveira PM, Mesquita LC, Gkantonas S, Giusti A, Mastorakos E (enero de 2021). "Evolución de la pulverización y el aerosol de las emisiones respiratorias: estimaciones teóricas para comprender la transmisión viral" . Actas de la Royal Society A: Ciencias Matemáticas, Físicas e Ingeniería . 477 (2245): 20200584. Bibcode : 2021RSPSA.47700584D . doi : 10.1098 / rspa.2020.0584 . PMC 7897643 . PMID 33633490 . S2CID 231643585 .
- ^ Mandavilli A (4 de julio de 2020). "239 expertos con una gran afirmación: el coronavirus se transmite por el aire: la OMS se ha resistido a la creciente evidencia de que las partículas virales que flotan en el interior son infecciosas, dicen algunos científicos. La agencia sostiene que la investigación aún no es concluyente" . The New York Times . Archivado desde el original el 17 de noviembre de 2020 . Consultado el 5 de julio de 2020 .
- ^ Tufekci Z (30 de julio de 2020). "Tenemos que hablar de ventilación" . El Atlántico . Archivado desde el original el 17 de noviembre de 2020 . Consultado el 8 de septiembre de 2020 .
- ^ Lewis D (julio de 2020). "La creciente evidencia sugiere que el coronavirus se transmite por el aire, pero los consejos de salud no se han puesto al día" . Naturaleza . 583 (7817): 510–513. Código Bibliográfico : 2020Natur.583..510L . doi : 10.1038 / d41586-020-02058-1 . PMID 32647382 . S2CID 220470431 .
- ^ Popa A, Genger JW, Nicholson MD, Penz T, Schmid D, Aberle SW, et al. (Diciembre de 2020). "La epidemiología genómica de los eventos de superpropagación en Austria revela la dinámica mutacional y las propiedades de transmisión del SARS-CoV-2" . Medicina traslacional de la ciencia . 12 (573): eabe2555. doi : 10.1126 / scitranslmed.abe2555 . PMC 7857414 . PMID 33229462 .
- ^ Schröder, Imke (11 de mayo de 2020). "COVID-19: una perspectiva de evaluación de riesgos" . Revista de salud y seguridad química . 27 (3): 160–169. doi : 10.1021 / acs.chas.0c00035 . ISSN 1871-5532 . PMC 7216769 .
- ^ Watanabe, Toru; Bartrand, Timothy A .; Weir, Mark H .; Omura, Tatsuo; Haas, Charles N. (julio de 2010). "Desarrollo de un modelo de respuesta a dosis para el coronavirus del SARS" . Análisis de riesgo . 30 (7): 1129-1138. doi : 10.1111 / j.1539-6924.2010.01427.x . ISSN 0272-4332 . PMC 7169223 . PMID 20497390 .
- ^ Artika, I. Hecho; Ma'roef, Chairin Nisa (mayo de 2017). "Bioseguridad de laboratorio para el manejo de virus emergentes". Revista Asia Pacífico de Biomedicina Tropical . 7 (5): 483–491. doi : 10.1016 / j.apjtb.2017.01.020 .
- ^ "Preparar su lugar de trabajo para COVID-19" (PDF) . Organización Mundial de la Salud . 27 de febrero de 2020. Archivado (PDF) desde el original el 2 de marzo de 2020 . Consultado el 3 de marzo de 2020 .
- ^ Yong E (20 de marzo de 2020). "Por qué el coronavirus ha tenido tanto éxito" . El Atlántico . Archivado desde el original el 20 de marzo de 2020 . Consultado el 20 de marzo de 2020 .
- ^ Gibbens S (18 de marzo de 2020). "Por qué es preferible el jabón a la lejía en la lucha contra el coronavirus" . National Geographic . Archivado desde el original el 2 de abril de 2020 . Consultado el 2 de abril de 2020 .
- ^ Holshue ML, DeBolt C, Lindquist S, Lofy KH, Wiesman J, Bruce H, et al. (Marzo de 2020). "Primer caso de nuevo coronavirus 2019 en los Estados Unidos" . La Revista de Medicina de Nueva Inglaterra . 382 (10): 929–936. doi : 10.1056 / NEJMoa2001191 . PMC 7092802 . PMID 32004427 .
- ^ Li D, Jin M, Bao P, Zhao W, Zhang S (mayo de 2020). "Características clínicas y resultados de las pruebas de semen en hombres con enfermedad por coronavirus 2019" . Red JAMA abierta . 3 (5): e208292. doi : 10.1001 / jamanetworkopen.2020.8292 . PMC 7206502 . PMID 32379329 .
- ^ Wölfel R, Corman VM, Guggemos W, Seilmaier M, Zange S, Müller MA, et al. (Mayo de 2020). "Evaluación virológica de pacientes hospitalizados con COVID-2019" . Naturaleza . 581 (7809): 465–469. Código Bib : 2020Natur.581..465W . doi : 10.1038 / s41586-020-2196-x . PMID 32235945 .
- ^ Kupferschmidt K (febrero de 2020). "El estudio que afirma que el nuevo coronavirus puede ser transmitido por personas sin síntomas fue defectuoso". Ciencia . doi : 10.1126 / science.abb1524 .
- ^ Para KK, Tsang OT, Leung WS, Tam AR, Wu TC, Lung DC, et al. (Mayo de 2020). "Perfiles temporales de carga viral en muestras de saliva orofaríngea posterior y respuestas de anticuerpos séricos durante la infección por SARS-CoV-2: un estudio de cohorte observacional" . La lanceta. Enfermedades infecciosas . 20 (5): 565–574. doi : 10.1016 / S1473-3099 (20) 30196-1 . PMC 7158907 . PMID 32213337 .
- ^ Avanzato, Victoria A .; Matson, M. Jeremiah; Seifert, Stephanie N .; Pryce, Rhys; Williamson, Brandi N .; Anzick, Sarah L .; Barbian, Kent; Judson, Seth D .; Fischer, Elizabeth R .; Martens, Craig; Bowden, Thomas A .; de Wit, Emmie; Riedo, Francis X .; Munster, Vincent J. (diciembre de 2020). "Estudio de caso: desprendimiento prolongado de SARS-CoV-2 infeccioso de un individuo inmunodeprimido asintomático con cáncer" . Celular . 183 (7): 1901–1912.e9. doi : 10.1016 / j.cell.2020.10.049 . PMC 7640888 . PMID 33248470 .
- ^ a b Hou YJ, Okuda K, Edwards CE, Martinez DR, Asakura T, Dinnon KH, et al. (Julio de 2020). "La genética inversa del SARS-CoV-2 revela un gradiente de infección variable en el tracto respiratorio" . Celular . 182 (2): 429–446.e14. doi : 10.1016 / j.cell.2020.05.042 . PMC 7250779 . PMID 32526206 .
- ^ Banerjee, Arinjay; Mossman, Karen; Baker, Michelle L. (febrero de 2021). "Potencial zooantroponótico del SARS-CoV-2 e implicaciones de la reintroducción en poblaciones humanas". Anfitrión celular y microbio . 29 (2): 160-164. doi : 10.1016 / j.chom.2021.01.004 .
- ^ "Preguntas y respuestas sobre el COVID-19: OIE - Organización Mundial de Sanidad Animal" . www.oie.int . Archivado desde el original el 31 de marzo de 2020 . Consultado el 16 de abril de 2020 .
- ^ Goldstein J (6 de abril de 2020). "El tigre del zoológico del Bronx está enfermo de coronavirus" . The New York Times . Archivado desde el original el 9 de abril de 2020 . Consultado el 10 de abril de 2020 .
- ^ "Declaración del USDA sobre la confirmación de COVID-19 en un tigre en Nueva York" . Departamento de Agricultura de los Estados Unidos . 5 de abril de 2020. Archivado desde el original el 15 de abril de 2020 . Consultado el 16 de abril de 2020 .
- ^ "Si tienes animales: enfermedad por coronavirus 2019 (COVID-19)" . Centros para el Control y la Prevención de Enfermedades (CDC) . 13 de abril de 2020. Archivado desde el original el 1 de abril de 2020 . Consultado el 16 de abril de 2020 .
- ^ Organización Mundial de la Salud (1 de febrero de 2020). Nuevo coronavirus (2019-nCoV): informe de situación, 12 (Informe). Organización Mundial de la Salud . hdl : 10665/330777 .
- ^ Nogrady B (noviembre de 2020). "Lo que dicen los datos sobre las infecciones COVID asintomáticas" . Naturaleza . 587 (7835): 534–535. Código Bib : 2020Natur.587..534N . doi : 10.1038 / d41586-020-03141-3 . PMID 33214725 .
- ^ Li R, Pei S, Chen B, Song Y, Zhang T, Yang W, Shaman J (mayo de 2020). "Una infección sustancial indocumentada facilita la rápida diseminación del nuevo coronavirus (SARS-CoV-2)" . Ciencia . 368 (6490): 489–493. Código Bibliográfico : 2020Sci ... 368..489L . doi : 10.1126 / science.abb3221 . PMC 7164387 . PMID 32179701 .
- ^ Daily Telegraph , jueves 28 de mayo de 2020, página 2 columna 1, que se refiere a la revista médica Thorax ; Tórax de mayo de 2020 el artículo COVID-19: los pasos de Ernest Shackleton archivado 30 de de mayo de 2020 como Wayback Machine
- ^ He X, Lau EH, Wu P, Deng X, Wang J, Hao X, et al. (Mayo de 2020). "Dinámica temporal en la diseminación viral y transmisibilidad de COVID-19" . Medicina de la naturaleza . 26 (5): 672–675. doi : 10.1038 / s41591-020-0869-5 . PMID 32296168 .
- ^ a b Ledford H (septiembre de 2020). "Reinfecciones por coronavirus: tres preguntas que se hacen los científicos" . Naturaleza . 585 (7824): 168–169. doi : 10.1038 / d41586-020-02506-y . PMID 32887957 . S2CID 221501940 .
- ^ a b Para KK, Hung IF, Ip JD, Chu AW, Chan WM, Tam AR, et al. (Agosto de 2020). "Reinfección por COVID-19 por una cepa filogenéticamente distinta del coronavirus-2 del SARS confirmada por secuenciación del genoma completo" . Enfermedades Infecciosas Clínicas : ciaa1275. doi : 10.1093 / cid / ciaa1275 . PMC 7499500 . PMID 32840608 . S2CID 221308584 .
- ^ a b Tillett RL, Sevinsky JR, Hartley PD, Kerwin H, Crawford N, Gorzalski A, et al. (Enero de 2021). "Evidencia genómica de reinfección con SARS-CoV-2: un estudio de caso" . La lanceta. Enfermedades infecciosas . 21 (1): 52–58. doi : 10.1016 / S1473-3099 (20) 30764-7 . PMC 7550103 . PMID 33058797 .
- ^ Eschner K (28 de enero de 2020). "Todavía no estamos seguros de dónde vino realmente el coronavirus de Wuhan" . Ciencia popular . Archivado desde el original el 30 de enero de 2020 . Consultado el 30 de enero de 2020 .
- ^ Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, et al. (Febrero de 2020). "Características clínicas de los pacientes infectados con el nuevo coronavirus de 2019 en Wuhan, China" . Lancet . 395 (10223): 497–506. doi : 10.1016 / S0140-6736 (20) 30183-5 . PMC 7159299 . PMID 31986264 .
- ^ a b Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, et al. (Febrero de 2020). "Características epidemiológicas y clínicas de 99 casos de neumonía por coronavirus nuevo de 2019 en Wuhan, China: un estudio descriptivo" . Lancet . 395 (10223): 507–513. doi : 10.1016 / S0140-6736 (20) 30211-7 . PMC 7135076 . PMID 32007143 .
- ^ a b Cyranoski D (marzo de 2020). "El misterio se profundiza sobre la fuente animal del coronavirus" . Naturaleza . 579 (7797): 18-19. Código Bib : 2020Natur.579 ... 18C . doi : 10.1038 / d41586-020-00548-w . PMID 32127703 .
- ^ Yu, Wen-Bin; Tang, Guang-Da; Zhang, Li; T. Corlett, Richard (2020). "Decodificación de la evolución y transmisiones del nuevo coronavirus de neumonía (SARS-CoV-2 / HCoV-19) utilizando datos genómicos completos" . Investigación Zoológica . 41 (3): 247–257. doi : 10.24272 / j.issn.2095-8137.2020.022 . PMC 7231477 . PMID 32351056 .
- ^ a b c d e f g Informe de la misión conjunta OMS-China sobre la enfermedad por coronavirus 2019 (COVID-19) (PDF) (Informe). Organización Mundial de la Salud (OMS). 24 de febrero de 2020. Archivado (PDF) desde el original el 29 de febrero de 2020 . Consultado el 5 de marzo de 2020 .
- ^ Kang, Lin; Él, Guijuan; Sharp, Amanda K .; Wang, Xiaofeng; Brown, Anne M .; Michalak, Pawel; Weger-Lucarelli, James (julio de 2021). "Un barrido selectivo en el gen Spike ha impulsado la adaptación humana del SARS-CoV-2". Móvil : S0092867421008333. doi : 10.1016 / j.cell.2021.07.007 .
- ^ Decaro, Nicola; Lorusso, Alessio (mayo de 2020). "Nuevo coronavirus humano (SARS-CoV-2): una lección de coronavirus animales". Microbiología veterinaria . 244 : 108693. doi : 10.1016 / j.vetmic.2020.108693 .
- ^ Robson F, Khan KS, Le TK, Paris C, Demirbag S, Barfuss P, et al. (Agosto de 2020). "Corrección de ARN de coronavirus: base molecular y orientación terapéutica [la corrección publicada aparece en Mol Cell. 2020 17 de diciembre; 80 (6): 1136-1138]" . Célula molecular . 79 (5): 710–727. doi : 10.1016 / j.molcel.2020.07.027 . PMC 7402271 . PMID 32853546 .
- ^ Benvenuto D, Giovanetti M, Ciccozzi A, Spoto S, Angeletti S, Ciccozzi M (abril de 2020). "La epidemia de coronavirus nuevo 2019: evidencia de la evolución del virus" . Revista de Virología Médica . 92 (4): 455–459. doi : 10.1002 / jmv.25688 . PMC 7166400 . PMID 31994738 .
- ^ "Bat-SL-CoVZC45 aislado de coronavirus similar al SARS de murciélago, genoma completo" . Centro Nacional de Información Biotecnológica (NCBI) . 15 de febrero de 2020. Archivado desde el original el 4 de junio de 2020 . Consultado el 15 de febrero de 2020 .
- ^ "Bat-SL-CoVZXC21 de aislamiento de coronavirus similar al SARS de murciélago, genoma completo" . Centro Nacional de Información Biotecnológica (NCBI) . 15 de febrero de 2020. Archivado desde el original el 4 de junio de 2020 . Consultado el 15 de febrero de 2020 .
- ^ "Bat coronavirus aislar RaTG13, genoma completo" . Centro Nacional de Información Biotecnológica (NCBI) . 10 de febrero de 2020. Archivado desde el original el 15 de mayo de 2020 . Consultado el 5 de marzo de 2020 .
- ^ "El 'argumento de la navaja de Occam' no ha cambiado a favor de una fuga de laboratorio" . Snopes.com . Snopes . Consultado el 18 de julio de 2021 .
- ^ Zhou, Hong; Ji, Jingkai; Chen, Xing; Bi, Yuhai; Li, Juan; Wang, Qihui; et al. (Junio de 2021). "La identificación de nuevos coronavirus de murciélago arroja luz sobre los orígenes evolutivos del SARS-CoV-2 y virus relacionados" . Móvil (Cambridge) : S0092867421007091. doi : 10.1016 / j.cell.2021.06.008 . ISSN 0092-8674 . PMC 8188299 . PMID 34147139 . Archivado desde el original el 18 de junio de 2021 . Consultado el 18 de junio de 2021 .
- ^ Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, et al. (Febrero de 2020). "Caracterización genómica y epidemiología del nuevo coronavirus de 2019: implicaciones para los orígenes del virus y la unión al receptor" . Lancet . 395 (10224): 565–574. doi : 10.1016 / S0140-6736 (20) 30251-8 . PMC 7159086 . PMID 32007145 .
- ^ O'Keeffe J, Freeman S, Nicol A (21 de marzo de 2021). Conceptos básicos de la transmisión del SARS-CoV-2 . Vancouver, BC: Centro Colaborador Nacional para la Salud Ambiental (NCCEH). ISBN 978-1-988234-54-0. Archivado desde el original el 12 de mayo de 2021 . Consultado el 12 de mayo de 2021 .
- ^ Xiao K, Zhai J, Feng Y, Zhou N, Zhang X, Zou JJ, et al. (Julio de 2020). "Aislamiento de coronavirus relacionados con el SARS-CoV-2 de pangolines malayos" . Naturaleza . 583 (7815): 286–289. Código Bib : 2020Natur.583..286X . doi : 10.1038 / s41586-020-2313-x . PMID 32380510 . S2CID 218557880 .
- ↑ Zhao J, Cui W, Tian BP (2020). "Los anfitriones intermedios potenciales para el SARS-CoV-2" . Fronteras en microbiología . 11 : 580137. doi : 10.3389 / fmicb.2020.580137 . PMC 7554366 . PMID 33101254 .
- ^ a b c Hu B, Guo H, Zhou P, Shi ZL (marzo de 2021). "Características de SARS-CoV-2 y COVID-19" . Reseñas de la naturaleza. Microbiología . 19 (3): 141-154. doi : 10.1038 / s41579-020-00459-7 . PMC 7537588 . PMID 33024307 .
- ^ Giovanetti M, Benedetti F, Campisi G, Ciccozzi A, Fabris S, Ceccarelli G, et al. (Enero de 2021). "Patrones de evolución de SARS-CoV-2: instantánea sobre sus variantes del genoma" . Comunicaciones de investigación bioquímica y biofísica . 538 : 88–91. doi : 10.1016 / j.bbrc.2020.10.102 . PMC 7836704 . PMID 33199021 . S2CID 226988090 .
- ↑ a b c V'kovski P, Kratzel A, Steiner S, Stalder H, Thiel V (marzo de 2021). "Biología y replicación del coronavirus: implicaciones para el SARS-CoV-2" . Reseñas de la naturaleza. Microbiología . 19 (3): 155-170. doi : 10.1038 / s41579-020-00468-6 . PMC 7592455 . PMID 33116300 .
- ^ Zhu N, Zhang D, Wang W, Li X, Yang B, Song J, et al. (Febrero de 2020). "Un nuevo coronavirus de pacientes con neumonía en China, 2019" . La Revista de Medicina de Nueva Inglaterra . 382 (8): 727–733. doi : 10.1056 / NEJMoa2001017 . PMC 7092803 . PMID 31978945 .
- ^ "Filogenia de betacoronavirus similares al SARS" . nextstrain . Archivado desde el original el 20 de enero de 2020 . Consultado el 18 de enero de 2020 .
- ^ Wong AC, Li X, Lau SK, Woo PC (febrero de 2019). "Epidemiología global de los coronavirus de murciélago" . Virus . 11 (2): 174. doi : 10.3390 / v11020174 . PMC 6409556 . PMID 30791586 .
- ↑ a b Singh D, Yi SV (abril de 2021). "Sobre el origen y evolución del SARS-CoV-2" . Medicina experimental y molecular . 53 (4): 537–547. doi : 10.1038 / s12276-021-00604-z . PMC 8050477 . PMID 33864026 .
- ^ a b "CoV2020" . GISAID EpifluDB . Archivado desde el original el 12 de enero de 2020 . Consultado el 12 de enero de 2020 .
- ^ Kim D, Lee JY, Yang JS, Kim JW, Kim VN, Chang H (mayo de 2020). "La arquitectura del transcriptoma SARS-CoV-2" . Celular . 181 (4): 914–921.e10. doi : 10.1016 / j.cell.2020.04.011 . PMC 7179501 . PMID 32330414 .
- ^ Para KK, Sridhar S, Chiu KH, Hung DL, Li X, Hung IF, et al. (Diciembre de 2021). "Lecciones aprendidas 1 año después de la aparición de SARS-CoV-2 que conduce a la pandemia de COVID-19" . Microbios e infecciones emergentes . 10 (1): 507–535. doi : 10.1080 / 22221751.2021.1898291 . PMC 8006950 . PMID 33666147 .
- ↑ Braun E, Sauter D (2019). "Procesamiento de proteínas mediado por furina en enfermedades infecciosas y cáncer" . Inmunología clínica y traslacional . 8 (8): e1073. doi : 10.1002 / cti2.1073 . PMC 6682551 . PMID 31406574 .
- ^ Vankadari N (agosto de 2020). "Estructura de la unión de la proteasa de furina a la glicoproteína de pico de SARS-CoV-2 e implicaciones para posibles objetivos y virulencia" . La Revista de Cartas de Química Física . 11 (16): 6655–6663. doi : 10.1021 / acs.jpclett.0c01698 . PMC 7409919 . PMID 32787225 .
- ^ a b Coutard B, Valle C, de Lamballerie X, Canard B, Seidah NG, Decroly E (abril de 2020). "La glicoproteína de pico del nuevo coronavirus 2019-nCoV contiene un sitio de escisión similar a furina ausente en CoV del mismo clado" . Investigación antiviral . 176 : 104742. doi : 10.1016 / j.cub.2020.03.022 . PMC 7114094 . PMID 32057769 .
- ^ Zhang T, Wu Q, Zhang Z (abril de 2020). "Origen probable del pangolín del SARS-CoV-2 asociado con el brote de COVID-19" . Biología actual . 30 (7): 1346-1351.e2. doi : 10.1016 / j.cub.2020.03.022 . PMC 7156161 . PMID 32197085 .
- ^ Wu Y, Zhao S (diciembre de 2020). "Los sitios de escisión de la furina ocurren naturalmente en los coronavirus" . Investigación con células madre . 50 : 102115. doi : 10.1016 / j.scr.2020.102115 . PMC 7836551 . PMID 33340798 .
- ^ Worobey M, Pekar J, Larsen BB, Nelson MI, Hill V, Joy JB, et al. (Octubre de 2020). "La aparición de SARS-CoV-2 en Europa y América del Norte" . Ciencia . 370 (6516): 564–570. doi : 10.1126 / science.abc8169 . PMC 7810038 . PMID 32912998 .
- ^ "Lanzamiento inicial del genoma del nuevo coronavirus" . Virológico . 11 de enero de 2020. Archivado desde el original el 12 de enero de 2020 . Consultado el 12 de enero de 2020 .
- ^ a b Bedford T, Neher R, Hadfield N, Hodcroft E, Ilcisin M, Müller N. "Análisis genómico de la propagación de nCoV: informe de situación 2020-01-30" . nextstrain.org . Archivado desde el original el 15 de marzo de 2020 . Consultado el 18 de marzo de 2020 .
- ^ Sol, Jiumeng; Él, Wan-Ting; Wang, Lifang; Lai, Alexander; Ji, Xiang; Zhai, Xiaofeng; Li, Gairu; Suchard, Marc A .; Tian, Jin; Zhou, Jiyong; Veit, Michael; Su, Shuo (mayo de 2020). "COVID-19: epidemiología, evolución y perspectivas interdisciplinarias" . Tendencias en Medicina Molecular . 26 (5): 483–495. doi : 10.1016 / j.molmed.2020.02.008 . PMC 7118693 . PMID 32359479 .
- ^ "Epidemiología genómica del nuevo coronavirus - submuestreo global" . Nextstrain . Archivado desde el original el 20 de abril de 2020 . Consultado el 7 de mayo de 2020 .
- ^ Grupo de estudio de Coronaviridae del Comité Internacional de Taxonomía de Virus (abril de 2020). "La especie Coronavirus relacionado con el síndrome respiratorio agudo severo: clasificando 2019-nCoV y nombrándolo SARS-CoV-2" . Microbiología de la naturaleza . 5 (4): 536–544. doi : 10.1038 / s41564-020-0695-z . PMC 7095448 . PMID 32123347 .
- ^ "La nueva cepa más infecciosa de COVID-19 ahora domina los casos globales de virus: estudio" . medicalxpress.com . Archivado desde el original el 17 de noviembre de 2020 . Consultado el 16 de agosto de 2020 .
- ^ Korber B, Fischer WM, Gnanakaran S, Yoon H, Theiler J, Abfalterer W, et al. (Agosto de 2020). "Seguimiento de cambios en pico de SARS-CoV-2: evidencia de que D614G aumenta la infectividad del virus COVID-19" . Celular . 182 (4): 812–827.e19. doi : 10.1016 / j.cell.2020.06.043 . PMC 7332439 . PMID 32697968 .
- ^ a b Dhama K, Khan S, Tiwari R, Sircar S, Bhat S, Malik YS, et al. (Septiembre de 2020). "Enfermedad por coronavirus 2019-COVID-19" . Revisiones de microbiología clínica . 33 (4). doi : 10.1128 / CMR.00028-20 . PMC 7405836 . PMID 32580969 .
- ^ Dockrill P (11 de noviembre de 2020). "Los científicos acaban de encontrar un 'gen misteriosamente oculto dentro de un gen' en el SARS-CoV-2" . ScienceAlert . Archivado desde el original el 17 de noviembre de 2020 . Consultado el 11 de noviembre de 2020 .
- ^ Nelson CW, Ardern Z, Goldberg TL, Meng C, Kuo CH, Ludwig C, et al. (Octubre de 2020). "Gen superpuesto novedoso en evolución dinámica como un factor en la pandemia de SARS-CoV-2" . eLife . 9 . doi : 10.7554 / eLife.59633 . PMC 7655111 . PMID 33001029 . Archivado desde el original el 17 de noviembre de 2020 . Consultado el 11 de noviembre de 2020 .
- ^ a b Zhou H, Ji J, Chen X, Bi Y, Li J, Wang Q, et al. (Junio de 2021). "La identificación de nuevos coronavirus de murciélago arroja luz sobre los orígenes evolutivos del SARS-CoV-2 y virus relacionados". Móvil : S0092867421007091. doi : 10.1016 / j.cell.2021.06.008 . PMID 34147139 .
- ^ a b Wacharapluesadee S, Tan CW, Maneeorn P, Duengkae P, Zhu F, Joyjinda Y, et al. (Febrero de 2021). "Evidencia de coronavirus relacionados con el SARS-CoV-2 que circulan en murciélagos y pangolines en el sudeste asiático" . Comunicaciones de la naturaleza . 12 (1): 972. doi : 10.1038 / s41467-021-21240-1 . PMC 7873279 . PMID 33563978 .
- ^ a b c Hul V, Delaune D, Karlsson EA, Hassanin A, Tey PO, Baidaliuk A, et al. (26 de enero de 2021). "Un nuevo coronavirus relacionado con el SARS-CoV-2 en murciélagos de Camboya" . bioRxiv . págs. 2021.01.26.428212. doi : 10.1101 / 2021.01.26.428212 .
- ^ Murakami S, Kitamura T, Suzuki J, Sato R, Aoi T, Fujii M, et al. (Diciembre de 2020). "Detección y caracterización del sarbecovirus de murciélago relacionado filogenéticamente con el SARS-CoV-2, Japón" . Enfermedades infecciosas emergentes . 26 (12): 3025-3029. doi : 10.3201 / eid2612.203386 . PMC 7706965 . PMID 33219796 .
- ^ a b Zhou H, Chen X, Hu T, Li J, Song H, Liu Y, et al. (Junio de 2020). "Un nuevo coronavirus de murciélago estrechamente relacionado con el SARS-CoV-2 contiene inserciones naturales en el sitio de división S1 / S2 de la proteína Spike". Biología actual . 30 (11): 2196–2203.e3. doi : 10.1016 / j.cub.2020.05.023 . PMID 32416074 .
- ^ Lam TT, Jia N, Zhang YW, Shum MH, Jiang JF, Zhu HC, et al. (Julio de 2020). "Identificación de coronavirus relacionados con el SARS-CoV-2 en pangolines malayos". Naturaleza . 583 (7815): 282–285. doi : 10.1038 / s41586-020-2169-0 . PMID 32218527 .
- ^ Liu P, Jiang JZ, Wan XF, Hua Y, Li L, Zhou J, et al. (Mayo de 2020). "¿Son los pangolines el hospedador intermediario del nuevo coronavirus de 2019 (SARS-CoV-2)?". PLoS Pathogens . 16 (5): e1008421. doi : 10.1371 / journal.ppat.1008421 . PMID 32407364 .
- ^ Zhou H, Chen X, Hu T, Li J, Song H, Liu Y, et al. (Junio de 2020). "Un nuevo coronavirus de murciélago estrechamente relacionado con el SARS-CoV-2 contiene inserciones naturales en el sitio de división S1 / S2 de la proteína Spike". Biología actual . 30 (11): 2196–2203.e3. doi : 10.1016 / j.cub.2020.05.023 . PMID 32416074 .
- ^ Koyama T, Platt D, Parida L (julio de 2020). "Análisis de variantes de genomas de SARS-CoV-2" . Boletín de la Organización Mundial de la Salud . 98 (7): 495–504. doi : 10.2471 / BLT.20.253591 . PMC 7375210 . PMID 32742035 .
Detectamos en total 65776 variantes con 5775 variantes distintas.
- ^ Alm E, Broberg EK, Connor T, Hodcroft EB, Komissarov AB, Maurer-Stroh S, et al. (Agosto de 2020). "Distribución geográfica y temporal de los clados del SARS-CoV-2 en la región europea de la OMS, de enero a junio de 2020" . Vigilancia del euro . 25 (32). doi : 10.2807 / 1560-7917.ES.2020.25.32.2001410 . PMC 7427299 . PMID 32794443 .
- ^ Organización Mundial de la Salud (31 de mayo de 2021). "Seguimiento de variantes del SARS-CoV-2" . Organización Mundial de la Salud . Archivado desde el original el 6 de junio de 2021 . Consultado el 8 de junio de 2021 .
- ^ "Cepa variante asociada al visón SARS-CoV-2 - Dinamarca" . QUIEN . 3 de diciembre de 2020. Archivado desde el original el 31 de diciembre de 2020 . Consultado el 30 de diciembre de 2020 .
- ^ a b c Wu C, Liu Y, Yang Y, Zhang P, Zhong W, Wang Y, et al. (Mayo de 2020). "Análisis de dianas terapéuticas para el SARS-CoV-2 y descubrimiento de fármacos potenciales por métodos computacionales" . Acta Pharmaceutica Sínica B . 10 (5): 766–788. doi : 10.1016 / j.apsb.2020.02.008 . PMC 7102550 . PMID 32292689 .
- ^ a b Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, et al. (Marzo de 2020). "Estructura Cryo-EM del pico de 2019-nCoV en la conformación de prefusión" . Ciencia . 367 (6483): 1260–1263. Código Bibliográfico : 2020Sci ... 367.1260W . doi : 10.1126 / science.abb2507 . PMC 7164637 . PMID 32075877 .
- ^ Mandelbaum RF (19 de febrero de 2020). "Los científicos crean una imagen de nivel atómico del potencial talón de Aquiles del nuevo coronavirus" . Gizmodo . Archivado desde el original el 8 de marzo de 2020 . Consultado el 13 de marzo de 2020 .
- ^ Kandeel M, Ibrahim A, Fayez M, Al-Nazawi M (junio de 2020). "De SARS y MERS CoVs a SARS-CoV-2: avanzando hacia un uso de codones más sesgado en genes virales estructurales y no estructurales" . Revista de Virología Médica . 92 (6): 660–666. doi : 10.1002 / jmv.25754 . PMC 7228358 . PMID 32159237 .
- ^ a b Hou W (septiembre de 2020). "Caracterización del patrón de uso de codones en SARS-CoV-2" . Revista de virología . 17 (1): 138. doi : 10.1186 / s12985-020-01395-x . PMC 7487440 . PMID 32928234 .
- ^ a b Wang Y, Mao JM, Wang GD, Luo ZP, Yang L, Yao Q, Chen KP (julio de 2020). "El SARS-CoV-2 humano ha evolucionado para reducir el dinucleótido CG en sus marcos de lectura abiertos" . Informes científicos . 10 (1): 12331. Código Bibliográfico : 2020NatSR..1012331W . doi : 10.1038 / s41598-020-69342-y . PMC 7378049 . PMID 32704018 .
- ^ Rice AM, Castillo Morales A, Ho AT, Mordstein C, Mühlhausen S, Watson S, et al. (Enero de 2021). "Evidencia de un fuerte sesgo de mutación hacia y selección contra el contenido de U en el SARS-CoV-2: implicaciones para el diseño de la vacuna" . Biología Molecular y Evolución . 38 (1): 67–83. doi : 10.1093 / molbev / msaa188 . PMC 7454790 . PMID 32687176 .
- ^ Gu H, Chu DK, Peiris M, Poon LL (enero de 2020). "Análisis multivariados del uso de codones de SARS-CoV-2 y otros betacoronavirus" . Evolución del virus . 6 (1): veaa032. doi : 10.1093 / ve / veaa032 . PMC 7223271 . PMID 32431949 .
- ^ Wang Q, Zhang Y, Wu L, Niu S, Song C, Zhang Z, et al. (Mayo de 2020). "Base estructural y funcional de la entrada de SARS-CoV-2 mediante el uso de ACE2 humano" . Celular . 181 (4): 894–904.e9. doi : 10.1016 / j.cell.2020.03.045 . PMC 7144619 . PMID 32275855 .
- ^ Xu X, Chen P, Wang J, Feng J, Zhou H, Li X, et al. (Marzo de 2020). "Evolución del nuevo coronavirus del brote en curso de Wuhan y modelado de su proteína de pico para el riesgo de transmisión humana" . Science China Life Sciences . 63 (3): 457–460. doi : 10.1007 / s11427-020-1637-5 . PMC 7089049 . PMID 32009228 .
- ^ Letko M, Munster V (enero de 2020). "Evaluación funcional de la entrada de células y el uso de receptores para el linaje B β-coronavirus, incluido 2019-nCoV" (PDF) . bioRxiv (preimpresión). doi : 10.1101 / 2020.01.22.915660 . PMC 7217099 . PMID 32511294 . Archivado (PDF) desde el original el 22 de abril de 2020 . Consultado el 5 de mayo de 2020 .
- ^ Letko M, Marzi A, Munster V (abril de 2020). "Evaluación funcional de la entrada celular y el uso de receptores para el SARS-CoV-2 y otros betacoronavirus de linaje B" . Microbiología de la naturaleza . 5 (4): 562–569. doi : 10.1038 / s41564-020-0688-y . PMC 7095430 . PMID 32094589 .
- ^ El Sahly HM. "Caracterización genómica del nuevo coronavirus 2019" . La Revista de Medicina de Nueva Inglaterra . Archivado desde el original el 17 de febrero de 2020 . Consultado el 9 de febrero de 2020 .
- ^ "La estructura del nuevo coronavirus revela objetivos para vacunas y tratamientos" . Institutos Nacionales de Salud (NIH) . 2 de marzo de 2020. Archivado desde el original el 1 de abril de 2020 . Consultado el 3 de abril de 2020 .
- ^ Wang K, Chen W, Zhou YS, Lian JQ, Zhang Z, Du P, et al. (14 de marzo de 2020). "El SARS-CoV-2 invade las células huésped a través de una ruta novedosa: proteína de pico CD147" (PDF) . bioRxiv (preimpresión). doi : 10.1101 / 2020.03.14.988345 . S2CID 214725955 . Archivado (PDF) desde el original el 11 de mayo de 2020 . Consultado el 5 de mayo de 2020 .
- ^ Zamorano Cuervo N, Grandvaux N (noviembre de 2020). "ACE2: evidencia de papel como receptor de entrada para SARS-CoV-2 e implicaciones en comorbilidades" . eLife . 9 . doi : 10.7554 / eLife.61390 . PMC 7652413 . PMID 33164751 .
- ^ "Anatomía de un asesino: comprensión del SARS-CoV-2 y las drogas que podrían disminuir su poder" . The Economist . 12 de marzo de 2020. Archivado desde el original el 14 de marzo de 2020 . Consultado el 14 de marzo de 2020 .
- ^ Beeching NJ, Fletcher TE, Fowler R (22 de mayo de 2020). "Mejores prácticas de BMJ: Enfermedad por coronavirus 2019 (COVID-19)" (PDF) . BMJ . Archivado (PDF) desde el original el 13 de junio de 2020 . Consultado el 25 de mayo de 2020 .
- ^ Oberholzer M, Febbo P (19 de febrero de 2020). "Lo que sabemos hoy sobre el coronavirus SARS-CoV-2 y hacia dónde vamos a partir de aquí" . Noticias de Ingeniería Genética y Biotecnología . Archivado desde el original el 14 de marzo de 2020 . Consultado el 13 de marzo de 2020 .
- ^ Ma J (13 de marzo de 2020). "Coronavirus: el primer caso confirmado de Covid-19 en China se remonta al 17 de noviembre" . Poste de la mañana del sur de China . Archivado desde el original el 13 de marzo de 2020 . Consultado el 16 de marzo de 2020 .
- ^ a b Pekar, Jonathan; Worobey, Michael; Moshiri, Niema; Scheffler, Konrad; Wertheim, Joel O. (23 de abril de 2021). "Programación del caso índice SARS-CoV-2 en la provincia de Hubei" . Ciencia . 372 (6540): 412–417. Código Bibliográfico : 2021Sci ... 372..412P . doi : 10.1126 / science.abf8003 . ISSN 1095-9203 . PMC 8139421 . PMID 33737402 .
- ^ Para, Kelvin Kai-Wang; Sridhar, Siddharth; Chiu, Kelvin Hei-Yeung; Hung, Derek Ling-Lung; Li, Xin; Hung, Ivan Fan-Ngai; Tam, Anthony Raymond; Chung, Tom Wai-Hin; Chan, Jasper Fuk-Woo; Zhang, Anna Jian-Xia; Cheng, Vincent Chi-Chung; Yuen, Kwok-Yung (22 de marzo de 2021). "Lecciones aprendidas 1 año después de la aparición de SARS-CoV-2 que conduce a la pandemia de COVID-19" . Microbios e infecciones emergentes . 10 (1): 507–535. doi : 10.1080 / 22221751.2021.1898291 . ISSN 2222-1751 . PMC 8006950 . PMID 33666147 .
- ^ a b c d e "Panel de COVID-19 por el Centro de ciencia e ingeniería de sistemas (CSSE) en la Universidad Johns Hopkins (JHU)" . ArcGIS . Universidad Johns Hopkins . Consultado el 1 de agosto de 2021 .
- ^ Informe de situación de la enfermedad por coronavirus 2019 (COVID-19) - 69 (Informe). Organización Mundial de la Salud . 29 de marzo de 2020. hdl : 10665/331615 .
- ^ Wee SL, McNeil Jr DG, Hernández JC (30 de enero de 2020). "La OMS declara emergencia mundial como se propaga el coronavirus de Wuhan" . The New York Times . Archivado desde el original el 30 de enero de 2020 . Consultado el 30 de enero de 2020 .
- ^ McKay B, Calfas J, Ansari T (11 de marzo de 2020). "Coronavirus declarado pandemia por la Organización Mundial de la Salud" . El Wall Street Journal . Archivado desde el original el 11 de marzo de 2020 . Consultado el 12 de marzo de 2020 .
- ^ Rocklöv J, Sjödin H, Wilder-Smith A (mayo de 2020). "Brote de COVID-19 en el crucero Diamond Princess: estimación del potencial epidémico y la eficacia de las contramedidas de salud pública" . Revista de Medicina del Viaje . 27 (3). doi : 10.1093 / jtm / taaa030 . PMC 7107563 . PMID 32109273 .
- ^ Branswell H (30 de enero de 2020). "Los datos limitados sobre el coronavirus pueden estar sesgando las suposiciones sobre la gravedad" . STAT . Archivado desde el original el 1 de febrero de 2020 . Consultado el 13 de marzo de 2020 .
- ^ Wu JT, Leung K, Leung GM (febrero de 2020). "Pronosticar y pronosticar la posible propagación nacional e internacional del brote de 2019-nCoV que se originó en Wuhan, China: un estudio de modelado" . Lancet . 395 (10225): 689–697. doi : 10.1016 / S0140-6736 (20) 30260-9 . PMC 7159271 . PMID 32014114 .
- ^ Boseley S, McCurry J (30 de enero de 2020). "Las muertes por coronavirus aumentan en China mientras los países luchan por evacuar a los ciudadanos" . The Guardian . Archivado desde el original el 6 de febrero de 2020 . Consultado el 10 de marzo de 2020 .
- ^ Paulinus A (25 de febrero de 2020). "Coronavirus: China para compensar a África en la protección de la salud pública" . El sol . Archivado desde el original el 9 de marzo de 2020 . Consultado el 10 de marzo de 2020 .
Error de cita: una referencia definida por lista llamada "NYT-SpikyBlob" no se utiliza en el contenido (consulte la página de ayuda ).
Otras lecturas
- Bar-On YM, Flamholz A, Phillips R, Milo R (abril de 2020). "SARS-CoV-2 (COVID-19) por los números" . eLife . 9 . arXiv : 2003.12886 . Código bibliográfico : 2020arXiv200312886B . doi : 10.7554 / eLife.57309 . PMC 7224694 . PMID 32228860 .
- Brüssow H (mayo de 2020). "El nuevo coronavirus - una instantánea del conocimiento actual" . Biotecnología microbiana . 13 (3): 607–612. doi : 10.1111 / 1751-7915.13557 . PMC 7111068 . PMID 32144890 .
- Cascella M, Rajnik M, Aleem A, Dulebohn S, Di Napoli R (enero de 2020). "Características, Evaluación y Tratamiento del Coronavirus (COVID-19)" . StatPearls . PMID 32150360 . Archivado desde el original el 6 de abril de 2020 . Consultado el 4 de abril de 2020 .
- Pruebas de laboratorio para la enfermedad por coronavirus 2019 (COVID-19) en casos humanos sospechosos (Informe). Organización Mundial de la Salud . 2 de marzo de 2020. hdl : 10665/331329 .
- Zoumpourlis V, Goulielmaki M, Rizos E, Baliou S, Spandidos DA (octubre de 2020). "[Comentario] La pandemia de COVID-19 como desafío científico y social en el siglo XXI" . Informes de medicina molecular (revisión). 22 (4): 3035-3048. doi : 10.3892 / mmr.2020.11393 . PMC 7453598 . PMID 32945405 .
enlaces externos
- "Enfermedad por coronavirus 2019 (COVID-19)" . Centros para el Control y la Prevención de Enfermedades (CDC) . 11 de febrero de 2020.
- "Pandemia de enfermedad por coronavirus (COVID-19)" . Organización Mundial de la Salud (OMS) .
- "Secuencias de SARS-CoV-2 (coronavirus 2 del síndrome respiratorio agudo severo)" . Centro Nacional de Información Biotecnológica (NCBI) .
- "Centro de recursos COVID-19" . The Lancet .
- "Coronavirus (Covid-19)" . La Revista de Medicina de Nueva Inglaterra .
- "Covid-19: brote de coronavirus nuevo" . Wiley .
- "SARS-CoV-2" . Recurso de análisis y base de datos de patógenos de virus .
- "Estructuras proteicas relacionadas con el SARS-CoV-2" . Banco de datos de proteínas .
| Temas relacionados con el acceso a portales |
|
| Obtenga más información sobre los proyectos Sister de Wikipedia |
|
| Clasificación | D
|
|---|
