| Glosario de acoplamiento |
|---|
|
| editar |
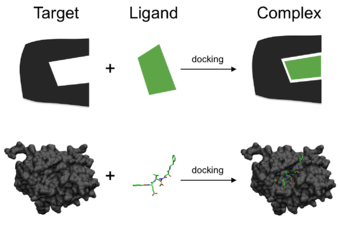
En el campo del modelado molecular , el acoplamiento es un método que predice la orientación preferida de una molécula a una segunda cuando se unen entre sí para formar un complejo estable . [1] El conocimiento de la orientación preferida, a su vez, puede usarse para predecir la fuerza de asociación o afinidad de unión entre dos moléculas usando, por ejemplo, funciones de puntuación .
Las asociaciones entre moléculas biológicamente relevantes como proteínas , péptidos , ácidos nucleicos , carbohidratos y lípidos juegan un papel central en la transducción de señales . Además, la orientación relativa de los dos compañeros que interactúan puede afectar el tipo de señal producida (p. Ej., Agonismo frente a antagonismo ). Por lo tanto, el acoplamiento es útil para predecir tanto la intensidad como el tipo de señal producida.
El acoplamiento molecular es uno de los métodos más utilizados en el diseño de fármacos basados en la estructura , debido a su capacidad para predecir la conformación de unión de ligandos de moléculas pequeñas al sitio de unión objetivo apropiado . La caracterización del comportamiento de unión juega un papel importante en el diseño racional de fármacos , así como para dilucidar los procesos bioquímicos fundamentales. [2] [3]
Definición de problema [ editar ]
Uno puede pensar en el acoplamiento molecular como un problema de "cerradura y llave" , en el que uno quiere encontrar la orientación relativa correcta de la "llave" que abrirá la "cerradura" (donde en la superficie de la cerradura está el orificio de la cerradura, en qué dirección girar la llave después de insertarla, etc.). Aquí, la proteína se puede considerar como el "bloqueo" y el ligando como una "llave". El acoplamiento molecular puede definirse como un problema de optimización, que describiría la orientación de "mejor ajuste" de un ligando que se une a una proteína de interés particular. Sin embargo, dado que tanto el ligando como la proteína son flexibles, una analogía de "mano en guante" es más apropiada que "cerradura y llave" . [4] Durante el curso del proceso de acoplamiento, el ligando y la proteína ajustan su conformación para lograr un "mejor ajuste" general y este tipo de ajuste conformacional que da como resultado la unión general se denomina "ajuste inducido" . [5]
La investigación del acoplamiento molecular se centra en la simulación computacional del proceso de reconocimiento molecular . Su objetivo es lograr una conformación optimizada tanto para la proteína como para el ligando y una orientación relativa entre la proteína y el ligando de manera que se minimice la energía libre del sistema general.
Enfoques de atraque [ editar ]
Dos enfoques son particularmente populares dentro de la comunidad de acoplamiento molecular. Un enfoque utiliza una técnica de emparejamiento que describe la proteína y el ligando como superficies complementarias. [6] [7] [8] El segundo enfoque simula el proceso de acoplamiento real en el que se calculan las energías de interacción ligando-proteína por pares. [9] Ambos enfoques tienen ventajas significativas, así como algunas limitaciones. Estos se describen a continuación.
Dar forma a la complementariedad [ editar ]
Los métodos de complementariedad de forma / coincidencia geométrica describen la proteína y el ligando como un conjunto de características que los hacen acoplables. [10] Estas características pueden incluir descriptores de superficie molecular / superficie complementaria . En este caso, la superficie molecular del receptor se describe en términos de su área de superficie accesible al disolvente.y la superficie molecular del ligando se describe en términos de su descripción de superficie coincidente. La complementariedad entre las dos superficies equivale a la descripción de coincidencia de formas que puede ayudar a encontrar la postura complementaria de acoplar el objetivo y las moléculas de ligando. Otro enfoque es describir las características hidrófobas de la proteína utilizando giros en los átomos de la cadena principal. Otro enfoque más consiste en utilizar una técnica de descriptor de forma de Fourier. [11] [12] [13]Mientras que los enfoques basados en la complementariedad de formas son típicamente rápidos y robustos, normalmente no pueden modelar los movimientos o cambios dinámicos en las conformaciones de ligando / proteína con precisión, aunque los desarrollos recientes permiten que estos métodos investiguen la flexibilidad del ligando. Los métodos de complementariedad de formas pueden escanear rápidamente a través de varios miles de ligandos en cuestión de segundos y realmente averiguar si pueden unirse al sitio activo de la proteína y, por lo general, son escalables incluso para interacciones proteína-proteína. También son mucho más adecuados para enfoques basados en farmacóforos, ya que utilizan descripciones geométricas de los ligandos para encontrar la unión óptima.
Simulación [ editar ]
Simular el proceso de acoplamiento es mucho más complicado. En este enfoque, la proteína y el ligando están separados por una cierta distancia física, y el ligando encuentra su posición en el sitio activo de la proteína después de un cierto número de "movimientos" en su espacio conformacional. Los movimientos incorporan transformaciones de cuerpos rígidos, como traslaciones y rotaciones, así como cambios internos en la estructura del ligando, incluidas las rotaciones del ángulo de torsión. Cada uno de estos movimientos en el espacio de conformación del ligando induce un costo energético total del sistema. Por tanto, la energía total del sistema se calcula después de cada movimiento.
La ventaja obvia de la simulación de acoplamiento es que la flexibilidad del ligando se incorpora fácilmente, mientras que las técnicas de complementariedad de forma deben utilizar métodos ingeniosos para incorporar flexibilidad en los ligandos. Además, modela con mayor precisión la realidad, mientras que las técnicas complementarias de formas son más una abstracción.
Claramente, la simulación es computacionalmente costosa, ya que tiene que explorar un gran panorama energético. Las técnicas basadas en cuadrículas, los métodos de optimización y el aumento de la velocidad de la computadora han hecho que la simulación de acoplamiento sea más realista.
Mecánica de acoplamiento [ editar ]
Para realizar una pantalla de acoplamiento, el primer requisito es una estructura de la proteína de interés. Por lo general, la estructura se ha determinado utilizando una técnica biofísica como la cristalografía de rayos X , la espectroscopia de RMN o la microscopía crioelectrónica (crio-EM) , pero también puede derivarse de la construcción de modelos de homología . Esta estructura de proteína y una base de datos de ligandos potenciales sirven como entradas para un programa de acoplamiento. El éxito de un programa de acoplamiento depende de dos componentes: el algoritmo de búsqueda y la función de puntuación .
Algoritmo de búsqueda [ editar ]
El espacio de búsqueda en teoría consiste en todas las posibles orientaciones y conformaciones de la proteína emparejadas con el ligando. Sin embargo, en la práctica con los recursos computacionales actuales, es imposible explorar exhaustivamente el espacio de búsqueda; esto implicaría enumerar todas las posibles distorsiones de cada molécula (las moléculas son dinámicas y existen en un conjunto de estados conformacionales) y todas las posibles orientaciones rotacionales y traslacionales de el ligando relativo a la proteína en un nivel dado de granularidad . La mayoría de los programas de acoplamiento en uso tienen en cuenta todo el espacio conformacional del ligando (ligando flexible) y varios intentan modelar un receptor de proteína flexible. Cada "instantánea" del par se denominapose .
Se han aplicado una variedad de estrategias de búsqueda conformacional al ligando y al receptor. Éstos incluyen:
- Búsquedas torsionales sistemáticas o estocásticas sobre enlaces rotativos
- simulaciones de dinámica molecular
- algoritmos genéticos para "evolucionar" nuevas conformaciones de baja energía y donde la puntuación de cada pose actúa como la función de aptitud utilizada para seleccionar individuos para la siguiente iteración.
Flexibilidad de ligando [ editar ]
Las conformaciones del ligando pueden generarse en ausencia del receptor y posteriormente acoplarse [14] o pueden generarse conformaciones sobre la marcha en presencia de la cavidad de unión al receptor, [15] o con total flexibilidad de rotación de cada ángulo diedro. utilizando un acoplamiento basado en fragmentos. [16] La evaluación de la energía del campo de fuerza se utiliza con mayor frecuencia para seleccionar conformaciones energéticamente razonables, [17] pero también se han utilizado métodos basados en el conocimiento. [18]
Los péptidos son moléculas altamente flexibles y de tamaño relativamente grande, lo que hace que modelar su flexibilidad sea una tarea desafiante. Se desarrollaron varios métodos para permitir el modelado eficiente de la flexibilidad de los péptidos durante el acoplamiento proteína-péptido. [19]
Flexibilidad del receptor [ editar ]
La capacidad computacional ha aumentado drásticamente durante la última década haciendo posible el uso de métodos más sofisticados y computacionalmente intensivos en el diseño de fármacos asistido por computadora. Sin embargo, lidiar con la flexibilidad del receptor en las metodologías de acoplamiento sigue siendo un tema espinoso. [20] La principal razón de esta dificultad es la gran cantidad de grados de libertad que hay que tener en cuenta en este tipo de cálculos. Sin embargo, ignorarlo en algunos de los casos puede conducir a resultados de acoplamiento deficientes en términos de predicción de pose de unión. [21]
A menudo se utilizan múltiples estructuras estáticas determinadas experimentalmente para la misma proteína en diferentes conformaciones para emular la flexibilidad del receptor. [22] Alternativamente, se pueden buscar bibliotecas de rotámeros de cadenas laterales de aminoácidos que rodean la cavidad de unión para generar conformaciones de proteínas alternativas pero energéticamente razonables. [23] [24]
Función de puntuación [ editar ]
Los programas de acoplamiento generan una gran cantidad de posibles poses de ligando, de las cuales algunas pueden ser rechazadas inmediatamente debido a choques con la proteína. El resto se evalúa utilizando alguna función de puntuación, que toma una pose como entrada y devuelve un número que indica la probabilidad de que la pose represente una interacción de unión favorable y clasifique un ligando en relación con otro.
La mayoría de las funciones de puntuación son campos de fuerza de la mecánica molecular basados en la física que estiman la energía de la pose dentro del sitio de unión. Las diversas contribuciones a la unión se pueden escribir como una ecuación aditiva:
Los componentes consisten en efectos solventes, cambios conformacionales en la proteína y el ligando, energía libre debido a interacciones proteína-ligando, rotaciones internas, energía de asociación del ligando y receptor para formar un solo complejo y energía libre debido a cambios en los modos vibracionales. [25] Una energía baja (negativa) indica un sistema estable y, por lo tanto, una probable interacción vinculante.
Un enfoque alternativo es derivar un potencial estadístico basado en el conocimiento para las interacciones a partir de una gran base de datos de complejos proteína-ligando, como el Protein Data Bank , y evaluar el ajuste de la pose de acuerdo con este potencial inferido.
Hay un gran número de estructuras de cristalografía de rayos X para complejos entre proteínas y ligandos de alta afinidad, pero comparativamente menos para ligandos de baja afinidad, ya que los complejos posteriores tienden a ser menos estables y, por lo tanto, más difíciles de cristalizar. Las funciones de puntuación entrenadas con estos datos pueden acoplar ligandos de alta afinidad correctamente, pero también darán conformaciones acopladas plausibles para ligandos que no se unen. Esto da una gran cantidad de resultados positivos falsos , es decir, ligandos que se predice que se unirán a la proteína que en realidad no lo hacen cuando se colocan juntos en un tubo de ensayo.
Una forma de reducir el número de falsos positivos es recalcular la energía de las posturas de puntuación más alta utilizando técnicas (potencialmente) más precisas pero computacionalmente más intensivas, como los métodos Generalized Born o Poisson-Boltzmann . [9]
Evaluación de atraque [ editar ]
La interdependencia entre la función de muestreo y la puntuación afecta la capacidad de acoplamiento para predecir poses plausibles o afinidades de unión para compuestos novedosos. Por lo tanto, generalmente se requiere una evaluación de un protocolo de acoplamiento (cuando hay datos experimentales disponibles) para determinar su capacidad predictiva. La evaluación de atraque se puede realizar utilizando diferentes estrategias, tales como:
- cálculo de precisión de atraque (DA);
- la correlación entre una puntuación de acoplamiento y la respuesta experimental o determinación del factor de enriquecimiento (EF); [26]
- la distancia entre un resto de unión a iones y el ión en el sitio activo;
- la presencia de modelos de ajuste inducido.
Precisión de acoplamiento [ editar ]
La precisión de acoplamiento [27] [28] representa una medida para cuantificar la aptitud de un programa de acoplamiento racionalizando la capacidad de predecir la postura correcta de un ligando con respecto a la observada experimentalmente. [29]
Factor de enriquecimiento [ editar ]
Las pantallas de acoplamiento también pueden evaluarse mediante el enriquecimiento de ligandos anotados de aglutinantes conocidos de entre una gran base de datos de moléculas " señuelo " supuestamente no vinculantes . [26] De esta manera, el éxito de una pantalla de acoplamiento se evalúa por su capacidad para enriquecer el pequeño número de compuestos activos conocidos en los rangos superiores de una pantalla entre un número mucho mayor de moléculas señuelo en la base de datos. El área bajo la curva de característica operativa del receptor (ROC) se usa ampliamente para evaluar su desempeño.
Prospectivo [ editar ]
Los aciertos resultantes de las pantallas de acoplamiento se someten a validación farmacológica (por ejemplo, IC 50 , mediciones de afinidad o potencia ). Solo los estudios prospectivos constituyen una prueba concluyente de la idoneidad de una técnica para un objetivo en particular. [30]
Evaluación comparativa [ editar ]
El potencial de los programas de acoplamiento para reproducir modos de unión determinados por cristalografía de rayos X puede evaluarse mediante una variedad de conjuntos de referencia de acoplamiento.
Para moléculas pequeñas, existen varios conjuntos de datos de referencia para acoplamiento y cribado virtual, por ejemplo, Astex Diverse Set que consta de estructuras cristalinas de rayos X de ligando de proteína de alta calidad [31] o el Directorio de señuelos útiles (DUD) para la evaluación del rendimiento del cribado virtual. [26]
Una evaluación de los programas de acoplamiento para determinar su potencial para reproducir modos de unión de péptidos se puede evaluar mediante Lecciones para la evaluación de la eficiencia del acoplamiento y la puntuación (LEADS-PEP). [32]
Aplicaciones [ editar ]
Una interacción de unión entre un ligando de molécula pequeña y una proteína enzimática puede resultar en la activación o inhibición de la enzima. Si la proteína es un receptor, la unión del ligando puede resultar en agonismo o antagonismo . El acoplamiento se usa más comúnmente en el campo del diseño de fármacos ; la mayoría de los medicamentos son moléculas orgánicas pequeñas y el acoplamiento se puede aplicar a:
- identificación de aciertos: el acoplamiento combinado con una función de puntuación se puede utilizar para seleccionar rápidamente grandes bases de datos de fármacos potenciales in silico para identificar moléculas que probablemente se unan a la proteína objetivo de interés (ver exploración virtual ).
- optimización de prospectos: el acoplamiento se puede usar para predecir dónde y en qué orientación relativa un ligando se une a una proteína (también conocido como el modo de unión o pose). Esta información, a su vez, puede usarse para diseñar análogos más potentes y selectivos.
- Biorremediación : el acoplamiento de ligandos de proteínas también se puede utilizar para predecir los contaminantes que pueden ser degradados por las enzimas. [33] [34]
Ver también [ editar ]
- Diseño de fármacos
- Algoritmo de Katchalski-Katzir
- Lista de sistemas de gráficos moleculares
- Acoplamiento macromolecular
- Mecánica molecular
- Estructura proteica
- Diseño de proteínas
- Software para modelado de mecánica molecular
- Lista de software de acoplamiento proteína-ligando
- Software de diseño molecular
- Docking @ Home
- Ibercivis
- Base de datos ZINC
- Buscador de prospectos
- Proyección virtual
- Funciones de puntuación para el acoplamiento
Referencias [ editar ]
- ^ Lengauer T, Rarey M (junio de 1996). "Métodos computacionales para el acoplamiento biomolecular". Opinión actual en biología estructural . 6 (3): 402–6. doi : 10.1016 / S0959-440X (96) 80061-3 . PMID 8804827 .
- ^ Cocina DB, Decornez H, Furr JR, Bajorath J (noviembre de 2004). "Acoplamiento y puntuación en el cribado virtual para el descubrimiento de fármacos: métodos y aplicaciones". Reseñas de la naturaleza. Descubrimiento de drogas . 3 (11): 935–49. doi : 10.1038 / nrd1549 . PMID 15520816 . S2CID 1069493 .
- ^ Mostashari-Rad, T; Arian, R; Mehridehnavi, A; Fassihi, A; Ghasemi, F (13 de junio de 2019). "Estudio de los inhibidores del receptor de quimiocinas CXCR4 utilizando metodologías de acoplamiento molecular y QSPR". Revista de Química Teórica y Computacional . 178 (4). doi : 10.1142 / S0219633619500184 .
- ^ Jorgensen WL (noviembre de 1991). "Oxidación del modelo de cerradura y llave para unión proteína-ligando". Ciencia . 254 (5034): 954–5. Código Bibliográfico : 1991Sci ... 254..954J . doi : 10.1126 / science.1719636 . PMID 1719636 .
- ^ Wei BQ, Weaver LH, Ferrari AM, Matthews BW, Shoichet BK (abril de 2004). "Prueba de un algoritmo de acoplamiento de receptor flexible en un sitio de enlace modelo". Revista de Biología Molecular . 337 (5): 1161–82. doi : 10.1016 / j.jmb.2004.02.015 . PMID 15046985 .
- ^ Goldman BB, Wipke WT (2000). "Descriptores de forma cuadrática QSD. 2. Acoplamiento molecular usando descriptores de forma cuadrática (QSDock)" . Las proteínas . 38 (1): 79–94. doi : 10.1002 / (SICI) 1097-0134 (20000101) 38: 1 <79 :: AID-PROT9> 3.0.CO; 2-U . PMID 10651041 .
- ^ Meng EC, Shoichet BK, Kuntz ID (1992). "Acoplamiento automatizado con evaluación energética basada en red". Revista de Química Computacional . 13 (4): 505–524. doi : 10.1002 / jcc.540130412 . S2CID 97778840 .
- ^ Morris GM, Goodsell DS, Halliday RS, Huey R, Hart WE, Belew RK, Olson AJ (1998). "Acoplamiento automatizado utilizando un algoritmo genético lamarckiano y una función empírica de energía libre de unión". Revista de Química Computacional . 19 (14): 1639-1662. CiteSeerX 10.1.1.471.5900 . doi : 10.1002 / (SICI) 1096-987X (19981115) 19:14 <1639 :: AID-JCC10> 3.0.CO; 2-B .
- ^ a b Feig M, Onufriev A, Lee MS, Im W, Case DA, Brooks CL (enero de 2004). "Comparación de rendimiento de métodos nacidos y Poisson generalizados en el cálculo de energías de solvatación electrostática para estructuras de proteínas". Revista de Química Computacional . 25 (2): 265–84. doi : 10.1002 / jcc.10378 . PMID 14648625 . S2CID 3191066 .
- ^ Shoichet BK, Identificación de Kuntz, Bodian DL (2004). "Acoplamiento molecular mediante descriptores de forma". Revista de Química Computacional . 13 (3): 380–397. doi : 10.1002 / jcc.540130311 . S2CID 42749294 .
- ^ Cai W, Shao X, Maigret B (enero de 2002). "Reconocimiento de proteína-ligando utilizando superficies moleculares armónicas esféricas: hacia un filtro rápido y eficiente para la detección de gran rendimiento virtual". Revista de modelado y gráficos moleculares . 20 (4): 313-28. doi : 10.1016 / S1093-3263 (01) 00134-6 . PMID 11858640 .
- ^ Morris RJ, Najmanovich RJ, Kahraman A, Thornton JM (mayo de 2005). "Coeficientes de expansión armónica esférica real como descriptores de forma 3D para comparaciones de ligando y bolsillo de unión a proteínas" . Bioinformática . 21 (10): 2347–55. doi : 10.1093 / bioinformática / bti337 . PMID 15728116 .
- ^ Kahraman A, Morris RJ, Laskowski RA, Thornton JM (abril de 2007). "Variación de forma en los bolsillos de unión a proteínas y sus ligandos". Revista de Biología Molecular . 368 (1): 283-301. doi : 10.1016 / j.jmb.2007.01.086 . PMID 17337005 .
- ^ Kearsley SK, Underwood DJ, Sheridan RP, Miller MD (octubre de 1994). "Flexibases: una forma de mejorar el uso de métodos de acoplamiento molecular". Revista de diseño molecular asistido por computadora . 8 (5): 565–82. Código bibliográfico : 1994JCAMD ... 8..565K . doi : 10.1007 / BF00123666 . PMID 7876901 . S2CID 8834526 .
- ^ Friesner RA, Banks JL, Murphy RB, Halgren TA, Klicic JJ, Mainz DT, Repasky MP, Knoll EH, Shelley M, Perry JK, Shaw DE, Francis P, Shenkin PS (marzo de 2004). "Glide: un nuevo enfoque para un atraque y puntuación rápidos y precisos. 1. Método y evaluación de la precisión del atraque". Revista de Química Medicinal . 47 (7): 1739–49. doi : 10.1021 / jm0306430 . PMID 15027865 .
- ^ Zsoldos Z, Reid D, Simon A, Sadjad SB, Johnson AP (julio de 2007). "eHiTS: un nuevo sistema de acoplamiento de ligando flexible, rápido y exhaustivo". Revista de modelado y gráficos moleculares . 26 (1): 198–212. doi : 10.1016 / j.jmgm.2006.06.002 . PMID 16860582 .
- ^ Wang Q, Pang YP (septiembre de 2007). Romesberg F (ed.). "Preferencia de moléculas pequeñas por conformaciones mínimas locales al unirse a proteínas" . PLOS ONE . 2 (9): e820. Código bibliográfico : 2007PLoSO ... 2..820W . doi : 10.1371 / journal.pone.0000820 . PMC 1959118 . PMID 17786192 .
- ^ Klebe G, Mietzner T (octubre de 1994). "Un método rápido y eficaz para generar conformaciones biológicamente relevantes". Revista de diseño molecular asistido por computadora . 8 (5): 583–606. Código bibliográfico : 1994JCAMD ... 8..583K . doi : 10.1007 / BF00123667 . PMID 7876902 . S2CID 206768542 .
- ^ Ciemny M, Kurcinski M, Kamel K, Kolinski A, Alam N, Schueler-Furman O, Kmiecik S (mayo de 2018). "Acoplamiento proteína-péptido: oportunidades y desafíos" . Descubrimiento de drogas hoy . 23 (8): 1530-1537. doi : 10.1016 / j.drudis.2018.05.006 . PMID 29733895 .
- ^ Antunes DA, Devaurs D, Kavraki LE (diciembre de 2015). "Comprender los desafíos de la flexibilidad de las proteínas en el diseño de fármacos" (PDF) . Opinión de expertos sobre el descubrimiento de fármacos . 10 (12): 1301-13. doi : 10.1517 / 17460441.2015.1094458 . hdl : 1911/88215 . PMID 26414598 . S2CID 6589810 .
- ^ Cerqueira NM, Bras NF, Fernandes PA, Ramos MJ (enero de 2009). "MADAMM: un acoplamiento multietapa con un protocolo de modelado molecular automatizado". Las proteínas . 74 (1): 192-206. doi : 10.1002 / prot.22146 . PMID 18618708 . S2CID 36656063 .
- ^ Totrov M, Abagyan R (abril de 2008). "Acoplamiento de ligando flexible a múltiples conformaciones de receptor: una alternativa práctica" . Opinión actual en biología estructural . 18 (2): 178–84. doi : 10.1016 / j.sbi.2008.01.004 . PMC 2396190 . PMID 18302984 .
- ^ Hartmann C, Antes I, Lengauer T (febrero de 2009). "Acoplamiento y puntuación con conformaciones alternativas de cadena lateral". Las proteínas . 74 (3): 712-26. doi : 10.1002 / prot.22189 . PMID 18704939 . S2CID 36088213 .
- ^ Taylor RD, Judiosbury PJ, Essex JW (octubre de 2003). "FDS: acoplamiento flexible de ligando y receptor con un modelo de disolvente continuo y función de energía de núcleo blando". Revista de Química Computacional . 24 (13): 1637–56. CiteSeerX 10.1.1.147.1131 . doi : 10.1002 / jcc.10295 . PMID 12926007 . S2CID 15814316 .
- ^ Murcko MA (diciembre de 1995). "Métodos computacionales para predecir la energía libre de unión en complejos ligando-receptor". Revista de Química Medicinal . 38 (26): 4953–67. doi : 10.1021 / jm00026a001 . PMID 8544170 .
- ↑ a b c Huang N, Shoichet BK, Irwin JJ (noviembre de 2006). "Conjuntos de evaluación comparativa para acoplamiento molecular" . Revista de Química Medicinal . 49 (23): 6789–801. doi : 10.1021 / jm0608356 . PMC 3383317 . PMID 17154509 .
- ^ Ballante F, Marshall GR (enero de 2016). "Una estrategia automatizada para la selección de pose de unión y evaluación de acoplamiento en el diseño de fármacos basado en estructura". Revista de información química y modelado . 56 (1): 54–72. doi : 10.1021 / acs.jcim.5b00603 . PMID 26682916 .
- ^ Bursulaya BD, Totrov M, Abagyan R, Brooks CL (noviembre de 2003). "Estudio comparativo de varios algoritmos para el acoplamiento flexible de ligandos". Revista de diseño molecular asistido por computadora . 17 (11): 755–63. Código bibliográfico : 2003JCAMD..17..755B . doi : 10.1023 / B: JCAM.0000017496.76572.6f . PMID 15072435 . S2CID 12569345 .
- ^ Ballante, Flavio (2018). "Acoplamiento proteína-ligando en el diseño de fármacos: evaluación del rendimiento y selección de la pose de unión". Diseño racional de fármacos . Métodos en Biología Molecular. 1824 . págs. 67–88. doi : 10.1007 / 978-1-4939-8630-9_5 . ISBN 978-1-4939-8629-3. ISSN 1940-6029 . PMID 30039402 .
- ↑ Irwin JJ (14 de febrero de 2008). "Puntos de referencia comunitarios para el cribado virtual". Revista de diseño molecular asistido por computadora . 22 (3–4): 193–9. Código Bibliográfico : 2008JCAMD..22..193I . doi : 10.1007 / s10822-008-9189-4 . PMID 18273555 . S2CID 26260725 .
- ^ Hartshorn MJ, Verdonk ML, Chessari G, Brewerton SC, Mooij WT, Mortenson PN, Murray CW (febrero de 2007). "Conjunto de pruebas diverso y de alta calidad para la validación del rendimiento de acoplamiento proteína-ligando". Revista de Química Medicinal . 50 (4): 726–41. doi : 10.1021 / jm061277y . PMID 17300160 .
- ^ Hauser AS, Windshügel B (diciembre de 2015). "Un conjunto de datos de referencia para la evaluación del rendimiento de acoplamiento de péptidos". Revista de información química y modelado . 56 (1): 188–200. doi : 10.1021 / acs.jcim.5b00234 . PMID 26651532 .
- ^ Suresh PS, Kumar A, Kumar R, Singh VP (enero de 2008). "Un enfoque in silico [corrección de insilico] para la biorremediación: lacasa como estudio de caso". Revista de modelado y gráficos moleculares . 26 (5): 845–9. doi : 10.1016 / j.jmgm.2007.05.005 . PMID 17606396 .
- ^ Basharat Z, Yasmin A, Bibi M (2020). "Implicaciones del ensayo de acoplamiento molecular para la biorremediación". Análisis de datos en medicina: conceptos, metodologías, herramientas y aplicaciones . IGI Global. págs. 1556-1577. ISBN 978-1799812043.
Enlaces externos [ editar ]
- Bikadi Z, Kovacs S, Demko L, Hazai E. "Molecular Docking Server - Ligand Protein Docking & Molecular Modelling" . Virtua Drogas Ltd . Consultado el 15 de julio de 2008 .
Servicio de Internet que calcula el sitio, la geometría y la energía de pequeñas moléculas que interactúan con las proteínas.
- Malinauskas T. "Instalación paso a paso de MGLTools 1.5.2 (AutoDockTools, Python Molecular Viewer y Visual Programming Environment) en Ubuntu Linux 8.04" . Archivado desde el original el 26 de febrero de 2009 . Consultado el 15 de julio de 2008 .
- Proyecto Docking @ GRID de muestreo conformacional y acoplamiento en cuadrículas: un objetivo es implementar algunos algoritmos de acoplamiento distribuidos intrínsecos en cuadrículas computacionales, descargue la versión de Linux de código abierto Docking @ GRID
- Click2Drug.org - Directorio de herramientas de diseño de fármacos computacionales.
- Ligando: Acoplamiento de receptor con MOE (entorno operativo molecular)