| Enterobacter cloacae | |
|---|---|
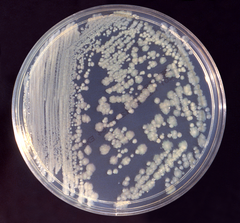 | |
| Enterobacter cloacae en agar tríptico de soja . | |
| clasificación cientifica | |
| Reino: | |
| Filo: | |
| Clase: | |
| Pedido: | |
| Familia: | |
| Género: | |
| Nombre binomial | |
| Enterobacter cloacae (Jordania 1890) Hormaeche y Edwards 1960 | |
| Subespecie | |
E. c. subsp. cloacae | |
| Sinónimos | |
Bacillus cloacae Jordan 1890 | |
Enterobacter cloacae es una clínicamente significativa Gram-negativas , facultativamente anaeróbico , en forma de barra bacteria .
Microbiología [ editar ]
En los laboratorios de microbiología, E. cloacae se cultiva frecuentemente a 30 ° C en agar nutritivo oa 35 ° C en caldo de soja tríptico . [1] Es una bacteria gramnegativa con forma de bastón, es facultativamente anaeróbica y tiene flagelos peritricosos . Es oxidasa negativa y catalasa positiva . [2]
Uso industrial [ editar ]
Enterobacter cloacae se ha utilizado en un método basado en biorreactores para la biodegradación de explosivos y en el control biológico de enfermedades de las plantas. [3]
Seguridad [ editar ]
E. cloacae se considera un organismo de nivel 1 de bioseguridad en los Estados Unidos y de nivel 2 en Canadá. [ cita requerida ]
Genómica [ editar ]
Un borrador de la secuencia del genoma de Enterobacter cloacae subsp. cloacae se anunció en 2012. Las bacterias utilizadas en el estudio se aislaron de las heces del panda gigante . [4]
Importancia clínica [ editar ]
Enterobacter cloacae es un miembro de la flora intestinal normal de muchos seres humanos y no suele ser un patógeno primario. [5] Algunas cepas se han asociado con infecciones del tracto urinario y respiratorio en individuos inmunodeprimidos. Se ha informado de tratamiento con cefepima y gentamicina . [6]
Un estudio de 2012 en el que Enterobacter cloacae trasplantado en ratones previamente libres de gérmenes resultó en un aumento de la obesidad en comparación con ratones libres de gérmenes alimentados con una dieta idéntica, lo que sugiere un vínculo entre la obesidad y la presencia de la flora intestinal de Enterobacter. [7]
Especies del complejo E. cloacae [ editar ]
E. cloacae fue descrita por primera vez en 1890 por Jordan [201] [ cita requerida ] como Bacillus cloacae , y luego sufrió numerosos cambios taxonómicos, convirtiéndose en 'Bacterium cloacae' en 1896 (Lehmann y Neumann), Cloaca cloacae en 1919 (Castellani y Chalmers), fue identificado como 'Aerobacter cloacae' en 1923 (Bergey et al.), Aerobacter cloacae en 1958 (Hormaeche y Edwards) y E. cloacae en 1960 (Hormaeche y Edwards), por lo que todavía se conoce hoy. [7] E. cloacaees omnipresente en ambientes terrestres y acuáticos (agua, alcantarillado, suelo y alimentos). Estas cepas ocurren como microflora comensal en el tracto intestinal de humanos y animales [1] y juegan un papel importante como patógenos en plantas e insectos. Esta diversidad de hábitats se refleja en la variedad genética de la especie E. cloacae . [6] La E. cloacae también es un patógeno nosocomial importante responsable de bacteriemia e infecciones del tracto respiratorio inferior, del tracto urinario e intraabdominales, así como de endocarditis. artritis séptica, osteomielitis e infecciones de piel y tejidos blandos. La piel y el tracto gastrointestinal son los sitios más comunes a través de los cuales se puede contraer E. cloacae . [1,29]
E. cloacae tiende a contaminar varios dispositivos médicos, intravenosos y otros dispositivos hospitalarios. Los brotes nosocomiales también se han asociado con la colonización de ciertos equipos quirúrgicos y soluciones de limpieza operativa. Otro reservorio potencial de bacteriemia nosocomial es la solución de heparina que se usa para irrigar ciertos dispositivos intravasculares de forma continua. Este líquido ha sido implicado como reservorio de brotes de bacteriemia asociada al dispositivo en varios casos. [30]
En los últimos años, E. cloacae se ha convertido en uno de los patógenos nosocomiales más comúnmente encontrados en las unidades neonatales, y se han notificado varios brotes de infección. [31] En 1998, van Nierop et al. reportaron un brote en una unidad de cuidados intensivos neonatales con nueve muertes, [32] y en 2003, Kuboyama et al. notificaron tres brotes con 42 infecciones sistémicas y una mortalidad del 34%. [33] Este microorganismo puede transmitirse a los recién nacidos a través de líquidos intravenosos contaminados, soluciones de nutrición parenteral total y equipo médico. Se han descrito muchos brotes de un solo clon, probablemente causados por transmisión cruzada a través de los trabajadores de la salud, lo que sugiere que los pacientes hospitalizados también pueden actuar como reservorio. [31] Las cepas tipo de la especie son E. cloacae ATCC 49162 y 13047. Esta última cepa es la primera secuencia completa del genoma de laE. cloacae especies y la cepa tipo es E. cloacae subsp. cloacae .
La subsp. Completa de E. cloacae . cloacae ATCC 13047 contiene un único cromosoma circular de 5.314.588 pb y dos plásmidos circulares, pECL_A y pECL_B, de 200.370 y 85.650 pb (números de acceso de GenBank CP001918, CP001919 y CP001920, respectivamente). [34]
Los otros genomas de E. cloacae que se han secuenciado se depositan en GenBank con los números de acceso CP002272, CP002886, FP929040 y AGSY00000000.
E. asburiae lleva el nombre de Mary Alyce Fife-Asbury, una bacterióloga estadounidense que hizo muchas contribuciones importantes a la clasificación de Enterobacteriaceae, particularmente en la descripción de nuevos serotipos de Klebsiella y Salmonella , [35-37] nuevos géneros y nuevas especies. [38-42 ] E. asburiae sp. nov. se describió en 1986 sobre la base del grupo entérico 17. [43] Este grupo se definió en 1978 como un grupo de cepas bioquímicamente similares aisladas de diferentes muestras humanas [44] y enviadas a los CDC. Antes de la designación de 'grupo entérico 17', estas cepas se habían informado como cepas de Citrobacter o Enterobacter no identificadas o atípicas. [44] Después de varios estudios, se demostró que estas cepas representan una sola nueva especie en el géneroEnterobacter , que se denominó E. asburiae .
Se han aislado cepas de E. asburiae del suelo e implicadas en la movilización de fosfato para la nutrición vegetal a partir del fosfato de calcio, pero la mayoría de las especies de E. asburiae se han aislado de fuentes humanas. La cepa tipo de la especie E. asburiae es ATCC 35953 y se aisló de los exudados de loquios de una mujer de 22 años en los EE. UU. [43] La única cepa secuenciada de E. asburiae es LF7a, que contiene un ADN circular (4.812.833 pb) y dos plásmidos circulares, pENTAS01 (166.725 pb) y pENTAS02 (32.574 pb), que fueron presentados por Lucas et al. en 2011 al Instituto Conjunto del Genoma del DOE de EE. UU. (CA, EE. UU.; números de acceso de GenBank CP003026.1, CP003027.1 y CP003028.1, respectivamente).
E. hormaechei lleva el nombre de Estenio Hormaeche, un microbiólogo uruguayo que (con PR Edwards) propuso y definió el género Enterobacter . [7] El nombre E. hormaechei antes se llamaba grupo entérico 75, que contenía 11 cepas que fueron enviadas a los CDC. para su identificación entre 1973 y 1984. Se recibieron doce cepas adicionales de 1985 a 1987, tres de las cuales eran aislados de sangre. E. hormaechei se describió por primera vez sobre la base de 23 aislamientos enviados a los CDC para su identificación. En ese momento, no se podían asignar a una especie porque dieron negativo en las pruebas de D-sorbitol y melibiosa y no se ajustaban al perfil bioquímico de ninguna especie de Enterobacter establecida. La especie E. hormaecheise propuso ser negativo para lactosa, D-sorbitol, rafinosa, melibiosa y esculina y 87% positivo para dulcitol. Estas especies fueron definidas originalmente por O'Hara et al. cuando se aisló un gran grupo de hibridación de organismos entéricos y se encontró que estaba asociado con infecciones del torrente sanguíneo. [10]
La cepa tipo de E. hormaechei es ATCC 49162 y se aisló del esputo de un hombre en California en 1977. [10] El proyecto de secuenciación de la escopeta de genoma completo se presentó en 2011 al Centro de secuenciación del genoma humano (TX, EE. UU.; Número de acceso de GenBank AFHR00000000).
E. hormaechei consta de tres subespecies diferentes: E. hormaechei subsp. oharae , E. hormaechei subsp. hormaechei y E. hormaechei subsp. steigerwaltii , que corresponde a los grupos genéticos VI, VII y VIII, respectivamente. [8] La diferenciación de estas subespecies se basa en sus propiedades particulares y pruebas bioquímicas. [11]
E. hormaechei se aísla comúnmente como un patógeno nosocomial de importancia clínica; [45,46] se ha informado en varios brotes de sepsis en unidades de cuidados intensivos neonatales en los EE.UU. [47] y en Brasil, donde el brote se originó por vía parenteral contaminada nutrición. [48]
E. kobei lleva el nombre de la ciudad de Kobe (Japón), donde se aisló la cepa tipo de esta especie. E. kobei fue descrito por primera vez por Kosako et al. basado en una colección de 23 cepas con los rasgos generales de E. cloacae y la diferencia fenotípica común de ser Voges-Proskauer-negativo. [49] El nombre E. kobei se propone para un grupo de organismos denominados grupo 21 de los NIH en los NIH de Tokio. Más tarde se descubrió que el grupo 21 de los NIH también se parecía al grupo entérico 69, [50] de los CDC y a E. kobeise comparó con este último. Sobre la base de la relación del ADN, ambos organismos podrían incluirse en un solo taxón. Sin embargo, el grupo entérico de los CDC 69 se describió como positivo en Voges-Proskauer y pigmentación amarilla, [50] mientras que todas las cepas de E. kobei fueron Voges-Proskauer- y pigmentación negativa. Estos hallazgos sugieren que la relación de ambos organismos es a nivel de subespecies o biogrupos. La cepa tipo de E. kobei es NIH 1485-1479 y se aisló mediante hemocultivo de un paciente diabético.
E. ludwigii , que lleva el nombre de Wolfgang Ludwig, un microbiólogo que trabaja en sistemática bacteriana [51] y que desarrolló las bases de datos del ARB y las hizo públicas [52]. Esta descripción se basa en los análisis filogenéticos de los datos de la secuencia parcial de hsp60 recopilados en un estudio genético de poblaciones [6], así como en ensayos de hibridación ADN-ADN y caracterizaciones fenotípicas.
La cepa tipo EN-119T se aisló de la orina del medio chorro de un paciente masculino de 18 años con una infección nosocomial del tracto urinario mientras estaba hospitalizado en el Hospital Universitario Grosshadern de Múnich, Alemania. El número de acceso de GenBank del rDNA 16S de la cepa EN-119T es AJ853891. [12]
E. nimipressuralis La especie E. nimipressuralis fue definida originalmente por Brenner et al. y antes se llamaba Erwinia nimipressuralis , que se aisló de fuentes no clínicas (por ejemplo, olmos con una enfermedad llamada madera húmeda). [43] Erwinia nimipressuralis se incluyó en las listas aprobadas de nombres bacterianos en 1980. Este microorganismo es bioquímicamente similar a E. cloacae , pero es diferente para la producción de ácido de sacarosa y rafinosa, mientras que E. cloacae es positivo en estas pruebas. La cepa tipo de E. nimipressuralis es ATCC 9912 y está aislada del olmo Ulmus spp. en los Estados Unidos (número de acceso de GenBank AJ567900).
La cepa PR-4 de E. cloacae subespecie cloacae se aisló e identificó mediante la secuencia del gen de rDNA 16S con vista de árbol filogenético desde suelo cargado de explosivos por P Ravikumar (número de acceso de GenBank KP261383). [8]
E. cloacae SG208 identificado como un microorganismo predominante en cultivo mixto aislado de lodos petroquímicos, IOCL, Guwahati es responsable de la degradación del benceno fue informado por Padhi y Gokhale (2016) [137].
Ver también [ editar ]
- Biohidrógeno
Referencias [ editar ]
- ^ Dalben M, Varkulja G, Basso M, Krebs VL, Gibelli MA, van der Heijden I, Rossi F, Duboc G, Levin AS, Costa SF (septiembre de 2008). "Investigación de un brote de Enterobacter cloacae en una unidad neonatal y revisión de la literatura". The Journal of Hospital Infection . 70 (1): 7–14. doi : 10.1016 / j.jhin.2008.05.003 . PMID 18632183 .
- ^ "Prueba bioquímica e identificación de Enterobacter cloacae" . microbiologyinfo. 24 de mayo de 2017 . Consultado el 26 de julio de 2017 .
- ^ Pudge IB, Daugulis AJ, Dubois C (2003). "El uso de Enterobacter cloacae ATCC 43560 en el desarrollo de un biorreactor de partición de dos fases para la destrucción de hexahidro-1,3,5-trinitro-1,3,5-s-triazina (RDX)". Revista de Biotecnología . 100 (1): 65–75. doi : 10.1016 / s0168-1656 (02) 00229-8 . PMID 12413787 .
- ^ Yan, Y; Zhao, CW; Zhang, YZ; Zhang, ZH; Pan, GL; Liu, WW; Ma, QY; Hora; Tan, XM (diciembre de 2012). "Proyecto de secuencia del genoma de Enterobacter cloacae subsp. Cloacae cepa 08XA1, una bacteria fecal de pandas gigantes" . Revista de bacteriología . 194 (24): 6928–9. doi : 10.1128 / JB.01790-12 . PMC 3510591 . PMID 23209197 .
- ^ Keller, R; Pedroso, MZ; Ritchmann, R; Silva, RM (febrero de 1998). "Aparición de propiedades asociadas a la virulencia en Enterobacter cloacae" . Infección e inmunidad . 66 (2): 645–9. doi : 10.1128 / IAI.66.2.645-649.1998 . PMC 113501 . PMID 9453621 .
- ^ Barnes BJ, Wiederhold NP, Micek ST, Polaco LB, Ritchie DJ (abril de 2003). "Ventriculitis por Enterobacter cloacae tratada con éxito con cefepima y gentamicina: reporte de caso y revisión de la literatura". Farmacoterapia . 23 (4): 537–42. doi : 10.1592 / phco.23.4.537.32126 . PMID 12680484 .
- ^ Na Fei; Liping Zhao (13 de diciembre de 2012). "Un patógeno oportunista aislado del intestino de un ser humano obeso causa obesidad en ratones libres de gérmenes" . El diario ISME . 7 (4): 880–884. doi : 10.1038 / ismej.2012.153 . PMC 3603399 . PMID 23235292 .
- ^ Ravikumar. P. GenBank Nuevo holotipo para Enterobacter cloacae subsp. cloacae cepa PR-4 aislada e identificada por la secuencia del gen 16S rDNA con vista de árbol filogenético, de suelo cargado de explosivos. Int J. de Res en Ingeniería y Ciencia, (6) 5 2016.
- Sanders WE Jr, Sanders CC. Enterobacter spp .: patógenos a punto de florecer en el cambio de siglo. Revisiones de microbiología clínica. 10, 220–241 (1997). Revisión exhaustiva del género Enterobacter que destaca las características microbiológicas, clínicas y epidemiológicas y la susceptibilidad a los antibióticos.
- Streit JM, Jones RN, Sader HS, Fritsche TR. Evaluación de la aparición de patógenos y perfiles de resistencia entre pacientes infectados en la unidad de cuidados intensivos: informe del Programa de Vigilancia Antimicrobiana SENTRY (Norteamérica, 2001). Revista Internacional de Agentes Antimicrobianos. 24, 111-118 (2004).
- Hidron AI, Edwards JR, Patel J et al .; para el equipo de la red nacional de seguridad sanitaria y las instalaciones de la red nacional de seguridad sanitaria participantes. Patógenos resistentes a los antimicrobianos asociados con infecciones asociadas a la atención médica: resumen anual de los datos notificados a la Red Nacional de Seguridad en la Atención Médica de los Centros para el Control y la Prevención de Enfermedades, 2006–2007. Control de Infecciones y Epidemiología Hospitalaria. 29 (11), 996–1011 (2008).
- Wisplinghoff H, Bischoff T, Tallent SM y col. Infecciones del torrente sanguíneo nosocomiales en hospitales de EE. UU .: análisis de 24.179 casos de un estudio de vigilancia prospectivo a nivel nacional. Enfermedades Clínicas Infecciosas. 39, 309–317 (2004).
- Paauw A, Caspers MP, Schuren FH y col. Diversidad genómica dentro del complejo Enterobacter cloacae. PLoS One3, e3018 (2008). Utilización de cuatro enfoques genéticos para discriminar dentro del complejo Enterobacter cloacae.
- Hoffmann H, Roggenkamp A. Genética de poblaciones de la especie Enterobacter cloacae. Microbiología ambiental aplicada. 69, 5306–5318 (2003). Agrupación genética que destaca las características taxonómicas y epidemiológicas del complejo E. cloacae.
- Hormaeche E, Edwards PR. Un género propuesto Enterobacter. Boletín Internacional de Nomenclatura y Taxonomía Bacteriológicas. 10, 71–74 (1960).
- Morand PC, Billoet A, Rottman M et al. Distribución específica dentro del complejo de Enterobacter cloacae de cepas aisladas de implantes ortopédicos infectados. Revista de microbiología clínica. 47 (8), 2489–2495 (2009).
- Wang GF, Xie GL, Zhu B y col. Identificación y caracterización del complejo Enterobacter que causa la enfermedad del marchitamiento de la morera (Morus alba) en China. Revista europea de fitopatología. 126, 465–478 (2010).
- O'Hara CM, Steigerwalt AG, Hill BC, Granjero JJ III, Fanning GG, Brenner DJ. Enterobacter hormaechei, una nueva especie de la familia Enterobacteriaceae anteriormente conocida como grupo entérico 75. Journal of Clinical Microbiology. 27, 2046-2049 (1989).
- Hoffmann H, Stindl S, Ludwig W y col. Enterobacter hormaechei subsp. oharae subsp. nov., E. hormaechei subsp. peine de hormaechei. nov. y E. hormaechei subsp. steigerwaltii subsp. nov., tres nuevas subespecies de importancia clínica Journal of Clinical Microbiology. 43, 3297–3303 (2005).
- Hoffmann H, Stindl S, Stumpf A y col. Descripción de Enterobacter ludwigii sp. nov., una nueva especie de Enterobacter de relevancia clínica. Microbiología sistemática y aplicada. 28 (3), 206–212 (2005).
- Hoffmann H, Stindl S, Ludwig W y col. La reasignación de Enterobacter se disuelve a Enterobacter cloacae cuando la subespecie de E. cloacae se disuelve en peine. nov. y descripción modificada de Enterobacter asburiae y Enterobacter kobei. Microbiología sistemática y aplicada. 28 (3), 196–205 (2005).
- Mshana SE, Gerwing L, Minde M y col. Brote de una nueva Enterobacter sp. llevando bla CTX-M-15 en una unidad neonatal de un hospital de atención terciaria en Tanzania. Revista Internacional de Agentes Antimicrobianos. 38 (3), 265–269 (2011).
- Pavlovic M, Konrad R, Iwobi AN, Sing A, Busch U, Huber I.Un enfoque dual que emplea MALDI-TOF MS y PCR en tiempo real para la identificación rápida de especies dentro del complejo Enterobacter cloacae. Letras de Microbiología FEMS. 328, 46–53 (2012).
- Hoffmann H, Schmoldt S, Trqlzsch K et al. Urosepsis nosocomial causada por Enterobacter kobei con fenotipo aberrante. Microbiología diagnóstica y enfermedades infecciosas. 53, 143-147 (2005).
- Townsend SM, Hurrell E, Caubilla-Barron J, Loc-Carrillo C, Forsythe SJ. Caracterización de un brote nosocomial de Enterobacter hormaechei de betalactamasa de espectro extendido y otros Enterobacter hormaechei identificados erróneamente como Cronobacter (Enterobacter) sakazakii. Microbiología. 154, 3659–3667 (2008).
- Garaizar J, Kaufmann ME, Pitt TL. Comparación de ribotipado con métodos convencionales para la identificación de tipo de Enterobacter cloacae. Revista de microbiología clínica. 29, 1303-1307 (1991).
- Haertl R, Bandlow G. Huella digital epidemiológica de Enterobacter cloacae mediante análisis de endonucleasas de restricción de fragmentos pequeños y electroforesis en gel de campo pulsado de fragmentos de restricción genómicos. Revista de microbiología clínica. 31, 128-133 (1993).
- Williams JGK, Kubelick AR, Livak KJ, Rafalski JA, Tingey SV. Los polimorfismos de ADN amplificados por cebadores arbitrarios son marcadores genéticos útiles. Investigación de ácidos nucleicos. 18, 6531–6535 (1990).
- Stumpf AN, Roggenkamp A, Hoffmann H. Especificidad del consenso intergénico repetitivo enterobacteriano y reacción en cadena de la polimerasa palindrómica extragénica repetitiva para la detección de clonalidad dentro del complejo Enterobacter cloacae. Microbiología diagnóstica y enfermedades infecciosas. 53 (1), 9 a 16 (2005).
- Barnes AI, Ortiz C, Paraje MG, Balanzino LE, Albesa I. Purificación y caracterización de una citotoxina de Enterobacter cloacae. Revista Canadiense de Microbiología. 43 (8), 729–733 (1997).
- Stuber K, Frey J, Burnens AP, Kuhnert P. Detección de genes de secreción de tipo III como indicador general de virulencia bacteriana. Sondas moleculares y celulares. 17, 25–32 (2003).
- Krzyminska S, Mokracka J, Koczura R, Kaznowski A. Actividad citotóxica de aislados humanos de Enterobacter cloacae. Inmunología FEMS y Microbiología Médica. 56, 248-252 (2009).
- Krzyminska S, Koczura R, Mokracka J, Puton T, Kaznowski A. Los aislados del complejo Enterobacter cloacae inducen la apoptosis de las células epiteliales intestinales humanas. Patogenia microbiana. 49, 83–89 (2010).
- Olsén A, Arnqvist A, Hammar M, Normark S. Regulación ambiental de la producción de curli en Escherichia coli. Agentes infecciosos y enfermedades. 2 (4), 272-274 (1993).
- Zogaj X, Bokranz W, Nimtz M, Romling U. Producción de fimbrias de celulosa y curli por miembros de la familia Enterobacteriaceae aisladas del tracto gastrointestinal humano. Infección e inmunidad. 71 (7), 4151–4158 (2003).
- Kim SM, Lee HW, Choi YW y col. Participación de las fimbrias curli en la formación de biopelículas de Enterobacter cloacae. Revista de Microbiología. 50 (1), 175-178 (2012).
- Lee SO, Kim YS, Kim BN, Kim MN, Woo JH, Ryu J. Impacto del uso previo de antibióticos en el desarrollo de resistencia a cefalosporinas de espectro extendido en pacientes con bacteriemia por enterobacter. Revista europea de microbiología clínica y enfermedades infecciosas. 8, 577–581 (2002).
- Musil I, Jensen V, Schilling J, Ashdown B, Kent T. Infección por Enterobacter cloacae de un injerto de derivación femoral-poplíteo de politetrafluoroetileno expandido: reporte de un caso. Revista de informes de casos médicos. 9 (4), 131 (2010).
- Dalben M, Varkulja G, Basso M et al. Investigación de un brote de Enterobacter cloacae en una unidad neonatal y revisión de la literatura. Revista de infección hospitalaria. 70, 7 a 14 (2008).
- van Nierop WH, Duse AG, Stewart RG, Bilgeri YR, Koornhof HJ. Epidemiología molecular de un brote de Enterobacter cloacae en la unidad de cuidados intensivos neonatales de un hospital provincial en Gauteng, Sudáfrica. Journal of Clinical Microbiology 36, 3085-3087 (1998).
- Kuboyama RH, de Oliveira HB, Moretti-Branchini ML. Epidemiología molecular de la infección sistémica por Enterobacter cloacae en una unidad de cuidados intensivos neonatales de alto riesgo. Control de Infecciones y Epidemiología Hospitalaria. 24, 490–494 (2003).
- Ren Y, Ren Y, Zhou Z y col. Secuencia completa del genoma de Enterobacter cloacae subsp. cepa tipo cloacae ATCC 13047. Journal of Bacteriology. 192 (9), 2463–2464 (2010).
- Edwards PR, Fife MA. Tipos capsulares de Klebsiella. Revista de Enfermedades Infecciosas. 91, 92-104 (1952).
- Edwards PR, Fife MS. Once serotipos de Arizona no descritos aislados del hombre. Antonie Van Leeuwenhoek. 28, 402–404 (1962).
- Fife MA, McWhorter AC, Edwards PR. Diez nuevos serotipos de Arizona aislados de animales y productos alimenticios para animales. Antonie Van Leeuwenhoek. 28, 369-372 (1962).
- Manzano D, Rojo P, Zubero Z, Alvarez M, Santamaria JM, Cisterna R. [Bacteremia polimicrobiana por Enterobacter gergoviae y Candida albicans.] Enfermedades Infecciosas y Microbiología Clínica. 9, 186-187 (1991).
- Farmer JJ 3rd, Fanning GR, Davis BR et al. Escherichia fergusonii y Enterobacter taylorae, dos nuevas especies de Enterobacteriaceae aisladas de muestras clínicas. Revista de microbiología clínica. 21 (1), 77–81 (1985).
- Ewing WH, Fife MA. Peine de Enterobacter agglomerans (Beijerinck). nov. (la bacteria Herbicola-Lathyri). Revista Internacional de Bacteriología Sistemática. 22, 4-11 (1972).
- Granjero JJ III, Asbury MA, Hickman FW, Brenner DJ; Grupo de estudio de enterobacterias. Enterobacter sakazakii: una nueva especie de "Enterobacteriaceae" aislada de muestras clínicas. Revista Internacional de Bacteriología Sistemática. 30, 569–584 (1980).
- Grimont PAD, Grimont F, Granjero JJ III, Asbury MA. Cedecea davisae gen. nov., sp. nov. y Cedecea lapagei sp. nov., nuevas Enterobacteriaceae a partir de muestras clínicas. Revista Internacional de Bacteriología Sistemática. 31, 317–326 (1981).
- Brenner DJ, McWhorter AC, Kai A, Steigerwalt AG, Granjero JJ III. Enterobacter asburiae sp. nov., una nueva especie encontrada en muestras clínicas, y reasignación de Erwinia dissolvens y Erwinia nimipressuralis al género Enterobacter como Enterobacter dissolvens comb. nov. y Enterobacter nimipressuralis comb. nov. Revista de microbiología clínica. 23, 1114-1120 (1986).
- Granjero JJ III, Davis BR, Hickman-Brenner FW et al. Identificación bioquímica de nuevas especies y biogrupos de Enterobacteriaceae aisladas de muestras clínicas. Revista de microbiología clínica. 21, 46-76 (1985).
- Davin-Regli A, Bosi C, Charrel R y col. Brote nosocomial por cepas de Enterobacter cloacae con el genotipo E. hormaechei en pacientes tratados con fluoroquinolonas. Revista de microbiología clínica. 35, 1008-1010 (1997).
- Paauw A, Caspers MPM, Leverstein-van Hall MA y col. Identificación de factores de resistencia y virulencia en una cepa epidémica del brote de Enterobacter hormaechei. Microbiología. 155, 1478–1488 (2009).
- Wenger PN, Tokars JI, Brennan P y col. Un brote de infección y colonización por Enterobacter hormaechei en un vivero de cuidados intensivos. Enfermedades Clínicas Infecciosas. 24 (6), 1243-1244 (1997).
- Campos LC, Lobianco LF, Seki LM, Santos RM, Asensi MD. Brote de septicemia por Enterobacter hormaechei en recién nacidos causada por nutrición parenteral contaminada en Brasil. Revista de infección hospitalaria. 66 (1), 95–97 (2007).
- Kosako Y, Tamura K, Sakazaki R, Miki K. Enterobacter kobei sp. nov., una nueva especie de la familia Enterobacteriaceae que se asemeja a Enterobacter cloacae. Microbiología actual. 33, 261-265 (1996).
- Granjero JJ. Enterobacteriaceae. En: Manual de Microbiología Clínica (6ª Edición). Murray PR, Baron EJ, Pfaller MA, Tenover FC, Yolken RH (Eds). Sociedad Estadounidense de Microbiología, Washington, DC, EE. UU., 438–449 (1994).
- Ludwig W, Klenk HP. Descripción general: una columna vertebral filogenética y un marco taxonómico para la sistemática procariótica, en: Manual de Bergey de bacteriología sistemática (segunda edición). Garrity G (Ed.). Springer, NY, EE. UU., 49–65 (2001).
- Ludwig W, Strunk O, Westram R y col. ARB: un entorno de software para datos de secuencia. Investigación de ácidos nucleicos. 32, 1363-1371 (2004).
- Stock I, Grüger T, Wiedemann B. Sensibilidad antibiótica natural de cepas del complejo Enterobacter cloacae. Revista Internacional de Agentes Antimicrobianos. 18 (6), 537–545 (2001). Evaluación de una amplia gama de antibióticos probados contra cepas de E. cloacae, Enterobacter hormaechei y Enterobacter asburiae, proporcionando una base de datos para su susceptibilidad natural.
- Kim DM, Jang SJ, Neupane GP y col. Enterobacter nimipressuralis como causa de pseudobacteremia. Enfermedades Infecciosas de BMC. 10, 315 (2010).
- Scotta C, Juan C, Cabot G et al. La microbiota ambiental representa un reservorio natural para la diseminación de metalo-beta-lactamasas clínicamente relevantes. Agentes antimicrobianos y quimioterapia. 55, 5376–5379 (2011).
- George AJ. Β-lactamasas AmpC. Revisiones de microbiología clínica. 22 (1), 161–182 (2009).
- Roh KH, Song W, Chung HS y col. Cefalosporinasa cromosómica en Enterobacter hormaechei como antepasado de la betalactamasa AmpC mediada por plásmido ACT-1. Revista de Microbiología Médica. 61 (1), 94–100 (2012).
- Choi SH, Lee JE, Park SJ y col. Prevalencia, microbiología y características clínicas de Enterobacter spp., Serratia marcescens, Citrobacter freundii y Morganella morganii productoras de betalactamasas de espectro extendido en Corea. Revista europea de microbiología clínica y enfermedades infecciosas. 26, 557–561 (2007).
- Smith Moland E, Sanders CC, Thomson KC. ¿Pueden los resultados obtenidos con los paneles de microdilución MicroScan disponibles comercialmente servir como indicador de la producción de beta-lactamasa entre los aislados de Escherichia coli y Klebsiella con resistencia oculta a las cefalosporinas de espectro expandido y al aztreonam? Revista de microbiología clínica. 36, 2575-2579 (1998).
- Tzelepi E, Giakkoupi P, Sofianou D, Loukova V, Kemeroglou A, Tsakris A. Detección de betalactamasas de espectro extendido en aislados clínicos de Enterobacter cloacae y Enterobacter aerogenes. Revista de microbiología clínica. 38 (2), 542–546 (2000).
- Tzouvelekis LS, Vatopoulos AC, Katsanis G, Tzelepi E. Caso raro de falla de un sistema automatizado para detectar betalactamasa de espectro extendido en un aislado de Klebsiella pneumoniae resistente a cefalosporinas. Revista de microbiología clínica. 37 (7), 2388 (1999).
- Girlich D, Poirel L, Leelaporn A y col. Epidemiología molecular de la betalactamasa de espectro extendido VEB-1 integrada en aislamientos de enterobacterias nosocomiales en Bangkok, Tailandia. Revista de microbiología clínica. 39, 175-182 (2001).
- Paterson DL. Resistencia en bacterias Gram-negativas: Enterobacteriaceae. Revista estadounidense de medicina. 119 (6 Supl. 1), S20 – S28 (2006).
- Paterson DL, Bonomo RA. Betalactamasas de espectro extendido: una actualización clínica. Revisiones de microbiología clínica. 18, 657–686 (2005).
- Jiang X, Ni Y, Jiang Y y col. Brote de infección causado por Enterobacter cloacae que produce la novedosa betalactamasa VEB-3 en China. Revista de microbiología clínica. 43 (2), 826–831 (2005).
- Ho PL, Shek RH, Chow KH y col. Detección y caracterización de betalactamasas de espectro extendido entre aislados del torrente sanguíneo de Enterobacter spp. en Hong Kong, 2000-2002. Revista de quimioterapia antimicrobiana. 55 (3), 326–332 (2005).
- Pitout JD, Laupland KB. Enterobacteriaceae productoras de betalactamasas de espectro extendido: un problema emergente de salud pública. Enfermedades Infecciosas Lancet. 8, 159-166 (2008).
- Poirel L, Pitout JD, Nordmann P. Carbapenemasas: diversidad molecular y consecuencias clínicas. Microbiología del futuro. 2 (5), 501–512 (2007).
- Panagea T, Galani I, Souli M, Adamou P, Antoniadou A, Giamarellou H. Evaluación de CHROMagar ™ KPC para la detección de enterobacterias productoras de carbapenemasas en cultivos de vigilancia rectal. Revista Internacional de Agentes Antimicrobianos. 37 (2), 124-128 (2011).
- Cohen Stuart J, Leverstein-Van Hall MA; Grupo de trabajo holandés sobre la detección de microorganismos altamente resistentes. Directriz para el cribado fenotípico y la confirmación de carbapenemasas en Enterobacteriaceae. Revista Internacional de Agentes Antimicrobianos. 36 (3), 205–210 (2010).
- Lo A, Verrall R, Williams J, Stratton C, Della-Latta P, Tang YW. Resistencia a carbapenémicos a través del gen bla KPC-2 en aislados de hemocultivo de Enterobacter cloacae. Revista médica del sur. 103 (5), 453–454 (2010).
- Bush K, Jacoby GA. Clasificación funcional actualizada de betalactamasas. Agentes antimicrobianos y quimioterapia. 54, 969–976 (2010).
- Nordmann P, Naas T, Poirel L. Diseminación global de enterobacterias productoras de carbapenemasas. Enfermedades infecciosas emergentes. 17, 1791-1798 (2011).
- Naas T, Nordmann P. Análisis de una betalactamasa de clase A hidrolizante de carbapenémicos de Enterobacter cloacae y de su proteína reguladora de tipo LysR. Actas de la Academia Nacional de Ciencias - EE. UU. 91, 7693-7697 (1994).
- Radice M, Power P, Gutkind G y col. Carbapenemasa de primera clase A aislada de Enterobacteriaceae en Argentina. Agentes antimicrobianos y quimioterapia. 48, 1068–1069 (2004).
- Pottumarthy S, Moland ES, Jeretschko S, Swanzy SR, Thomson KS, Fritsche TR. Enzima hidrolizante de carbapenémicos NmcA en Enterobacter cloacae en América del Norte. Enfermedades infecciosas emergentes. 9, 999-1002 (2003).
- Naas T, Cattoen C, Bernusset S, Cuzon G, Nordmann P. Primera identificación de bla IMI-1 en un aislado clínico de Enterobacter cloacae de Francia. Agentes antimicrobianos y quimioterapia. 56 (3), 1664-1665 (2012).
- Rasmussen BA, Bush K, Keeney D y col. Caracterización de la betalactamasa IMI-1, una enzima hidrolizante de carbapenémicos de clase A de Enterobacter cloacae. Agentes antimicrobianos y quimioterapia. 40, 2080-2086 (1996).
- Yun-Song Y, Xiao-Xing D, Zhi-Hui Z, Ya-Gang C, Lan-Juan L.Primer aislamiento de bla IMI-2 en un aislado clínico de Enterobacter cloacae de China. Agentes antimicrobianos y quimioterapia. 50, 1610-1611 (2006).
- Nordmann P, Mariotte S, Naas T, Labia R, Nicolas MH. Propiedades bioquímicas de una betalactamasa que hidroliza carbapenémicos de Enterobacter cloacae y clonación del gen en Escherichia coli. Agentes antimicrobianos y quimioterapia. 37 (5), 939–946 (1993).
- Queenan AM, Bush K. Carbapenemasas: las beta-lactamasas versátiles. Revisiones de microbiología clínica. 20, 440–458 (2007). Se centra en información actualizada sobre las características epidemiológicas y bioquímicas de las carbapenemasas de E. cloacae.
- Aubron C, Poirel L, Ash RJ, Nordmann P. Enterobacteriaceae productoras de carbapenemasas, ríos de EE. UU. Enfermedades infecciosas emergentes. 11, 260-264 (2005).
- Giakkoupi P, Tzouvelekis LS, Tsakris A, Loukova V, Sofianou D, Tzelepi E. IBC-1, una nueva betalactamasa de clase A asociada a integrones con propiedades de espectro extendido producidas por una cepa clínica. Agentes antimicrobianos y quimioterapia. 44, 2247-2253 (2000).
- Bratu S, Landman D, Alam M, Tolentino E, Quale J. Detección de enzimas hidrolizantes de carbapenémicos KPC en Enterobacter spp. de Brooklyn, Nueva York. Agentes antimicrobianos y quimioterapia. 49, 776–778 (2005).
- Cornaglia G, Giamarellou H, Rossolini GM. Metalo-beta-lactamasas: ¿una última frontera para los beta-lactámicos? Enfermedades Infecciosas Lancet. 11 (5), 381–393 (2011).
- Deshpande LM, Jones RN, Fritsche TR, Sader HS. Ocurrencia y caracterización de enterobacterias productoras de carbapenemasas: informe del Programa de vigilancia de antimicrobianos SENTRY (2000-2004). Resistencia a fármacos microbianos. 12 (4), 223–230 (2006).
- Yan JJ, Ko WC, Chuang CL, Wu JJ. Aislamientos de Enterobacteriaceae productoras de metalo-beta-lactamasas en un hospital universitario de Taiwán: prevalencia de IMP-8 en Enterobacter cloacae y primera identificación de VIM-2 en Citrobacter freundii. Revista de quimioterapia antimicrobiana. 50, 503-511 (2002).
- Lee MF, Peng CF, Hsu HJ, Chen YH. Caracterización molecular de los genes de metalo-beta-lactamasa en bacterias Gram-negativas resistentes al imipenem de un hospital universitario en el sur de Taiwán. Revista Internacional de Agentes Antimicrobianos. 32, 475–480 (2008).
- Luzzaro F, Docquier JD, Colinon C et al. Aparición en aislados clínicos de Klebsiella pneumoniae y Enterobacter cloacae de la metalo-beta-lactamasa VIM-4 codificada por un plásmido conjugativo. Agentes antimicrobianos y quimioterapia. 48, 648–650 (2004).
- Perilli MG, Mezzatesta ML, Marco F y col. Carbapenemasa bla VIM-1 de clase I transmitida por integrones en una cepa de Enterobacter cloacae responsable de un caso de neumonía mortal. Resistencia a fármacos microbianos. 14 (1), 45–47 (2008).
- Falcone M, Mezzatesta ML, Perilli MG y col. Infecciones por Enterobacter cloacae productor de metalo-beta-lactamasa VIM-1 y su correlación con el resultado clínico. Revista de microbiología clínica. 47 (11), 3514–3519 (2009).
- Panopoulou M, Alepopoulou E, Ikonomidis A, Grapsa A, Paspalidou E, Kartali-Ktenidou S. Aparición de VIM-12 en Enterobacter cloacae. Revista de microbiología clínica. 48 (9), 3414–3415 (2010).
- Souli M, Kontopidou FV, Papadomichelakis E, Galani I, Armaganidis A, Giamarellou H. Experiencia clínica de infecciones graves causadas por enterobacterias productoras de metalo-beta-lactamasa VIM-1 en un hospital universitario griego. Enfermedades Clínicas Infecciosas. 46, 847–854 (2008).
- Tato M, Coque TM, Ruiz-Garbajosa P et al. Epidemiología compleja clonal y plasmídica en el primer brote de infección por Enterobacteriaceae con metalo-beta-lactamasa VIM-1 en España: ¿hacia la endemicidad? Enfermedades Clínicas Infecciosas. 45, 1171-1178 (2007).
- Yong D, Toleman MA, Giske CG et al. Caracterización de un nuevo gen de metalo-beta-lactamasa, bla NDM-1, y un nuevo gen de eritromicina esterasa portado por una estructura genética única en la secuencia de Klebsiella pneumoniae tipo 14 de la India. Agentes antimicrobianos y quimioterapia. 53, 5046–5054 (2009).
- Brink AJ, Coetzee J, Clay CG et al. Aparición de la metalo-beta-lactamasa de Nueva Delhi (NDM-1) y la carbapenemasa de Klebsiella pneumoniae (KPC-2) en Sudáfrica. Revista de microbiología clínica. 50 2), 525–527 (2012).
- Bogaerts P, Bouchahrouf W, Rezende de Castro R et al. Aparición de enterobacterias productoras de NDM-1 en Bélgica. Agentes antimicrobianos y quimioterapia. 55, 3036-3038 (2011).
- Carrer A, Poirel L, Yilmaz M et al. Propagación del plásmido que codifica OXA-48 en Turquía y más allá. Agentes antimicrobianos y quimioterapia. 54 (3), 1369-1373 (2010).
- Poirel L, Castanheira M, Carrër A y col. OXA-163, una betalactamasa de clase D relacionada con OXA-48 con actividad extendida hacia cefalosporinas de espectro expandido. Agentes antimicrobianos y quimioterapia. 55 (6), 2546-2551 (2011).
- Glupczynskia Y, Huanga TD, Bouchahroufa W et al. Emergencia rápida y diseminación de aislados de Enterobacteriaceae resistentes a carbapenémicos productores de OXA-48 en hospitales belgas. Revista Internacional de Agentes Antimicrobianos. 39, 168-172 (2012).
- Poirel L, Ros A, Carrër A y col. Transmisión transfronteriza de Enterobacter cloacae productor de OXA-48 desde Marruecos a Francia. Revista de quimioterapia antimicrobiana. 66, 1181-1182 (2011).
- Szabó D, Silveira F, Hujer AM et al. Los cambios en las proteínas de la membrana externa y la expresión de la bomba de salida juntos pueden conferir resistencia al ertapenem en Enterobacter cloacae. Agentes antimicrobianos y quimioterapia. 50 (8), 2833–2835 (2006).
- Chow JW, Fine MJ, Shlaes DM y col. Bacteremia por Enterobacter: características clínicas y aparición de resistencia a los antibióticos durante la terapia. Annals of Internal Medicine. 115, 585-590 (1991).
- Choi SH, Lee JE, Park SJ y col. Aparición de resistencia a los antibióticos durante la terapia de infecciones causadas por enterobacterias productoras de ampC beta-lactamasa implicaciones para el uso de antibióticos. Agentes antimicrobianos y quimioterapia. 52, 995–1000 (2008).
- Baucheron S, Imberechts H, Chaslus-Dancla E, Cloeckaert A. El transportador multifármaco AcrB juega un papel importante en la resistencia de alto nivel a las fluoroquinolonas en el fago de Salmonella enterica serovar Typhimurium tipo DT204. Resistencia a fármacos microbianos. 8, 281-289 (2002).
- Ruiz J. Mecanismos de resistencia a las quinolonas: alteraciones de la diana, disminución de la acumulación y protección de la ADN girasa. Revista de quimioterapia antimicrobiana. 51, 1109-1117 (2003).
- Perichon B, Courvalin P, Galimand M. Resistencia transferible a aminoglucósidos por metilación de G1405 en ARNr 16S ya fluoroquinolonas hidrófilas por eflujo mediado por QepA en Escherichia coli. Agentes antimicrobianos y quimioterapia. 51, 2464–2469 (2007).
- Cano ME, Rodríguez-Martínez JM, Aguero J et al. Detección de genes de resistencia a quinolonas mediados por plásmidos en aislados clínicos de Enterobacter spp. en España. Revista de microbiología clínica. 47 (7), 2033-2039 (2009).
- Robicsek A, Jacoby GA, Hooper DC. La aparición mundial de la resistencia a las quinolonas mediada por plásmidos. Enfermedades Infecciosas Lancet. 6, 629–640 (2006).
- Martínez-Martínez L, Cano ME, Rodríguez-Martínez JM, Calvo J, Pascual A. Resistencia a quinolonas mediada por plásmidos. Revisión de expertos de la terapia antiinfecciosa. 6, 685–711 (2008).
- Park CH, Robicsek A, Jacoby GA, Sahm D, Hooper DC. Prevalencia en los Estados Unidos de aac (6 ') - Ib-cr que codifica una enzima modificadora de ciprofloxacina. Agentes antimicrobianos y quimioterapia. 50, 3953–3955 (2006).
- Jacoby GA, Chow N, Waites KB. Prevalencia de resistencia a quinolonas mediada por plásmidos. Agentes antimicrobianos y quimioterapia. 47, 559–562 (2003).
- Park YJ, Yu JK, Lee S, Oh EJ, Woo GJ. Prevalencia y diversidad de alelos qnr en Enterobacter cloacae, Enterobacter aerogenes, Citrobacter freundii y Serratia marcescens productores de AmpC: un estudio multicéntrico de Corea. Revista de quimioterapia antimicrobiana. 60, 868–871 (2007).
- Nordmann P, Poirel L. Emergencia de resistencia mediada por plásmidos a quinolonas en Enterobacteriaceae. Revista de quimioterapia antimicrobiana. 56, 463–469 (2005).
- Wu JJ, Ko WC, Tsai SH, Yan JJ. Prevalencia de los determinantes de resistencia a quinolonas QnrA, QnrB y QnrS mediados por plásmidos entre los aislados clínicos de Enterobacter cloacae en un hospital taiwanés. Agentes antimicrobianos y quimioterapia. 51, 1223-1227 (2007).
- Chmelnitsky I, Navon-Venezia S, Strahilevitz J, Carmeli Y. qnrB2 mediada por plásmido y gen de carbapenemasa bla KPC-2 transportados en el mismo plásmido en aislados de Enterobacter cloacae susceptibles a ciprofloxacina resistentes a carbapenémicos. Agentes antimicrobianos y quimioterapia. 52 (8), 2962–2965 (2008).
- Jacoby G, Cattoir V, Hooper D y col. nomenclatura del gen qnr. Agentes antimicrobianos y quimioterapia. 52, 2297–2299 (2008).
- Neonakis I, Gikas A, Scoulica E, Manios A, Georgiladakis A, Tselentis Y. Evolución de los fenotipos de resistencia a los aminoglucósidos de cuatro bacterias gramnegativas: una encuesta de 8 años en un hospital universitario de Grecia. Revista Internacional de Agentes Antimicrobianos. 22, 526-531 (2003).
- Kim SY, Park YJ, Yu JK, Kim YS, Han K.Prevalencia y características de aac (6 ') - Ib-cr en Enterobacter cloacae, Citrobacter freundii y Serratia marcescens productores de AmpC: un estudio multicéntrico de Corea. Microbiología diagnóstica y enfermedades infecciosas. 63, 314–318 (2009).
- Galani I, Souli M, Chryssouli Z, Orlandou K, Giamarellou H. Caracterización de un nuevo integrón que contiene bla VIM-1 y aac (6 ') - IIc en un aislado clínico de Enterobacter cloacae de Grecia. Revista de quimioterapia antimicrobiana. 55, 634–638 (2005).
- Xavier B, Dowzicky MJ. Susceptibilidad antimicrobiana entre cepas gramnegativas recolectadas de unidades de cuidados intensivos en América del Norte, Europa, la región de Asia y el Pacífico, América Latina, Oriente Medio y África entre 2004 y 2009 como parte del ensayo de evaluación y vigilancia de tigeciclina. Terapéutica clínica. 34 (1), 124-137 (2012).
- Anthony KB, Fishman NO, Linkin DR, Gasink LB, Edelstein PH, Lautenbach E. Resultados clínicos y microbiológicos de infecciones graves con organismos gramnegativos multirresistentes tratados con tigeciclina. Enfermedades Clínicas Infecciosas. 46, 567–570 (2008).
- Keeney D, Ruzin A, Bradford PA, RamA, un regulador transcripcional, y AcrAB, una bomba de eflujo tipo RND, están asociados con una menor susceptibilidad a la tigeciclina en Enterobacter cloacae. Resistencia a fármacos microbianos. 13 (1), 1 a 6 (2007).
- Daurel C, Fiant AL, Brémont S, Courvalin P, Leclercq R. Emergencia de una cepa de Enterobacter hormaechei con susceptibilidad reducida a tigeciclina bajo terapia con tigeciclina. Agentes antimicrobianos y quimioterapia. 53, 4953–4954 (2009).
- Falagas ME, Kasiakou SK. Colistina: la reactivación de las polimixinas para el tratamiento de infecciones bacterianas gramnegativas multirresistentes. Enfermedades Clínicas Infecciosas. 40 (9), 1333-1341 (2006). Errata en: Enfermedades Infecciosas Clínicas. 42 (12), 1819 (2006).
- Precio DJE, Graham DI. Efecto de grandes dosis de sulfometato de colistina sobre la función renal. Revista médica británica. 4, 525-527 (1970).
- Koch-Weser J, Sidel VW, Federman EB, Kanarek P, Finer DC, Eaton AE. Efectos adversos del metato de colistina sódica: manifestaciones y tasas de reacción específicas durante 317 cursos de terapia. Annals of Internal Medicine. 72, 857–868 (1970).
- Li J, Nation RL, Milne RW, Turnidge JD, Coulthard K. Evaluación de la colistina como agente contra bacterias Gram negativas multirresistentes. Revista Internacional de Agentes Antimicrobianos. 25, 11-25 (2005).
- Plachouras D, Karvanen M, Friberg LE et al. Análisis farmacocinético poblacional de metanosulfonato de colistina y colistina después de la administración intravenosa en pacientes críticamente enfermos con infecciones causadas por bacterias gramnegativas. Agentes antimicrobianos y quimioterapia. 53 (8), 3430–3436 (2009).
- Groisman EA, Kayser J, Soncini FC. Regulación de la resistencia a la polimixina y adaptación a ambientes bajos en Mg2 +. Revista de bacteriología. 179 (22), 7040–7045 (1997).
- Lo-Ten-Foe JR, de Smet AM, Diederen BM, Kluytmans JA, van Keulen PH. Evaluación comparativa de los métodos de prueba de susceptibilidad de Vitek 2, difusión en disco, etest, microdilución en caldo y dilución en agar para la colistina en aislados clínicos, incluidas las cepas heterorresistentes de Enterobacter cloacae y Acinetobacter baumannii. Agentes antimicrobianos y quimioterapia. 51 (10), 3726–3730 (2007).
- Tascini C, Urbani L, Biancofiore G et al. Colistina en combinación con rifampicina e imipenem para el tratamiento de una infección diseminada por Enterobacter cloacae productora de metalo-beta-lactamasa bla VIM-1 en un paciente con trasplante de hígado. Minerva Anestesiologica. 74 (1–2), 47–49 (2007).
- Centros de Control y Prevención de Enfermedades. Orientación para el control de infecciones por enterobacterias resistentes a carbapenemas o productoras de carbapenemasas en centros de cuidados intensivos. Informe Semanal de Morbilidad y Mortalidad. 58, 256–260 (2009).
- Lucet JC, Decre D, Fichelle A et al. Control de un brote prolongado de enterobacterias productoras de betalactamasas de espectro extendido en un hospital universitario. Enfermedades Clínicas Infecciosas. 29, 1411-1418 (1999).
- Samra Z, Bahar J, Madar-Shapiro L, Aziz N, Israel S, Bishara J. Evaluación de CHROMagar KPC para la detección rápida de enterobacterias resistentes a carbapenémicos. Revista de microbiología clínica. 46, 3110–3111 (2008).
- Ravikumar P. GenBank Nuevo holotipo para Enterobacter cloacae subsp. cloacae cepa PR-4 aislada e identificada por la secuencia del gen 16S rDNA con vista de árbol filogenético, de suelo cargado de explosivos. Revista Internacional de Investigación en Ingeniería y Ciencias Aplicadas. 6 (5) 53-65 (2016).
- Padhi, SK, Gokhale, S., 2016. Biodegradación de benceno por cultivo microbiano mixto indígena: modelado cinético y optimización de procesos. Biodeterioro y biodegradación internacional. https://dx.doi.org/10.1016/j.ibiod.2016.10.011
Enlaces externos [ editar ]
- Tipo de cepa de Enterobacter cloacae en Bac Dive - la base de metadatos de diversidad bacteriana