En biología molecular , la tapa de cinco primos ( tapa 5 ' ) es un nucleótido especialmente alterado en el extremo 5' de algunas transcripciones primarias como el ARN mensajero precursor . Este proceso, conocido como recubrimiento de ARNm , está altamente regulado y es vital en la creación de ARN mensajero estable y maduro capaz de someterse a traducción durante la síntesis de proteínas . El ARNm mitocondrial [1] y el ARNm cloroplástico [2] no están protegidos.
Estructura [ editar ]
En eucariotas , el casquete 5 ′ (cap-0), que se encuentra en el extremo 5 ′ de una molécula de ARNm, consiste en un nucleótido de guanina conectado al ARNm a través de un enlace trifosfato de 5 ′ a 5 ′ inusual . Esta guanosina se metila en la posición 7 directamente después de tapar in vivo por una metiltransferasa . [3] [4] [5] [6] Se le conoce como tapa de 7-metilguanilato , abreviado m 7 G.
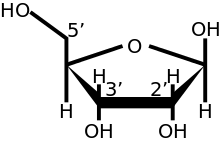
En eucariotas multicelulares y algunos virus, [7] existen más modificaciones, incluida la metilación de los grupos hidroxi 2 ' de los 2 primeros azúcares ribosa del extremo 5' del ARNm. cap-1 tiene un grupo 2'-hidroxi metilado en el primer azúcar ribosa, mientras que cap-2 tiene grupos 2'-hidroxi metilado en los dos primeros azúcares ribosa, que se muestran a la derecha. La tapa 5 'es químicamente similar al extremo 3' de una molécula de ARN (el carbono 5 'de la tapa ribosa está unido y el 3' no unido). Esto proporciona una resistencia significativa a las exonucleasas 5 ' . [ cita requerida ]
Los ARN nucleares pequeños contienen casquillos 5 ′ únicos. Los snRNA de clase Sm se encuentran con tapas de 5′-trimetilguanosina, mientras que los snRNA de clase Lsm se encuentran con tapas de 5′-monometilfosfato. [8]
En las bacterias , y potencialmente también en organismos superiores, algunos ARN se tapan con NAD + , NADH , o 3 'dephospho-coenzima A . [9] [10]
En todos los organismos, las moléculas de ARNm se pueden decapar en un proceso conocido como decapitación del ARN mensajero .
Proceso de taponado [ editar ]
El punto de partida para la protección con 7-metilguanilato es el extremo 5 'inalterado de una molécula de ARN, que termina en un grupo trifosfato. Este presenta un nucleótido final seguido de tres grupos fosfato unidos al carbono 5 '. [3] El proceso de limitación se inicia antes de que se complete la transcripción, ya que se sintetiza el pre-ARNm naciente.
- Uno de los grupos fosfato terminales es eliminado por la ARN trifosfatasa , dejando un grupo bisfosfato (es decir, 5 '(ppN) [pN] n );
- La guanililtransferasa de ARNm , la guanililtransferasa , añade GTP al bisfosfato terminal , y en el proceso se pierde un pirofosfato del sustrato de GTP. Esto da como resultado el enlace trifosfato 5′-5 ′, que produce 5 ′ (Gp) (ppN) [pN] n ;
- El nitrógeno 7 de la guanina es metilado por el ARNm (guanina- N 7 -) - metiltransferasa , con S -adenosil- L- metionina desmetilada para producir S -adenosil- L -homocisteína , lo que da como resultado 5 ′ (m7Gp) (ppN) [pN] n (cap-0);
- Pueden producirse modificaciones adyacentes a la tapa, normalmente al primer y segundo nucleótidos, produciendo hasta 5 '(m7Gp) (ppN *) (pN *) [pN] n (cap-1 y cap-2); [7]
- Si el nucleótido cap-adyacente más cercano es 2'- O ribosa metil-adenosina (es decir, 5 '(m7Gp) (PDMA) [pN] n ), puede ser metilado aún más en la posición de metilo N6 para formar N 6 -methyladenosine , dando como resultado 5 '(m7Gp) (ppm6Am) [pN] n . [3]
El mecanismo de protección con NAD + , NADH o 3′-desfosfocoenzima A es diferente. El taponamiento con NAD + , NADH o 3′-desfosfo-coenzima A se logra mediante un "mecanismo de taponamiento ab initio", en el que NAD + , NADH o 3′-desfosfo-coenzima A sirve como un "nucleótido iniciador no canónico "(NCIN) para el inicio de la transcripción por la ARN polimerasa y, por lo tanto, se incorpora directamente al producto de ARN. [9] Tanto la ARN polimerasa bacteriana como la ARN polimerasa II eucariota pueden llevar a cabo este "mecanismo de protección ab initio". [9]
Orientación [ editar ]
Para la protección con 7-metilguanilato, el complejo enzimático de protección (CEC) se une a la ARN polimerasa II antes de que comience la transcripción. Tan pronto como el extremo 5 'de la nueva transcripción emerge de la ARN polimerasa II, el CEC lleva a cabo el proceso de taponamiento (este tipo de mecanismo asegura el taponamiento, como ocurre con la poliadenilación ). [11] [12] [13] [14] Las enzimas para la protección solo pueden unirse a la ARN polimerasa II , lo que garantiza la especificidad solo para estas transcripciones, que son casi en su totalidad ARNm. [12] [14]
El recubrimiento con NAD + , NADH o 3'-desfosfo-coenzima A es el objetivo de la secuencia promotora . [9] La protección con NAD +, NADH o 3'-defosfo-coenzima A ocurre solo en los promotores que tienen ciertas secuencias en e inmediatamente aguas arriba del sitio de inicio de la transcripción y por lo tanto ocurre solo para los ARN sintetizados a partir de ciertos promotores. [9]
Función [ editar ]
El tapón de 5 ′ tiene cuatro funciones principales:
- Regulación de la exportación nuclear; [15] [16]
- Prevención de la degradación por exonucleasas ; [9] [17] [18] [19]
- Promoción de la traducción (ver ribosoma y traducción ); [3] [4] [5]
- Promoción de la escisión del intrón proximal 5 '. [20]
La exportación nuclear de ARN está regulada por el complejo de unión cap (CBC), que se une exclusivamente al ARN protegido con 7-metilguanilato. El CBC es luego reconocido por el complejo de poros nucleares y exportado. Una vez en el citoplasma después de la ronda pionera de traducción, el CBC es reemplazado por los factores de traducción eIF4E y eIF4G del complejo eIF4F . [6] Este complejo es luego reconocido por otra maquinaria de iniciación de la traducción, incluido el ribosoma. [21]
El taponamiento con 7-metilguanilato evita la degradación de 5 'de dos formas. En primer lugar, se evita la degradación del ARNm por las exonucleasas 5 '(como se mencionó anteriormente) pareciendo funcionalmente un extremo 3'. En segundo lugar, el CBC y el eIF4E / eIF4G bloquean el acceso de las enzimas de desencadenamiento a la tapa. Esto aumenta la vida media del ARNm, esencial en eucariotas, ya que los procesos de exportación y traducción toman un tiempo significativo.
El decapitado de un ARNm protegido con 7-metilguanilato es catalizado por el complejo de decaptación formado por al menos Dcp1 y Dcp2, que debe competir con eIF4E para unirse al casquete. Por lo tanto, el casquete de 7-metilguanilato es un marcador de un ARNm que se traduce activamente y es utilizado por las células para regular la vida media del ARNm en respuesta a nuevos estímulos. Los ARNm indeseables se envían a los cuerpos P para su almacenamiento temporal o descampado, cuyos detalles aún se están resolviendo. [22]
El mecanismo de promoción de la escisión del intrón proximal 5 'no se comprende bien, pero la tapa de 7-metilguanilato parece girar e interactuar con el espliceosoma en el proceso de empalme, promoviendo la escisión del intrón.
Ver también [ editar ]
- Desencapsulado del ARN mensajero
- Factor de iniciación eucariota 4F (eIF4F)
- Expresión génica del análisis de cap
Referencias [ editar ]
- ^ Temperley RJ, Wydro M, Lightowlers RN, Chrzanowska-Lightowlers ZM (junio de 2010). "ARNm mitocondriales humanos, como miembros de todas las familias, similares pero diferentes" . Biochimica et Biophysica Acta (BBA) - Bioenergética . 1797 (6–7): 1081–1085. doi : 10.1016 / j.bbabio.2010.02.036 . PMC 3003153 . PMID 20211597 .
- ^ Monde RA, Schuster G, Stern DB (7 de junio de 2000). "Procesamiento y degradación de ARNm de cloroplasto". Biochimie . 82 (6–7): 573–582. doi : 10.1016 / S0300-9084 (00) 00606-4 . PMID 10946108 .
- ↑ a b c d Shatkin, A (diciembre de 1976). "Tapado de ARNm eucarióticos". Celular . 9 (4): 645–653. doi : 10.1016 / 0092-8674 (76) 90128-8 . PMID 1017010 . S2CID 26743858 .
- ↑ a b Banerjee AK (junio de 1980). "Estructura del casquete 5′-terminal en ácidos ribonucleicos mensajeros eucarióticos" . Revisiones microbiológicas . 44 (2): 175–205. doi : 10.1128 / mmbr.44.2.175-205.1980 . PMC 373176 . PMID 6247631 .
- ↑ a b Sonenberg N, Gingras AC (abril de 1998). "La proteína de unión a la tapa 5 'de ARNm eIF4E y el control del crecimiento celular". Opinión actual en biología celular . 10 (2): 268-275. doi : 10.1016 / S0955-0674 (98) 80150-6 . PMID 9561852 .
- ↑ a b Marcotrigiano J, Gingras AC, Sonenberg N, Burley SK (junio de 1997). "Estructura de cocristal de la proteína de unión a la tapa 5 'del ARN mensajero (eIF4E) unida a 7-metil-GDP". Celular . 89 (6): 951–961. doi : 10.1016 / S0092-8674 (00) 80280-9 . PMID 9200613 . S2CID 15200116 .
- ↑ a b Fechter P, Brownlee GG (mayo de 2005). "Reconocimiento de estructuras de casquete de ARNm por proteínas virales y celulares" . La Revista de Virología General . 86 (Parte 5): 1239-1249. doi : 10.1099 / vir.0.80755-0 . PMID 15831934 .
- ^ Matera AG, Charranes RM, Charranes MP (marzo de 2007). "ARN no codificantes: lecciones de los pequeños ARN nucleares y nucleolares pequeños". Reseñas de la naturaleza. Biología celular molecular . 8 (3): 209–220. doi : 10.1038 / nrm2124 . PMID 17318225 . S2CID 30268055 .
- ^ a b c d e f Bird JG, Zhang Y, Tian Y, Panova N, Barvík I, Greene L, Liu M, Buckley B, Krásný L, Lee JK, Kaplan CD, Ebright RH, Nickels BE (julio de 2016). "El mecanismo de protección del ARN 5 ′ con NAD +, NADH y desfosfo-CoA" . Naturaleza . 535 (7612): 444–447. Código bibliográfico : 2016Natur.535..444B . doi : 10.1038 / nature18622 . PMC 4961592 . PMID 27383794 .
- ^ Cahová H, Winz ML, Höfer K, Nübel G, Jäschke A (marzo de 2015). "NAD captureSeq indica NAD como una tapa bacteriana para un subconjunto de ARN reguladores". Naturaleza . 519 (7543): 374–377. Código bibliográfico : 2015Natur.519..374C . doi : 10.1038 / nature14020 . PMID 25533955 . S2CID 4446837 .
- ^ Cho EJ, Takagi T, Moore CR, Buratowski S (diciembre de 1997). "La enzima de protección de ARNm se recluta en el complejo de transcripción por fosforilación del dominio carboxi-terminal de la ARN polimerasa II" . Genes y desarrollo . 11 (24): 3319–3326. doi : 10.1101 / gad.11.24.3319 . PMC 316800 . PMID 9407025 .
- ↑ a b Fabrega C, Shen V, Shuman S, Lima CD (junio de 2003). "Estructura de una enzima de protección de ARNm unida al dominio carboxi-terminal fosforilado de la ARN polimerasa II". Célula molecular . 11 (6): 1549-1561. doi : 10.1016 / S1097-2765 (03) 00187-4 . PMID 12820968 .
- ^ Ho CK, Lehman K, Shuman S (diciembre de 1999). "Un motivo de superficie esencial (WAQKW) de ARN trifosfatasa de levadura media la formación del complejo enzimático de protección de ARNm con ARN guanililtransferasa" . Investigación de ácidos nucleicos . 27 (24): 4671–4678. doi : 10.1093 / nar / 27.24.4671 . PMC 148765 . PMID 10572165 .
- ↑ a b Hirose Y, Manley JL (junio de 2000). "ARN polimerasa II y la integración de eventos nucleares" . Genes y desarrollo . 14 (12): 1415-1429. doi : 10.1101 / gad.14.12.1415 (inactivo 2021-01-11). PMID 10859161 . Consultado el 23 de noviembre de 2014 . Mantenimiento de CS1: DOI inactivo a partir de enero de 2021 ( enlace )
- ^ Visa N, Izaurralde E, Ferreira J, Daneholt B, Mattaj IW (abril de 1996). "Un complejo de unión de casquete nuclear se une cotranscripcionalmente al pre-ARNm del anillo de Balbiani y acompaña a la partícula de ribonucleoproteína durante la exportación nuclear" . The Journal of Cell Biology . 133 (1): 5–14. doi : 10.1083 / jcb.133.1.5 . PMC 2120770 . PMID 8601613 .
- ^ Lewis JD, Izaurralde E (julio de 1997). "El papel de la estructura de la tapa en el procesamiento de ARN y la exportación nuclear". Revista europea de bioquímica . 247 (2): 461–469. doi : 10.1111 / j.1432-1033.1997.00461.x . PMID 9266685 .
- ^ Evdokimova V, Ruzanov P, Imataka H, Raught B, Svitkin Y, Ovchinnikov LP, Sonenberg N (octubre de 2001). "La principal proteína asociada a ARNm, YB-1, es un potente estabilizador de ARNm dependiente de la capa 5 '" . El diario EMBO . 20 (19): 5491–5502. doi : 10.1093 / emboj / 20.19.5491 . PMC 125650 . PMID 11574481 .
- ^ Gao M, Fritz DT, Ford LP, Wilusz J (marzo de 2000). "La interacción entre una ribonucleasa específica de poli (A) y la tapa 5 'influye en las tasas de desadenilación del ARNm in vitro" . Célula molecular . 5 (3): 479–488. doi : 10.1016 / S1097-2765 (00) 80442-6 . PMC 2811581 . PMID 10882133 .
- ^ Burkard KT, Butler JS (enero de 2000). "Una exonucleasa nuclear 3′-5 ′ implicada en la degradación del mRNA interactúa con la polimerasa Poly (A) y la proteína hnRNA Npl3p" . Biología Molecular y Celular . 20 (2): 604–616. doi : 10.1128 / MCB.20.2.604-616.2000 . PMC 85144 . PMID 10611239 .
- ^ Konarska MM, Padgett RA, Sharp PA (octubre de 1984). "Reconocimiento de la estructura de la tapa en el empalme in vitro de precursores de ARNm". Celular . 38 (3): 731–736. doi : 10.1016 / 0092-8674 (84) 90268-X . PMID 6567484 . S2CID 10721149 .
- ^ Kapp LD, Lorsch JR (2004). "La mecánica molecular de la traducción eucariota". Revisión anual de bioquímica . 73 (1): 657–704. doi : 10.1146 / annurev.biochem.73.030403.080419 . PMID 15189156 .
- ^ Parker R, Sheth U (marzo de 2007). "Cuerpos P y el control de la traducción y degradación de ARNm". Célula molecular . 25 (5): 635–646. doi : 10.1016 / j.molcel.2007.02.011 . PMID 17349952 .
Enlaces externos [ editar ]
- "Tapas de ARN" . Encabezado de tema médico de PubMed (MeSH) . Institutos Nacionales de Salud.