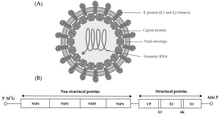
Alphavirus es un género de virus de ARN , el único género de lafamilia Togaviridae . Los alfavirus pertenecen al grupo IV de la clasificación de virus de Baltimore , con ungenoma de ARN monocatenario de sentido positivo . Hay 32 alfavirus, que infectan a varios vertebrados como humanos, roedores, peces, aves y mamíferos más grandes como caballos, así como a invertebrados . Los alfavirus que podrían infectar tanto a vertebrados como a artrópodos se denominan alfavirus de huésped dual, mientras que los alfavirus específicos de insectos como el virus Eilat y el virus Yada yada están restringidos a su vector artrópodo competente. [1]La transmisión entre especies e individuos se produce principalmente a través de mosquitos, lo que hace que los alfavirus formen parte de la colección de arbovirus , o virus transmitidos por artrópodos . Las partículas de alfavirus están envueltas, tienen un diámetro de 70 nm, tienden a ser esféricas (aunque ligeramente pleomórficas ) y tienen una nucleocápside isométrica de 40 nm . [2]
Genoma [ editar ]
| Alpha_E1_glycop | ||||||||
|---|---|---|---|---|---|---|---|---|
Estructura cristalina del homotrímero de la glicoproteína de fusión E1 del virus Semliki Forest | ||||||||
| Identificadores | ||||||||
| Símbolo | Alpha_E1_glycop | |||||||
| Pfam | PF01589 | |||||||
| InterPro | IPR002548 | |||||||
| SCOP2 | 1rer / SCOPe / SUPFAM | |||||||
| TCDB | 1.G | |||||||
| Superfamilia OPM | 109 | |||||||
| Proteína OPM | 1rer | |||||||
| ||||||||
| Alpha_E2_glycop | ||||||||
|---|---|---|---|---|---|---|---|---|
Mapeo de la glicoproteína E2 de los alfavirus | ||||||||
| Identificadores | ||||||||
| Símbolo | Alpha_E2_glycop | |||||||
| Pfam | PF00943 | |||||||
| InterPro | IPR000936 | |||||||
| TCDB | 1.G | |||||||
| Superfamilia OPM | 109 | |||||||
| Proteína OPM | 2yew | |||||||
| ||||||||
| Alpha_E3_glycop | ||||||||
|---|---|---|---|---|---|---|---|---|
| Identificadores | ||||||||
| Símbolo | Alpha_E3_glycop | |||||||
| Pfam | PF01563 | |||||||
| InterPro | IPR002533 | |||||||
| TCDB | 1.G | |||||||
| Superfamilia OPM | 109 | |||||||
| ||||||||
Los alfavirus son virus pequeños, esféricos y envueltos con un genoma de una sola hebra de ARN de sentido positivo. La longitud total del genoma varía entre 11.000 y 12.000 nucleótidos, y tiene una tapa de 5 ' y una cola de poli-A de 3' . Los cuatro genes de proteínas no estructurales están codificados en los dos tercios 5 'del genoma, mientras que las tres proteínas estructurales se traducen a partir de un ARNm subgenómico colineal con el tercio 3' del genoma.
Hay dos marcos de lectura abiertos (ORF) en el genoma, no estructural y estructural. El primero es no estructural y codifica proteínas (nsP1-nsP4) necesarias para la transcripción y replicación del ARN viral. La segunda codifica tres proteínas estructurales : la proteína C de la nucleocápside central y las proteínas de la envoltura P62 y E1, que se asocian como un heterodímero . Las glicoproteínas de superficie ancladas a la membrana viral son responsables del reconocimiento del receptor y la entrada en las células diana a través de la fusión de la membrana .
Proteínas estructurales [ editar ]
La maduración proteolítica de P62 en E2 y E3 provoca un cambio en la superficie viral. Juntos, los "picos" de glicoproteína E1, E2 y, a veces, E3, forman un dímero E1 / E2 o un trímero E1 / E2 / E3, donde E2 se extiende desde el centro hasta los vértices, E1 llena el espacio entre los vértices y E3, si está presente, está en el extremo distal de la espiga. [3] Tras la exposición del virus a la acidez del endosoma , E1 se disocia de E2 para formar un homotrímero E1 , que es necesario para que el paso de fusión impulse las membranas celular y viral.juntos. La glicoproteína alfaviral E1 es una proteína de fusión viral de clase II, que es estructuralmente diferente de las proteínas de fusión de clase I que se encuentran en el virus de la influenza y el VIH. La estructura del virus Semliki Forest reveló una estructura que es similar a la de la glicoproteína E flaviviral, con tres dominios estructurales en la misma disposición de secuencia primaria . [4] La glucoproteína E2 funciona para interactuar con la nucleocápside a través de su dominio citoplásmico , mientras que su ectodominio es responsable de unirse a un receptor celular.. La mayoría de los alfavirus pierden la proteína periférica E3, pero en los virus Semliki permanece asociada con la superficie viral.
Proteínas no estructurales [ editar ]
Cuatro proteínas no estructurales (nsP1–4) que se producen como una sola poliproteína constituyen la maquinaria de replicación del virus. [5] El procesamiento de la poliproteína ocurre de una manera altamente regulada, con la escisión en la unión P2 / 3 que influye en el uso de la plantilla de ARN durante la replicación del genoma. Este sitio está ubicado en la base de una hendidura estrecha y no es de fácil acceso. Una vez escindida, nsP3 crea una estructura de anillo que rodea a nsP2. Estas dos proteínas tienen una interfaz extensa.
Las mutaciones en nsP2 que producen virus no citopáticos o fenotipos sensibles a la temperatura se agrupan en la región de la interfaz P2 / P3. Las mutaciones de P3 opuestas a la ubicación de las mutaciones no citopáticas de nsP2 evitan la escisión eficaz de P2 / 3. Esto, a su vez, afecta la infectividad del ARN alterando los niveles de producción de ARN viral.
Virología [ editar ]
El virus tiene un diámetro de 60 a 70 nanómetros . Tiene envoltura, es esférico y tiene un genoma de ARN de cadena positiva de ~ 12 kilobases. El genoma codifica dos poliproteínas. La primera poliproteína consta de cuatro unidades no estructurales: en orden desde el terminal N al terminal C: nsP1, nsP2, nsP3 y nsP4. La segunda es una poliproteína estructural compuesta por cinco unidades de expresión: desde el terminal N al terminal C: cápside, E3, E2, 6K y E1. Un ARN subgenómico de cadena positiva, el ARN 26S, se replica a partir de un intermedio de ARN de cadena negativa. Esto sirve como molde para la síntesis de proteínas estructurales virales. La mayoría de los alfavirus tienen dominios conservados implicados en la regulación de la síntesis de ARN viral.
La nucleocápside, de 40 nanómetros de diámetro, contiene 240 copias de la proteína de la cápside y tiene una simetría icosaédrica T = 4. Las glicoproteínas virales E1 y E2 están incrustadas en la bicapa lipídica. Las moléculas individuales E1 y E2 se asocian para formar heterodímeros. Los heterodímeros E1-E2 forman contactos uno a uno entre la proteína E2 y los monómeros de la nucleocápside. Las proteínas E1 y E2 median el contacto entre el virus y la célula huésped.
Se han identificado varios receptores. Estos incluyen prohibitina , fosfatidilserina , glicosaminoglicanos y subunidad β de ATP sintasa .
La replicación ocurre dentro del citoplasma y los viriones maduran por gemación a través de la membrana plasmática, donde se asimilan las glicoproteínas de superficie E2 y E1 codificadas por virus.
Estas dos glicoproteínas son el objetivo de numerosas reacciones y pruebas serológicas, incluida la neutralización y la inhibición de la hemaglutinación. Los alfavirus muestran varios grados de reactividad cruzada antigénica en estas reacciones y esto forma la base de los siete complejos antigénicos, 32 especies y muchos subtipos y variedades. La proteína E2 es el sitio de la mayoría de los epítopos neutralizantes, mientras que la proteína E1 contiene epítopos de reacción cruzada más conservados.
Evolución [ editar ]
Un estudio de este taxón sugiere que este grupo de virus tiene un origen marino, específicamente el Océano Austral, y que posteriormente se han propagado tanto al Viejo como al Nuevo Mundo. [6]
Hay tres subgrupos en este género: el subgrupo del virus Semliki Forest (virus Semliki Forest, O'nyong-nyong y Ross River); el subgrupo del virus de la encefalitis equina del este (virus de la encefalitis equina del este y de la encefalitis equina venezolana) y el subgrupo del virus Sindbis. [7] El virus Sindbis, geográficamente restringido al Viejo Mundo, está más estrechamente relacionado con el subgrupo de encefalitis equina oriental, que son virus del Nuevo Mundo, que con el subgrupo de virus Semliki Forest, que también se encuentra en el Viejo Mundo.
Taxonomía [ editar ]
Las siguientes especies están asignadas al género: [8]
- Virus del aura
- Virus del bosque de Barmah
- Virus Bebaru
- Virus Caaingua
- Virus de Cabassou
- Virus chikungunya
- Virus de la encefalitis equina del este
- Virus de Eilat
- Virus de los Everglades
- Virus de Fort Morgan
- Virus Getah
- Virus Highlands J
- Virus Madariaga
- Virus Mayaro
- Virus de Middelburg
- Virus Mosso das Pedras
- Virus Mucambo
- Virus Ndumu
- Virus O'nyong'nyong
- Virus Pixuna
- Virus de Río Negro
- Virus del río Ross
- Virus de la enfermedad del páncreas del salmón
- Virus del bosque de Semliki
- Virus Sindbis
- Virus del elefante marino del sur
- Tonar virus
- Virus Trocara
- Una virus
- Virus de la encefalitis equina venezolana
- Virus de la encefalitis equina occidental
- Virus Whataroa
Los siete complejos son:
- Complejo de virus del bosque de Barmah
- Virus del bosque de Barmah
- Complejo de encefalitis equina del este
- Virus de la encefalitis equina del este (siete tipos antigénicos)
- Complejo del virus de Middelburg
- Virus de Middelburg
- Complejo del virus Ndumu
- Virus Ndumu
- Complejo de virus Semliki Forest
- Virus Bebaru
- Virus chikungunya
- Virus Getah
- Virus Mayaro
- Subtipo: virus Una
- Virus O'nyong'nyong
- Subtipo: virus Igbo-Ora
- Virus del río Ross
- Subtipo: virus Sagiyama
- Virus del bosque de Semliki
- Subtipo: virus Me Tri
- Complejo de encefalitis equina venezolana
- Virus de Cabassou
- Virus de los Everglades
- Virus Mosso das Pedras
- Virus Mucambo
- Virus de paramana
- Virus Pixuna
- Virus de Río Negro
- Virus Trocara
- Subtipo: virus Bijou Bridge
- Virus de la encefalitis equina venezolana
- Complejo de encefalitis equina occidental
- Virus del aura
- Virus de Babanki
- Virus Kyzylagach
- Virus Sindbis
- Virus Ockelbo
- Virus Whataroa
- Recombinantes dentro de este complejo
- Virus Buggy Creek
- Virus de Fort Morgan
- Virus Highlands J
- Virus de la encefalitis equina occidental
- Desclasificado
- Virus de Eilat
- Mwinilunga alfavirus
- Alfavirus de los salmónidos
- Virus del elefante marino del sur
- Tonar virus
- Virus de Caaingua [9]
Notas [ editar ]
El virus Barmah Forest está relacionado con el virus Semliki Forest. El virus de Middelburg, aunque clasificado como un complejo separado, puede ser miembro del grupo de virus Semliki Forest.
Parece probable que el género haya evolucionado en el Viejo Mundo a partir de un virus vegetal transmitido por insectos. [10]
El virus Sindbis puede haberse originado en América del Sur. [11] Los virus de la encefalitis equina y el virus Sindbis están relacionados.
Los virus del Viejo Mundo y del Nuevo Mundo parecen haber divergido entre 2000 y 3000 años atrás. [12] La divergencia entre el virus de la encefalitis equina venezolana y el virus equino del este parece haber sido hace unos 1400 años. [13]
El clado que infecta a los peces parece ser basal para las otras especies.
El virus del elefante marino del sur parece estar relacionado con el clado Sinbis.
Patogenia y respuesta inmune [ editar ]
| Virus | Enfermedad humana | Depósito de vertebrados | Distribución |
|---|---|---|---|
| Virus del bosque de Barmah |
| Humanos | Australia |
| Virus chikungunya | Erupción, artritis | Primates, humanos | África, América Latina, India , Sudeste de Asia |
| Virus de la encefalitis equina del este | Encefalitis | Aves | Américas |
| Virus Mayaro | Erupción, artritis | Primates, humanos | Sudamerica |
| Virus O'nyong'nyong | Erupción, artritis | Primates , humanos | África |
| Virus del río Ross | Erupción, artritis | Mamíferos, humanos | Australia, Pacífico Sur |
| Virus del bosque de Semliki | Erupción, artritis | Aves | África |
| Virus Sindbis | Erupción, artritis | Aves | Europa , África , Australia |
| Tonar virus | Encefalitis | Humanos | Sudamerica |
| Una virus | Erupción, artritis | Primates, humanos | Sudamerica |
| Virus de la encefalitis equina venezolana | Encefalitis | Roedores , caballos | Américas |
| Virus de la encefalitis equina occidental | Encefalitis | Aves, mamíferos | América del norte |
Hay muchos alfavirus distribuidos por todo el mundo con la capacidad de causar enfermedades en los seres humanos. La artritis infecciosa , encefalitis , erupciones cutáneas y fiebre son los síntomas más comúnmente observados. Los mamíferos más grandes, como los humanos y los caballos, suelen ser huéspedes sin salida o desempeñan un papel menor en la transmisión viral; sin embargo, en el caso de la encefalitis equina venezolana, el virus se amplifica principalmente en caballos. En la mayoría de los demás casos, el virus se mantiene en la naturaleza en mosquitos, roedores y aves.
Las infecciones por alfavirus terrestres se transmiten mediante insectos vectores como los mosquitos. Una vez que un mosquito infectado pica a un ser humano, el virus puede ingresar al torrente sanguíneo y causar viremia . El alfavirus también puede ingresar al SNC, donde puede crecer y multiplicarse dentro de las neuronas. Esto puede provocar encefalitis , que puede ser fatal.
Cuando un individuo está infectado con este virus en particular, su sistema inmunológico puede desempeñar un papel en la eliminación de las partículas del virus. Los alfavirus pueden provocar la producción de interferones . También están involucrados los anticuerpos y las células T. Los anticuerpos neutralizantes también juegan un papel importante para prevenir una mayor infección y propagación.
Diagnóstico, prevención y control [ editar ]
Los diagnósticos se basan en muestras clínicas de las que el virus se puede aislar e identificar fácilmente. Actualmente no hay vacunas contra el alfavirus disponibles. El control de vectores con repelentes, ropa protectora, destrucción de sitios de reproducción y fumigación son las medidas preventivas de elección.
Investigación [ editar ]
Los alfavirus son de interés para la terapia génica investigadores, en particular el virus del río Ross, virus Sindbis , virus del bosque Semliki , y la encefalitis equina venezolana virus han sido utilizados para desarrollar vectores virales para la entrega de genes. Son de particular interés los virus quiméricos que pueden formarse con envolturas alfavirales y cápsides retrovirales. Estas quimeras se denominan virus pseudotipados. Los pseudotipos de envoltura alfaviral de retrovirus o lentivirus pueden integrarselos genes que llevan a la amplia gama de células hospedadoras potenciales que son reconocidas e infectadas por las proteínas de la envoltura alfavírica E2 y E1. La integración estable de genes virales está mediada por los interiores retrovirales de estos vectores. Existen limitaciones para el uso de alfavirus en el campo de la terapia génica.Sin embargo, debido a su falta de dirección, a través de la introducción de dominios de anticuerpos variables en un bucle no conservado en la estructura de E2, se han dirigido poblaciones específicas de células. Además, el uso de alfavirus completos para terapia génica tiene una eficacia limitada tanto porque varias proteínas alfavirales internas están implicadas en la inducción de apoptosis tras la infección como porque la cápside alfaviral media solo la introducción transitoria de ARNm en las células huésped. Ninguna de estas limitaciones se extiende a los pseudotipos de la envoltura alfaviral de los retrovirus o lentivirus. Sin embargo, la expresión de las envolturas del virus Sindbis puede conducir a la apoptosis, y su introducción en las células huésped tras la infección por retrovirus pseudotipados de la envoltura del virus Sindbis también puede conducir a la muerte celular.La toxicidad de las envolturas virales de Sindbis puede ser la causa de los títulos de producción muy bajos obtenidos a partir de células de empaquetamiento construidas para producir pseudotipos de Sindbis. Otra rama de la investigación que involucra alfavirus es la vacunación. Los alfavirus pueden ser diseñados para crearvectores replicón que inducen eficazmente respuestas inmunes humorales y de células T. Por lo tanto, podrían usarse para vacunar contra antígenos virales, bacterianos, protozoarios y tumorales.
Historia [ editar ]
Inicialmente, la familia Togaviridae incluía lo que ahora se llama Flavivirus , dentro del género Alphavirus . Los flavivirus se formaron en su propia familia cuando se observaron suficientes diferencias con los alfavirus debido al desarrollo de la secuenciación. [14] El virus de la rubéola se incluyó anteriormente en la familia Togaviridae en su propio género Rubivirus , pero ahora se clasifica en su propia familia Matonaviridae . [15] Alphavirus es ahora el único género de la familia.
- 1930 - El virus de la encefalitis equina occidental se aísla por primera vez en los Estados Unidos (el primer alfavirus jamás aislado)
- 1933 - El virus de la encefalitis equina del este se aísla por primera vez en los Estados Unidos.
- 1938 - Se aísla la encefalitis equina venezolana .
- 1941 - Se observa una epidemia de encefalitis equina occidental en los Estados Unidos. Afecta a 300.000 caballos y 3.336 humanos.
- 1941 - Norman Gregg nota un gran número de niños con cataratas después de un brote de rubéola. Este y otros defectos se clasifican luego en el síndrome de rubéola congénita .
- 1942 - Se aísla el virus del bosque Semliki en Buliyama, condado de Bwamba, Uganda .
- 1952 - Se aísla el virus Sindbis en el distrito de salud de Sindbis, a 64 km al norte de El Cairo, Egipto .
- 1959 - El virus del río Ross se aísla de los mosquitos Aedes vigilax (ahora conocidos como Ochlerotatus vigilax ) [16] que quedaron atrapados en el río Ross en Australia.
- 1963 - El virus Ross River, que causa poliartritis epidémica (visto principalmente en Australia), es aislado por Doherty y sus colegas. [17]
- 1971 - Se observa la última epidemia de encefalitis equina venezolana en caballos en el sur de Texas . [18]
- 1986 - Se identifica el virus del bosque de Barmah como causante de enfermedades humanas en Australia. [19]
- 2001 - Los científicos resolvieron la estructura cristalina de la capa de glicoproteína del virus del bosque Semliki .
- 2005–2006 - Gran epidemia del virus chikungunya en la isla de La Reunión y las islas circundantes en el Océano Índico [20]
- 2006 - Gran epidemia del virus chikungunya en la India con más de 1,5 millones de casos notificados [21]
Ver también [ editar ]
- Infección por alfavirus
Fuentes [ editar ]
- "Arbovirus" . Virología en línea .
- "Fuentes de ICTV" . ICTV . Archivado desde el original el 12 de febrero de 2006.
- Vectores de alfavirus: de la producción de proteínas a la terapia génica, C Smerdou & P Liljestrom, Gene Therapy and Regulation Vol 1 No 1 2000 pp. 33–63
- Rayner JO, Dryga SA, Kamrud KI (2002). "Vectores de alfavirus y vacunación". Reseñas en Virología Médica . 12 (5): 279–96. doi : 10.1002 / rmv.360 . PMID 12211042 .
- https://web.archive.org/web/20070302184833/http://ep.physoc.org/cgi/content/full/90/1/45
- https://www.ncbi.nlm.nih.gov/books/NBK7633/
Referencias [ editar ]
- ^ Elrefaey AM, Abdelnabi R, Rosales Rosas AL, Wang L, Basu S, Delang L (31 de agosto de 2020). "Comprensión de los mecanismos subyacentes a la restricción de host de virus específicos de insectos" . Virus . 12 (9): 964. doi : 10.3390 / v12090964 .
- ^ Chen R, Mukhopadhyay S, Fondo A, Bolling B, Nasar F, Coffey LL, et al. (Junio de 2018). "Perfil de taxonomía de virus ICTV: Togaviridae" . La Revista de Virología General . 99 (6): 761–762. doi : 10.1099 / jgv.0.001072 . PMID 29745869 .
- ^ Vénien-Bryan C, Fuller SD (febrero de 1994). "La organización del complejo de picos del virus Semliki Forest". J. Mol. Biol . 236 (2): 572–83. doi : 10.1006 / jmbi.1994.1166 . PMID 8107141 .
- ^ Lescar J, Roussel A, Wien MW, Navaza J, Fuller SD, Wengler G, Wengler G, Rey FA (abril de 2001). "La cáscara de glicoproteína de fusión del virus Semliki Forest: un ensamblaje icosaédrico preparado para la activación fusogénica a pH endosomal". Celular . 105 (1): 137–48. doi : 10.1016 / S0092-8674 (01) 00303-8 . PMID 11301009 .
- ^ Shin G, Yost SA, Miller MT, Elrod EJ, Grakoui A, Marcotrigiano J (2012) Información estructural y funcional sobre el procesamiento y patogénesis de poliproteínas de alfavirus. Proc Natl Acad Sci EE. UU.
- ^ Forrester NL, Palacios G, Tesh RB, Savji N, Guzman H, Sherman M, Weaver SC, Lipkin WI (diciembre de 2011). "La filogenia a escala del genoma del género Alphavirus sugiere un origen marino" . J Virol . 86 (5): 2729–38. doi : 10.1128 / JVI.05591-11 . PMC 3302268 . PMID 22190718 .
- ^ Levinson RS, Strauss JH, Strauss EG (1990). "Secuencia completa del ARN genómico del virus O'nyong-nyong y su uso en la construcción de árboles filogenéticos de alfavirus". Virología . 175 (1): 110-123. doi : 10.1016 / 0042-6822 (90) 90191-s .
- ^ "Taxonomía de virus: versión 2020" . Comité Internacional de Taxonomía de Virus (ICTV). Marzo de 2021 . Consultado el 15 de mayo de 2021 .
- ^ Tschá MK, Suzukawa AA, Gräf T, Piancini LD, da Silva AM, Faoro H, et al. (2019). "Identificación de un nuevo alfavirus relacionado con los complejos de encefalitis que circulan en el sur de Brasil" . Microbios e infecciones emergentes . 8 (1): 920–933. doi : 10.1080 / 22221751.2019.1632152 . PMID 31237479 .
- ^ Powers AM, Brault AC, Shirako Y, Strauss EG, Kang W, Strauss JH, Weaver SC (noviembre de 2001). "Relaciones evolutivas y sistemática de los alfavirus" . J. Virol . 75 (21): 10118–31. doi : 10.1128 / JVI.75.21.10118-10131.2001 . PMC 114586 . PMID 11581380 .
- ^ Lundström JO, Pfeffer M (noviembre de 2010). "Estructura filogeográfica e historia evolutiva del virus Sindbis". Enfermedad zoonótica transmitida por vectores . 10 (9): 889–907. doi : 10.1089 / vbz.2009.0069 . PMID 20420530 .
- ^ Weaver SC, Hagenbaugh A, Bellew LA, Netesov SV, Volchkov VE, Chang GJ, Clarke DK, Gousset L, Scott TW, Trent DW (noviembre de 1993). "Una comparación de las secuencias de nucleótidos de los virus de la encefalomielitis equina oriental y occidental con las de otros alfavirus y virus de ARN relacionados". Virología . 197 (1): 375–90. doi : 10.1006 / viro.1993.1599 . PMID 8105605 .
- ^ Weaver SC, Rico-Hesse R, Scott TW (1992). "Diversidad genética y lentas tasas de evolución en alfavirus del Nuevo Mundo". Curr. Cima. Microbiol. Immunol . 176 : 99-117. PMID 1318187 .
- ^ "Togaviridae" . stanford.edu.
- ^ "Lista de taxonomía de ICTV" . Consultado el 5 de mayo de 2020 .
- ^ "Aedes vigilax" . Programa de Vigilancia de Arbovirus y Monitoreo de Vectores de Nueva Gales del Sur . Programa de Vigilancia de Arbovirus y Monitoreo de Mosquitos de Nueva Gales del Sur . Consultado el 5 de junio de 2010 .
Tenga en cuenta que 'Ochlerotatus vigilax' antes de 2000, se conocía como 'Aedes vigilax'
- ^ Doherty RL, Carley JG, Best JC (mayo de 1972). "Aislamiento del virus del río Ross del hombre". La Revista Médica de Australia . 1 (21): 1083–4. PMID 5040017 .
- ^ Calisher CH (enero de 1994). "Arbovirus médicamente importantes de Estados Unidos y Canadá" . Revisiones de microbiología clínica . 7 (1): 89-116. doi : 10.1128 / CMR.7.1.89 . PMC 358307 . PMID 8118792 .
- ^ Boughton CR, Hawkes RA, Naim HM (febrero de 1988). "Enfermedad causada por un virus similar al bosque de Barmah en Nueva Gales del Sur". La Revista Médica de Australia . 148 (3): 146–7. PMID 2828896 .
- ^ Tsetsarkin K, Higgs S, McGee CE, De Lamballerie X, Charrel RN, Vanlandingham DL (2006). "Clones infecciosos del virus Chikungunya (aislado de La Réunion) para estudios de competencia de vectores". Enfermedades zoonóticas y transmitidas por vectores . 6 (4): 325–37. doi : 10.1089 / vbz.2006.6.325 . PMID 17187566 .
- ^ Lahariya C, Pradhan SK (diciembre de 2006). "Aparición del virus chikungunya en el subcontinente indio después de 32 años: una revisión". Revista de enfermedades transmitidas por vectores . 43 (4): 151–60. PMID 17175699 .
Enlaces externos [ editar ]
- Informe de ICTV Togaviridae
- Viralzone : Alphavirus
- Recurso de análisis y base de datos de patógenos de virus (ViPR): Togaviridae