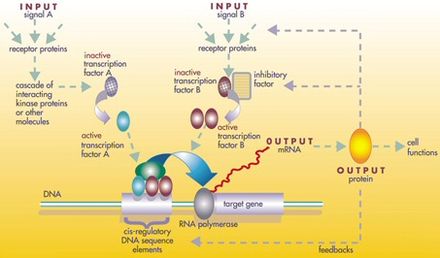
Una red reguladora de genes (o genética ) ( GRN ) es una colección de reguladores moleculares que interactúan entre sí y con otras sustancias en la célula para gobernar los niveles de expresión génica de ARNm y proteínas que, a su vez, determinan la función de la célula. . Los GRN también juegan un papel central en la morfogénesis , la creación de estructuras corporales, que a su vez es fundamental para la biología del desarrollo evolutivo (evo-devo).
El regulador puede ser ADN , ARN , proteínas y complejos de estos. La interacción puede ser directa o indirecta (a través de ARN transcrito o proteína traducida). En general, cada molécula de ARNm pasa a producir una proteína específica (o conjunto de proteínas). En algunos casos, esta proteína será estructural y se acumulará en la membrana celular o dentro de la célula para darle propiedades estructurales particulares. En otros casos, la proteína será una enzima , es decir, una micro-máquina que cataliza una determinada reacción, como la descomposición de una fuente de alimento o una toxina. Sin embargo, algunas proteínas solo sirven para activar otros genes, y estos son los factores de transcripción.que son los principales actores en las redes o cascadas regulatorias. Al unirse a la región promotora al comienzo de otros genes, los activan, iniciando la producción de otra proteína, y así sucesivamente. Algunos factores de transcripción son inhibidores. [1]
En los organismos unicelulares, las redes reguladoras responden al entorno externo, optimizando la célula en un momento dado para sobrevivir en este entorno. Por lo tanto, una célula de levadura, que se encuentra en una solución de azúcar, activará los genes para producir enzimas que procesan el azúcar en alcohol. [2] Este proceso, que asociamos con la elaboración del vino, es la forma en que la célula de levadura se gana la vida, ganando energía para multiplicarse, lo que en circunstancias normales mejoraría sus perspectivas de supervivencia.
En los animales multicelulares, el mismo principio se ha puesto al servicio de las cascadas de genes que controlan la forma del cuerpo. [3] Cada vez que una célula se divide, resultan dos células que, aunque contienen el mismo genoma en su totalidad, pueden diferir en qué genes se activan y producen proteínas. A veces, un "circuito de retroalimentación autosostenida" asegura que una célula mantenga su identidad y la transmita. Menos conocido es el mecanismo de la epigenética mediante el cual la modificación de la cromatina puede proporcionar memoria celular al bloquear o permitir la transcripción. Una característica importante de los animales multicelulares es el uso de morfógeno.gradientes, que de hecho proporcionan un sistema de posicionamiento que le dice a una célula en qué parte del cuerpo se encuentra y, por tanto, en qué tipo de célula debe convertirse. Un gen que se activa en una célula puede producir un producto que sale de la célula y se difunde a través de las células adyacentes, ingresando a ellas y activando los genes solo cuando está presente por encima de cierto nivel de umbral. Por lo tanto, estas células son inducidas a un nuevo destino e incluso pueden generar otros morfógenos que envían señales a la célula original. En distancias más largas, los morfógenos pueden utilizar el proceso activo de transducción de señales . Dicha señalización controla la embriogénesis , la construcción de un plan corporal.desde cero a través de una serie de pasos secuenciales. También controlan y mantienen los cuerpos de los adultos a través de procesos de retroalimentación , y la pérdida de dicha retroalimentación debido a una mutación puede ser responsable de la proliferación celular que se observa en el cáncer . Paralelamente a este proceso de construcción de estructuras, la cascada de genes activa genes que producen proteínas estructurales que le dan a cada célula las propiedades físicas que necesita.
Resumen [ editar ]
En un nivel, las células biológicas pueden considerarse como "bolsas parcialmente mezcladas" de sustancias químicas biológicas; en la discusión de las redes reguladoras de genes, estas sustancias químicas son principalmente los ARN mensajeros (ARNm) y proteínas que surgen de la expresión génica. Estos ARNm y proteínas interactúan entre sí con varios grados de especificidad. Algunos se difunden alrededor de la celda. Otros están unidos a las membranas celulares , interactuando con moléculas en el medio ambiente. Aún otros pasan a través de las membranas celulares y median señales de largo alcance a otras células en un organismo multicelular. Estas moléculas y sus interacciones comprenden una red reguladora de genes . Una red de regulación genética típica se parece a esto:
Los nodos de esta red pueden representar genes, proteínas, ARNm, complejos proteína / proteína o procesos celulares. Los nodos que se representan a lo largo de líneas verticales están asociados con las interfaces de celda / entorno, mientras que los otros son flotantes y pueden difundirse. Los bordes entre nodos representan interacciones entre los nodos, que pueden corresponder a reacciones moleculares individuales entre ADN, ARNm, miARN, proteínas o procesos moleculares a través de los cuales los productos de un gen afectan a los de otro, aunque la falta de información obtenida experimentalmente a menudo implica que algunos las reacciones no se modelan con un nivel de detalle tan fino. Estas interacciones pueden ser inductivas (generalmente representadas por puntas de flecha o el signo +), con un aumento en la concentración de una que conduce a un aumento en la otra, inhibitorias (representadas con círculos rellenos, flechas sin punta o el signo menos), con un aumento en uno conduce a una disminución en el otro, o dual, cuando dependiendo de las circunstancias el regulador puede activar o inhibir el nodo objetivo. Los nodos pueden regularse a sí mismos directa o indirectamente,creando ciclos de retroalimentación, que forman cadenas cíclicas de dependencias en la red topológica. La estructura de la red es una abstracción de la dinámica química o molecular del sistema, que describe las múltiples formas en que una sustancia afecta a todas las demás a las que está conectada. En la práctica, tales GRN se infieren de la literatura biológica sobre un sistema dado y representan una destilación del conocimiento colectivo sobre un conjunto de reacciones bioquímicas relacionadas. Para acelerar la curación manual de GRN, algunos esfuerzos recientes intentan utilizartales GRN se infieren de la literatura biológica sobre un sistema dado y representan una destilación del conocimiento colectivo sobre un conjunto de reacciones bioquímicas relacionadas. Para acelerar la curación manual de GRN, algunos esfuerzos recientes intentan utilizartales GRN se infieren de la literatura biológica sobre un sistema dado y representan una destilación del conocimiento colectivo sobre un conjunto de reacciones bioquímicas relacionadas. Para acelerar la curación manual de GRN, algunos esfuerzos recientes intentan utilizarminería de texto , bases de datos seleccionadas, inferencia de red a partir de datos masivos, verificación de modelos y otras tecnologías de extracción de información para este propósito. [4]
Los genes pueden verse como nodos en la red, siendo la entrada proteínas como los factores de transcripción y las salidas el nivel de expresión génica . El valor del nodo depende de una función que depende del valor de sus reguladores en los pasos de tiempo anteriores (en la red booleana descrita a continuación, estas son funciones booleanas , típicamente Y, O y NO). Estas funciones se han interpretado como una especie de procesamiento de información.dentro de la célula, lo que determina el comportamiento celular. Los impulsores básicos dentro de las células son las concentraciones de algunas proteínas, que determinan las coordenadas espaciales (ubicación dentro de la célula o tejido) y temporales (ciclo celular o etapa de desarrollo) de la célula, como una especie de "memoria celular". Las redes de genes apenas están comenzando a entenderse, y el siguiente paso de la biología es intentar deducir las funciones de cada "nodo" genético, para ayudar a comprender el comportamiento del sistema en niveles crecientes de complejidad, desde el gen hasta la vía de señalización. , nivel celular o tisular. [5]
Se han desarrollado modelos matemáticos de GRN para capturar el comportamiento del sistema que se está modelando y, en algunos casos, generar predicciones correspondientes a observaciones experimentales. En algunos otros casos, se ha demostrado que los modelos hacen predicciones novedosas y precisas, que pueden probarse experimentalmente, lo que sugiere nuevos enfoques para explorar en un experimento que a veces no se consideraría en el diseño del protocolo de un laboratorio experimental. Técnicas de modelado incluyen ecuaciones diferenciales (ODEs), redes de operadores booleanos , redes de Petri , redes bayesianas , gráficas modelos de red de Gauss , estocástico , y Proceso Calculi . [6]Por el contrario, se han propuesto técnicas para generar modelos de GRN que expliquen mejor un conjunto de observaciones de series de tiempo . Recientemente se ha demostrado que la señal de ChIP-seq de modificación de histonas está más correlacionada con motivos de factores de transcripción en los promotores en comparación con el nivel de ARN. [7] Por lo tanto, se propone que la modificación de histonas de series de tiempo ChIP-seq podría proporcionar una inferencia más confiable de redes reguladoras de genes en comparación con los métodos basados en niveles de expresión.
Estructura y evolución [ editar ]
Característica global [ editar ]
Generalmente se piensa que las redes reguladoras de genes están formadas por unos pocos nodos ( hubs ) altamente conectados y muchos nodos mal conectados anidados dentro de un régimen regulatorio jerárquico. Por tanto, las redes reguladoras de genes se aproximan a una topología de red libre de escala jerárquica . [8] Esto es consistente con la opinión de que la mayoría de los genes tienen pleiotropía limitada y operan dentro de módulos reguladores . [9] Se cree que esta estructura evoluciona debido a la unión preferencial de genes duplicados a genes más conectados. [8]Trabajos recientes también han demostrado que la selección natural tiende a favorecer las redes con escasa conectividad. [10]
Existen principalmente dos formas en que las redes pueden evolucionar, y ambas pueden ocurrir simultáneamente. La primera es que la topología de la red se puede cambiar mediante la adición o sustracción de nodos (genes) o partes de la red (módulos) se pueden expresar en diferentes contextos. La vía de señalización de Drosophila Hippo es un buen ejemplo. La vía de señalización del hipopótamo controla tanto el crecimiento mitótico como la diferenciación celular posmitótica. [11] Recientemente se descubrió que la red en la que opera la vía de señalización Hippo difiere entre estas dos funciones, lo que a su vez cambia el comportamiento de la vía de señalización Hippo. Esto sugiere que la vía de señalización de Hippo funciona como un módulo regulador conservado que se puede utilizar para múltiples funciones según el contexto. [11] Por lo tanto, cambiar la topología de la red puede permitir que un módulo conservado sirva para múltiples funciones y altere la salida final de la red. La segunda forma en que las redes pueden evolucionar es cambiando la fuerza de las interacciones entre los nodos, como la fuerza con la que un factor de transcripción puede unirse a un elemento regulador en cis. Se ha demostrado que tal variación en la fuerza de los bordes de la red subyace a la variación entre especies en el patrón de destino de las células de la vulva de los gusanos Caenorhabditis . [12]
Característica local [ editar ]
Otra característica ampliamente citada de la red reguladora de genes es su abundancia de ciertas subredes repetitivas conocidas como motivos de red . Los motivos de red se pueden considerar como patrones topológicos repetitivos cuando se divide una red grande en bloques pequeños. El análisis anterior encontró varios tipos de motivos que aparecían con más frecuencia en las redes reguladoras de genes que en las redes generadas al azar. [13] [14] [15] Como ejemplo, uno de esos motivos se llama bucles de alimentación hacia adelante, que constan de tres nodos. Este motivo es el más abundante entre todos los motivos posibles compuesto por tres nodos, como se muestra en las redes reguladoras de genes de moscas, nematodos y humanos. [15]
Se ha propuesto que los motivos enriquecidos sigan una evolución convergente , lo que sugiere que son "diseños óptimos" para ciertos propósitos regulatorios. [16] Por ejemplo, el modelado muestra que los bucles de retroalimentación son capaces de coordinar el cambio en el nodo A (en términos de concentración y actividad) y la dinámica de expresión del nodo C, creando diferentes comportamientos de entrada-salida. [17] [18] El sistema de utilización de galactosa de E. coli contiene un bucle de alimentación que acelera la activación del operón de utilización de galactosa galETK , lo que potencialmente facilita la transición metabólica a galactosa cuando se agota la glucosa. [19]El ciclo de retroalimentación en los sistemas de utilización de arabinosa de E. coli retrasa la activación del operón y transportadores del catabolismo de la arabinosa, evitando potencialmente la transición metabólica innecesaria debido a fluctuaciones temporales en las vías de señalización aguas arriba. [20] De manera similar, en la vía de señalización Wnt de Xenopus , el bucle de alimentación hacia adelante actúa como un detector de cambio de pliegue que responde al cambio de pliegue, en lugar del cambio absoluto, en el nivel de β-catenina, aumentando potencialmente la resistencia a fluctuaciones en los niveles de β-catenina. [21] Siguiendo la hipótesis de la evolución convergente, el enriquecimiento de los bucles de alimentación directa sería una adaptaciónpara una respuesta rápida y resistencia al ruido. Una investigación reciente encontró que la levadura cultivada en un ambiente de glucosa constante desarrolló mutaciones en las vías de señalización de la glucosa y la vía de regulación del crecimiento, lo que sugiere que los componentes reguladores que responden a los cambios ambientales son prescindibles en un ambiente constante. [22]
Por otro lado, algunos investigadores plantean la hipótesis de que el enriquecimiento de los motivos de la red no es adaptativo. [23] En otras palabras, las redes reguladoras de genes pueden evolucionar hacia una estructura similar sin la selección específica del comportamiento de entrada-salida propuesto. El apoyo a esta hipótesis a menudo proviene de simulaciones computacionales. Por ejemplo, las fluctuaciones en la abundancia de bucles de alimentación hacia adelante en un modelo que simula la evolución de las redes reguladoras de genes mediante el recableado aleatorio de nodos pueden sugerir que el enriquecimiento de los bucles de alimentación hacia adelante es un efecto secundario de la evolución. [24]En otro modelo de evolución de redes de reguladores de genes, la proporción de las frecuencias de duplicación de genes y deleción de genes muestra una gran influencia en la topología de la red: ciertas proporciones conducen al enriquecimiento de bucles de alimentación hacia adelante y crean redes que muestran características de redes libres de escala jerárquica. Se ha demostrado computacionalmente la evolución de novo de bucles de retroalimentación coherentes de tipo 1 en respuesta a la selección de su función hipotética de filtrar una señal espuria corta, lo que respalda la evolución adaptativa, pero para el ruido no idealizado, un sistema de alimentación basado en la dinámica En cambio, se favoreció la regulación directa con topología diferente. [25]
Redes reguladoras bacterianas [ editar ]
Las redes reguladoras permiten que las bacterias se adapten a casi todos los nichos ambientales del planeta. [26] [27] Las bacterias utilizan una red de interacciones entre diversos tipos de moléculas que incluyen ADN, ARN, proteínas y metabolitos para lograr la regulación de la expresión génica. En las bacterias, la función principal de las redes reguladoras es controlar la respuesta a los cambios ambientales, por ejemplo, el estado nutricional y el estrés ambiental. [28] Una organización compleja de redes permite al microorganismo coordinar e integrar múltiples señales ambientales. [26]
Modelado [ editar ]
Ecuaciones diferenciales ordinarias acopladas [ editar ]
Es común modelar una red de este tipo con un conjunto de ecuaciones diferenciales ordinarias acopladas (ODE) o SDE , que describen la cinética de reacción de las partes constituyentes. Supongamos que nuestra red reguladora tiene nodos, y representemos las concentraciones de las sustancias correspondientes en el momento . Entonces la evolución temporal del sistema se puede describir aproximadamente por
donde las funciones expresan la dependencia de de las concentraciones de otras sustancias presentes en la célula. Las funciones se derivan en última instancia de principios básicos de cinética química o expresiones simples derivadas de estos, por ejemplo, cinética enzimática de Michaelis-Menten . Por lo tanto, las formas funcionales de los se eligen generalmente como polinomios de bajo orden o funciones de Hill que sirven como ansatz para la dinámica molecular real. Luego, estos modelos se estudian utilizando las matemáticas de la dinámica no lineal . Información específica del sistema, como velocidad de reacciónconstantes y sensibilidades, se codifican como parámetros constantes. [29]
Resolviendo para el punto fijo del sistema:
para todos , se obtienen (posiblemente varios) perfiles de concentración de proteínas y ARNm que son teóricamente sostenibles (aunque no necesariamente estables ). Por tanto, los estados estacionarios de las ecuaciones cinéticas corresponden a tipos de células potenciales y las soluciones oscilatorias de la ecuación anterior a los tipos de células cíclicas naturales. La estabilidad matemática de estos atractores generalmente se puede caracterizar por el signo de derivadas superiores en puntos críticos, y luego corresponde a la estabilidad bioquímica del perfil de concentración. Puntos críticos y bifurcacionesen las ecuaciones corresponden a estados de celda críticos en los que pequeñas perturbaciones de estado o parámetros podrían cambiar el sistema entre uno de varios destinos de diferenciación estables. Las trayectorias corresponden al despliegue de vías biológicas y transitorios de las ecuaciones a eventos biológicos a corto plazo. Para una discusión más matemática, vea los artículos sobre no linealidad , sistemas dinámicos , teoría de la bifurcación y teoría del caos .
Red booleana [ editar ]
El siguiente ejemplo ilustra cómo una red booleana puede modelar un GRN junto con sus productos genéticos (las salidas) y las sustancias del medio ambiente que lo afectan (las entradas). Stuart Kauffman fue uno de los primeros biólogos en utilizar la metáfora de las redes booleanas para modelar redes reguladoras genéticas. [30] [31]
- Cada gen, cada entrada y cada salida están representados por un nodo en un gráfico dirigido en el que hay una flecha de un nodo a otro si y solo si hay un vínculo causal entre los dos nodos.
- Cada nodo del gráfico puede estar en uno de dos estados: activado o desactivado.
- Para un gen, "on" corresponde al gen que se expresa; para entradas y salidas, "apagado" corresponde a la sustancia presente.
- Se considera que el tiempo transcurre en pasos discretos. En cada paso, el nuevo estado de un nodo es una función booleana de los estados anteriores de los nodos con flechas apuntando hacia él.
La validez del modelo se puede probar comparando los resultados de la simulación con las observaciones de series de tiempo. Una validación parcial de un modelo de red booleano también puede provenir de probar la existencia prevista de una conexión reguladora aún desconocida entre dos factores de transcripción particulares, cada uno de los cuales son nodos del modelo. [32]
Redes continuas [ editar ]
Los modelos de red continua de GRN son una extensión de las redes booleanas descritas anteriormente. Los nodos todavía representan genes y las conexiones entre ellos influyen en la regulación de la expresión génica. Los genes en los sistemas biológicos muestran un rango continuo de niveles de actividad y se ha argumentado que el uso de una representación continua captura varias propiedades de las redes reguladoras de genes que no están presentes en el modelo booleano. [33] Formalmente, la mayoría de estos enfoques son similares a una red neuronal artificial , ya que las entradas a un nodo se resumen y el resultado sirve como entrada para una función sigmoidea, por ejemplo, [34] pero las proteínas a menudo controlan la expresión génica de forma sinérgica. , es decir, de forma no lineal. [35] Sin embargo, ahora existe un modelo de red continuo[36] que permite agrupar entradas a un nodo realizando así otro nivel de regulación. Este modelo está formalmente más cerca de una red neuronal recurrente de orden superior. El mismo modelo también se ha utilizado para imitar la evolución de la diferenciación celular [37] e incluso la morfogénesis multicelular. [38]
Redes de genes estocásticos [ editar ]
Resultados experimentales recientes [39] [40] han demostrado que la expresión génica es un proceso estocástico. Así, muchos autores están utilizando ahora el formalismo estocástico, después del trabajo de Arkin et al. [41] Los trabajos sobre la expresión de un solo gen [42] y pequeñas redes genéticas sintéticas, [43] [44] como el interruptor de palanca genético de Tim Gardner y Jim Collins , proporcionaron datos experimentales adicionales sobre la variabilidad fenotípica y la naturaleza estocástica del gen. expresión. Las primeras versiones de los modelos estocásticos de expresión génica implicaron solo reacciones instantáneas y fueron impulsadas por el algoritmo de Gillespie . [45]
Dado que algunos procesos, como la transcripción de genes, implican muchas reacciones y no se pueden modelar correctamente como una reacción instantánea en un solo paso, se propuso modelar estas reacciones como reacciones retardadas múltiples de un solo paso para tener en cuenta el tiempo que toma para todo el proceso debe estar completo. [46]
A partir de aquí, se propuso un conjunto de reacciones [47] que permiten generar GRNs. Luego, estos se simulan utilizando una versión modificada del algoritmo de Gillespie, que puede simular múltiples reacciones retardadas en el tiempo (reacciones químicas en las que a cada uno de los productos se le proporciona un retardo de tiempo que determina cuándo se lanzará en el sistema como un "producto terminado"). .
Por ejemplo, la transcripción básica de un gen se puede representar mediante la siguiente reacción de un solo paso (RNAP es la ARN polimerasa, RBS es el sitio de unión al ribosoma del ARN y Pro i es la región promotora del gen i ):
Además, parece haber una compensación entre el ruido en la expresión génica, la velocidad con la que los genes pueden cambiar y el costo metabólico asociado a su funcionamiento. Más específicamente, para cualquier nivel dado de costo metabólico, existe una compensación óptima entre el ruido y la velocidad de procesamiento y el aumento del costo metabólico conduce a mejores compensaciones entre velocidad y ruido. [48] [49] [50]
Un trabajo reciente propuso un simulador (SGNSim, Stochastic Gene Networks Simulator ), [51] que puede modelar GRN donde la transcripción y la traducción se modelan como múltiples eventos retardados y su dinámica es impulsada por un algoritmo de simulación estocástico (SSA) capaz de lidiar con múltiples eventos retrasados en el tiempo. Los retrasos de tiempo se pueden extraer de varias distribuciones y las velocidades de reacción de funciones complejas o de parámetros físicos. SGNSim puede generar conjuntos de GRN dentro de un conjunto de parámetros definidos por el usuario, como la topología. También se puede utilizar para modelar GRN específicos y sistemas de reacciones químicas. También se pueden modelar perturbaciones genéticas tales como deleciones de genes, sobreexpresión de genes, inserciones, mutaciones de cambio de marco.
El GRN se crea a partir de un gráfico con la topología deseada, imponiendo distribuciones de grado de entrada y de salida. Las actividades de los promotores de genes se ven afectadas por otros productos de expresión de genes que actúan como insumos, en forma de monómeros o combinados en multímeros y establecidos como directos o indirectos. A continuación, cada entrada directa se asigna a un sitio de operador y se puede permitir, o no, que diferentes factores de transcripción compitan por el mismo sitio de operador, mientras que las entradas indirectas reciben un objetivo. Finalmente, se asigna una función a cada gen, que define la respuesta del gen a una combinación de factores de transcripción (estado del promotor). Las funciones de transferencia (es decir, cómo responden los genes a una combinación de entradas) se pueden asignar a cada combinación de estados del promotor según se desee.
En otro trabajo reciente, se han desarrollado modelos multiescala de redes reguladoras de genes que se centran en aplicaciones de biología sintética. Se han utilizado simulaciones que modelan todas las interacciones biomoleculares en la transcripción, traducción, regulación e inducción de redes reguladoras de genes, guiando el diseño de sistemas sintéticos. [52]
Predicción [ editar ]
Otro trabajo se ha centrado en predecir los niveles de expresión génica en una red reguladora de genes. Los enfoques utilizados para modelar redes reguladoras de genes se han visto limitados a ser interpretables y, como resultado, generalmente son versiones simplificadas de la red. Por ejemplo, las redes booleanas se han utilizado debido a su simplicidad y capacidad para manejar datos ruidosos pero pierden información de datos al tener una representación binaria de los genes. Además, las redes neuronales artificiales omiten el uso de una capa oculta para que puedan ser interpretadas, perdiendo la capacidad de modelar correlaciones de orden superior en los datos. Usando un modelo que no está limitado a ser interpretable, se puede producir un modelo más preciso.Ser capaz de predecir las expresiones génicas con mayor precisión proporciona una forma de explorar cómo las drogas afectan un sistema de genes, así como de encontrar qué genes están interrelacionados en un proceso. Esto ha sido fomentado por la competencia DREAM.[53] que promueve una competencia por los mejores algoritmos de predicción. [54] Otro trabajo reciente ha utilizado redes neuronales artificiales con una capa oculta. [55]
Aplicaciones [ editar ]
Esclerosis múltiple [ editar ]
Hay tres clases de esclerosis múltiple: recurrente-remitente (EMRR), progresiva primaria (EMPP) y progresiva secundaria (EMPS). La red de regulación genética (GRN) juega un papel vital para comprender el mecanismo de la enfermedad en estas tres clases diferentes de esclerosis múltiple. [56]
Ver también [ editar ]
- Plan de cuerpo
- Módulo cis-regulatorio
- Genenetwork (base de datos)
- Morfógeno
- Operón
- Expresión sinóptica
- Biologia de sistemas
- Análisis ponderado de redes de coexpresión de genes
Referencias [ editar ]
- ^ Latchman DS (septiembre de 1996). "Factores de transcripción inhibitorios". La Revista Internacional de Bioquímica y Biología Celular . 28 (9): 965–74. doi : 10.1016 / 1357-2725 (96) 00039-8 . PMID 8930119 .
- ^ Lee TI, Rinaldi NJ, Robert F, Odom DT, Bar-Joseph Z, Gerber GK, et al. (Octubre de 2002). "Redes reguladoras de la transcripción en Saccharomyces cerevisiae" . Ciencia . Young Lab. 298 (5594): 799–804. Código Bibliográfico : 2002Sci ... 298..799L . doi : 10.1126 / science.1075090 . PMID 12399584 . S2CID 4841222 .
- ^ Davidson E, Levin M (abril de 2005). "Redes reguladoras de genes" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (14): 4935. Código bibliográfico : 2005PNAS..102.4935D . doi : 10.1073 / pnas.0502024102 . PMC 556010 . PMID 15809445 .
- ^ Leitner F, Krallinger M, Tripathi S, Kuiper M, Lægreid A, Valencia A (julio de 2013). "Minería de redes de transcripción cis-reguladoras de la literatura". Actas de BioLINK SIG 2013 : 5–12.
- ^ Azpeitia E, Muñoz S, González-Tokman D, Martínez-Sánchez ME, Weinstein N, Naldi A, Álvarez-Buylla ER, Rosenblueth DA, Mendoza L (febrero de 2017). "La combinación de las funcionalidades de los circuitos de retroalimentación es determinante para el número y tamaño de los atractores en redes booleanas similares a vías" . Informes científicos . 7 : 42023. Bibcode : 2017NatSR ... 742023A . doi : 10.1038 / srep42023 . PMC 5301197 . PMID 28186191 .
- ^ Banf, Michael; Rhee, Seung Y. (enero de 2017). "Inferencia computacional de redes reguladoras de genes: enfoques, limitaciones y oportunidades" . Biochimica et Biophysica Acta (BBA) - Mecanismos reguladores de genes . 1860 (1): 41–52. doi : 10.1016 / j.bbagrm.2016.09.003 . ISSN 1874-9399 . PMID 27641093 .
- ^ Kumar V, Muratani M, Rayan NA, Kraus P, Lufkin T, Ng HH, Prabhakar S (julio de 2013). "Procesamiento de señal uniforme y óptimo de datos de secuenciación profunda mapeados" . Biotecnología de la naturaleza . 31 (7): 615-22. doi : 10.1038 / nbt.2596 . PMID 23770639 .
- ↑ a b Barabási AL, Oltvai ZN (febrero de 2004). "Biología de la red: comprensión de la organización funcional de la célula". Reseñas de la naturaleza. Genética . 5 (2): 101–13. doi : 10.1038 / nrg1272 . PMID 14735121 . S2CID 10950726 .
- ^ Wagner GP, Zhang J (marzo de 2011). "La estructura pleiotrópica del mapa genotipo-fenotipo: la capacidad de evolución de organismos complejos". Reseñas de la naturaleza. Genética . 12 (3): 204-13. doi : 10.1038 / nrg2949 . PMID 21331091 . S2CID 8612268 .
- ^ Leclerc RD (agosto de 2008). "Supervivencia de los más dispersos: las redes de genes robustas son parsimoniosas" . Biología de sistemas moleculares . 4 (1): 213. doi : 10.1038 / msb.2008.52 . PMC 2538912 . PMID 18682703 .
- ^ a b Jukam D, Xie B, Rister J, Terrell D, Charlton-Perkins M, Pistillo D, Gebelein B, Desplan C, Cook T (octubre de 2013). "Retroalimentaciones opuestas en la vía del hipopótamo para el control del crecimiento y el destino neuronal" . Ciencia . 342 (6155): 1238016. doi : 10.1126 / science.1238016 . PMC 3796000 . PMID 23989952 .
- ^ Hoyos E, Kim K, Milloz J, Barkoulas M, Pénigault JB, Munro E, Félix MA (abril de 2011). "Variación cuantitativa en la señalización autocrina y diafonía de la vía en la red vulvar Caenorhabditis" . Biología actual . 21 (7): 527–38. doi : 10.1016 / j.cub.2011.02.040 . PMC 3084603 . PMID 21458263 .
- ^ Shen-Orr SS, Milo R, Mangan S, Alon U (mayo de 2002). "Motivos de red en la red de regulación transcripcional de Escherichia coli". Genética de la naturaleza . 31 (1): 64–8. doi : 10.1038 / ng881 . PMID 11967538 . S2CID 2180121 .
- ^ Lee TI, Rinaldi NJ, Robert F, Odom DT, Bar-Joseph Z, Gerber GK, Hannett NM, Harbison CT, Thompson CM, Simon I, Zeitlinger J, Jennings EG, Murray HL, Gordon DB, Ren B, Wyrick JJ , Tagne JB, Volkert TL, Fraenkel E, Gifford DK, Young RA (octubre de 2002). "Redes reguladoras de la transcripción en Saccharomyces cerevisiae". Ciencia . 298 (5594): 799–804. Código Bibliográfico : 2002Sci ... 298..799L . doi : 10.1126 / science.1075090 . PMID 12399584 . S2CID 4841222 .
- ^ a b Boyle AP, Araya CL, Brdlik C, Cayting P, Cheng C, Cheng Y, et al. (Agosto de 2014). "Análisis comparativo de información regulatoria y circuitos a través de especies distantes" . Naturaleza . 512 (7515): 453–6. Código Bibliográfico : 2014Natur.512..453B . doi : 10.1038 / nature13668 . PMC 4336544 . PMID 25164757 .
- ^ Conant GC, Wagner A (julio de 2003). "Evolución convergente de circuitos genéticos". Genética de la naturaleza . 34 (3): 264–6. doi : 10.1038 / ng1181 . PMID 12819781 . S2CID 959172 .
- ^ Mangan S, Alon U (octubre de 2003). "Estructura y función del motivo de la red de bucle de alimentación" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 100 (21): 11980–5. Código bibliográfico : 2003PNAS..10011980M . doi : 10.1073 / pnas.2133841100 . PMC 218699 . PMID 14530388 .
- ^ Goentoro L, Shoval O, Kirschner MW, Alon U (diciembre de 2009). "El bucle feedforward incoherente puede proporcionar detección de cambio de pliegues en la regulación de genes" . Célula molecular . 36 (5): 894–9. doi : 10.1016 / j.molcel.2009.11.018 . PMC 2896310 . PMID 20005851 .
- ^ Mangan S, Itzkovitz S, Zaslaver A, Alon U (marzo de 2006). "El bucle de avance incoherente acelera el tiempo de respuesta del sistema gal de Escherichia coli". Revista de Biología Molecular . 356 (5): 1073–81. CiteSeerX 10.1.1.184.8360 . doi : 10.1016 / j.jmb.2005.12.003 . PMID 16406067 .
- ^ Mangan S, Zaslaver A, Alon U (noviembre de 2003). "El bucle de retroalimentación coherente sirve como un elemento de retardo sensible a los signos en las redes de transcripción". Revista de Biología Molecular . 334 (2): 197-204. CiteSeerX 10.1.1.110.4629 . doi : 10.1016 / j.jmb.2003.09.049 . PMID 14607112 .
- ^ Goentoro L, Kirschner MW (diciembre de 2009). "La evidencia de que el cambio de pliegue, y no el nivel absoluto, de beta-catenina dicta la señalización de Wnt" . Célula molecular . 36 (5): 872–84. doi : 10.1016 / j.molcel.2009.11.017 . PMC 2921914 . PMID 20005849 .
- ^ Kvitek DJ, Sherlock G (noviembre de 2013). "El genoma completo, la secuenciación de toda la población revela que la pérdida de redes de señalización es la principal estrategia de adaptación en un entorno constante" . PLOS Genetics . 9 (11): e1003972. doi : 10.1371 / journal.pgen.1003972 . PMC 3836717 . PMID 24278038 .
- ^ Lynch M (octubre de 2007). "La evolución de las redes genéticas por procesos no adaptativos". Reseñas de la naturaleza. Genética . 8 (10): 803-13. doi : 10.1038 / nrg2192 . PMID 17878896 . S2CID 11839414 .
- ^ Cordero OX, Hogeweg P (octubre de 2006). "Circuitos de bucle de alimentación hacia adelante como efecto secundario de la evolución del genoma" . Biología Molecular y Evolución . 23 (10): 1931–6. doi : 10.1093 / molbev / msl060 . PMID 16840361 .
- ^ Xiong, Kun; Lancaster, Alex K .; Siegal, Mark L .; Masel, Joanna (3 de junio de 2019). "La regulación feed-forward evoluciona de forma adaptativa a través de la dinámica en lugar de la topología cuando hay ruido intrínseco" . Comunicaciones de la naturaleza . 10 (1): 2418. Bibcode : 2019NatCo..10.2418X . doi : 10.1038 / s41467-019-10388-6 . PMC 6546794 . PMID 31160574 .
- ^ a b Filloux AA, ed. (2012). Redes reguladoras bacterianas . Prensa Académica Caister . ISBN 978-1-908230-03-4.
- ^ R bruto, Beier D, eds. (2012). Sistemas de dos componentes en bacterias . Prensa Académica Caister . ISBN 978-1-908230-08-9.
- ^ Requena JM, ed. (2012). Respuesta al estrés en microbiología . Prensa Académica Caister . ISBN 978-1-908230-04-1.
- ^ Chu D, Zabet NR, Mitavskiy B (abril de 2009). "Modelos de unión del factor de transcripción: sensibilidad de las funciones de activación a los supuestos del modelo" (PDF) . Revista de Biología Teórica . 257 (3): 419–29. doi : 10.1016 / j.jtbi.2008.11.026 . PMID 19121637 .
- ^ Kauffman SA (1993). Los orígenes del orden . ISBN 978-0-19-505811-6.
- ^ Kauffman SA (marzo de 1969). "Estabilidad metabólica y epigénesis en redes genéticas construidas aleatoriamente". Revista de Biología Teórica . 22 (3): 437–67. doi : 10.1016 / 0022-5193 (69) 90015-0 . PMID 5803332 .
- ^ Lovrics A, Gao Y, Juhász B, Bock I, Byrne HM, Dinnyés A, Kovács KA (noviembre de 2014). "El modelado booleano revela nuevas conexiones reguladoras entre los factores de transcripción que orquestan el desarrollo de la médula espinal ventral" . PLOS ONE . 9 (11): e111430. Código Bibliográfico : 2014PLoSO ... 9k1430L . doi : 10.1371 / journal.pone.0111430 . PMC 4232242 . PMID 25398016 .
- ^ Vohradsky J (septiembre de 2001). "Modelo neuronal de la red genética" . La Revista de Química Biológica . 276 (39): 36168–73. doi : 10.1074 / jbc.M104391200 . PMID 11395518 .
- ^ Geard N, Wiles J (2005). "Un modelo de red de genes para el desarrollo de linajes celulares". Vida artificial . 11 (3): 249–67. CiteSeerX 10.1.1.1.4742 . doi : 10.1162 / 1064546054407202 . PMID 16053570 . S2CID 8664677 .
- ^ Schilstra MJ, Bolouri H (2 de enero de 2002). "Modelado de la regulación de la expresión génica en redes reguladoras genéticas" . Grupo de biocomputación, Universidad de Hertfordshire. Archivado desde el original el 13 de octubre de 2007.
- ^ Knabe JF, Nehaniv CL, Schilstra MJ, Quick T (2006). "Evolución de los relojes biológicos mediante redes de regulación genética". Actas de la Conferencia Artificial Life X (Alife 10) . MIT Press. págs. 15-21. CiteSeerX 10.1.1.72.5016 .
- ^ Knabe JF, Nehaniv CL, Schilstra MJ (2006). "Robustez evolutiva de la diferenciación en redes reguladoras genéticas". Actas del 7º taller alemán sobre vida artificial 2006 (GWAL-7) . Berlín: Akademische Verlagsgesellschaft Aka. págs. 75–84. CiteSeerX 10.1.1.71.8768 .
- ^ Knabe JF, Schilstra MJ, Nehaniv CL (2008). "Evolución y morfogénesis de organismos multicelulares diferenciados: gradientes de difusión generados de forma autónoma para información posicional" (PDF) . Vida artificial XI: Actas de la XI Conferencia Internacional sobre Simulación y Síntesis de Sistemas Vivientes . MIT Press.
- ^ Elowitz MB, Levine AJ, Siggia ED, Swain PS (agosto de 2002). "Expresión de genes estocásticos en una sola célula" (PDF) . Ciencia . 297 (5584): 1183–6. Código Bibliográfico : 2002Sci ... 297.1183E . doi : 10.1126 / science.1070919 . PMID 12183631 . S2CID 10845628 .
- ^ Blake WJ, KAErn M, Cantor CR, Collins JJ (abril de 2003). "Ruido en la expresión de genes eucariotas". Naturaleza . 422 (6932): 633–7. Código Bibliográfico : 2003Natur.422..633B . doi : 10.1038 / nature01546 . PMID 12687005 . S2CID 4347106 .
- ^ Arkin A, Ross J, McAdams HH (agosto de 1998). "Análisis cinético estocástico de la bifurcación de la vía de desarrollo en células de Escherichia coli infectadas con fago lambda" . Genética . 149 (4): 1633–48. PMC 1460268 . PMID 9691025 .
- ^ Raser JM, O'Shea EK (septiembre de 2005). "Ruido en la expresión génica: orígenes, consecuencias y control" . Ciencia . 309 (5743): 2010–3. Código bibliográfico : 2005Sci ... 309.2010R . doi : 10.1126 / science.1105891 . PMC 1360161 . PMID 16179466 .
- ^ Elowitz MB, Leibler S (enero de 2000). "Una red oscilatoria sintética de reguladores transcripcionales". Naturaleza . 403 (6767): 335–8. Código Bibliográfico : 2000Natur.403..335E . doi : 10.1038 / 35002125 . PMID 10659856 . S2CID 41632754 .
- ^ Gardner TS, Cantor CR, Collins JJ (enero de 2000). "Construcción de un interruptor de palanca genético en Escherichia coli". Naturaleza . 403 (6767): 339–42. Código Bibliográfico : 2000Natur.403..339G . doi : 10.1038 / 35002131 . PMID 10659857 . S2CID 345059 .
- ^ Gillespie DT (1976). "Un método general para simular numéricamente la evolución temporal estocástica de reacciones químicas acopladas". J. Comput. Phys . 22 (4): 403–34. Código bibliográfico : 1976JCoPh..22..403G . doi : 10.1016 / 0021-9991 (76) 90041-3 .
- ^ Roussel MR, Zhu R (diciembre de 2006). "Validación de un algoritmo para retardar la simulación estocástica de la transcripción y traducción en la expresión de genes procariotas". Biología física . 3 (4): 274–84. Código Bibliográfico : 2006PhBio ... 3..274R . doi : 10.1088 / 1478-3975 / 3/4/005 . PMID 17200603 .
- ^ Ribeiro A, Zhu R, Kauffman SA (noviembre de 2006). "Una estrategia de modelado general para redes reguladoras de genes con dinámica estocástica". Revista de Biología Computacional . 13 (9): 1630–9. doi : 10.1089 / cmb.2006.13.1630 . PMID 17147485 . S2CID 6629364 .
- ^ Zabet NR, Chu DF (junio de 2010). "Límites computacionales de genes binarios" . Revista de la Royal Society, Interface . 7 (47): 945–54. doi : 10.1098 / rsif.2009.0474 . PMC 2871807 . PMID 20007173 .
- ^ Chu DF, Zabet NR, Hone AN (mayo-junio de 2011). "Configuración de parámetros óptimos para el procesamiento de información en redes reguladoras de genes" (PDF) . Bio Systems . 104 (2-3): 99-108. doi : 10.1016 / j.biosystems.2011.01.006 . PMID 21256918 .
- ^ Zabet NR (septiembre de 2011). "Retroalimentación negativa y límites físicos de los genes". Revista de Biología Teórica . 284 (1): 82–91. arXiv : 1408.1869 . doi : 10.1016 / j.jtbi.2011.06.021 . PMID 21723295 . S2CID 14274912 .
- ^ Ribeiro AS, Lloyd-Price J (marzo de 2007). "SGN Sim, un simulador de redes genéticas estocásticas" . Bioinformática . 23 (6): 777–9. doi : 10.1093 / bioinformatics / btm004 . PMID 17267430 .
- ^ Kaznessis YN (noviembre de 2007). "Modelos de biología sintética" . Biología de sistemas BMC . 1 : 47. doi : 10.1186 / 1752-0509-1-47 . PMC 2194732 . PMID 17986347 .
- ^ "El proyecto DREAM" . Centro de la Universidad de Columbia para el análisis multiescala de redes genómicas y celulares (MAGNet).
- ^ Gustafsson M, Hörnquist M (febrero de 2010). "Predicción de la expresión génica por integración suave y el mejor rendimiento neto elástico del desafío de expresión génica DREAM3" . PLOS ONE . 5 (2): e9134. Código bibliográfico : 2010PLoSO ... 5.9134G . doi : 10.1371 / journal.pone.0009134 . PMC 2821917 . PMID 20169069 .
- ^ Smith MR, Clemente M, Martínez T, Snell Q (2010). "Predicción de la expresión génica de series de tiempo utilizando redes neuronales con capas ocultas" (PDF) . Actas del 7º Simposio de Biotecnología y Bioinformática (BIOT 2010) . págs. 67–69.
- ^ Gnanakkumaar, Perumal; Murugesan, Ram; Ahmed, Shiek SSJ (4 de septiembre de 2019). "Redes reguladoras de genes en células mononucleares periféricas revela módulos reguladores críticos y reguladores de la esclerosis múltiple" . Informes científicos . 9 (1): 12732. Bibcode : 2019NatSR ... 912732G . doi : 10.1038 / s41598-019-49124-x . PMC 6726613 . PMID 31484947 .
Lectura adicional [ editar ]
- Bolouri H, Bower JM (2001). Modelado computacional de redes genéticas y bioquímicas . Cambridge, Mass: MIT Press. ISBN 978-0-262-02481-5.
- Kauffman SA (marzo de 1969). "Estabilidad metabólica y epigénesis en redes genéticas construidas aleatoriamente". Revista de Biología Teórica . 22 (3): 437–67. doi : 10.1016 / 0022-5193 (69) 90015-0 . PMID 5803332 .
Enlaces externos [ editar ]
- Base de datos de factores de transcripción de plantas y plataforma de análisis y datos de regulación de transcripción de plantas
- Servicio web de código abierto para análisis GRN
- BIB: Navegador de interacciones biológicas de levadura
- Modelos gráficos gaussianos para datos del genoma : inferencia de redes de asociación de genes con GGM
- Una bibliografía sobre el aprendizaje de redes causales de interacciones genéticas , actualizada periódicamente, contiene cientos de enlaces a artículos de bioinformática, estadísticas y aprendizaje automático.
- https://web.archive.org/web/20060907074456/http://mips.gsf.de/proj/biorel/ BIOREL es un recurso basado en la web para la estimación cuantitativa del sesgo de la red genética en relación con la información disponible de la base de datos sobre actividad génica / función / propiedades / asociaciones / interactio.
- Evolución de los relojes biológicos mediante redes de regulación genética : página de información con código fuente modelo y subprograma Java.
- Redes genéticas diseñadas
- Tutorial: Algoritmos genéticos y su aplicación a la evolución artificial de redes reguladoras genéticas
- BEN: un recurso basado en la web para explorar las conexiones entre genes, enfermedades y otras entidades biomédicas
- Interacción proteína-proteína global y red de regulación génica de Arabidopsis thaliana