Pseudogén
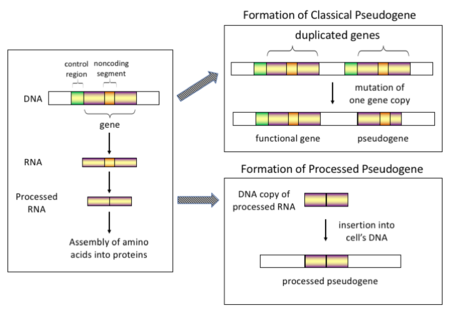
Los pseudogenes son segmentos de ADN no funcionales que se asemejan a los genes funcionales . La mayoría surgen como copias superfluas de genes funcionales, ya sea directamente por duplicación de ADN o indirectamente por transcripción inversa de una transcripción de ARNm . Los pseudogenes generalmente se identifican cuando el análisis de la secuencia del genoma encuentra secuencias similares a genes que carecen de secuencias reguladoras necesarias para la transcripción o traducción , o cuyas secuencias codificantes son obviamente defectuosas debido a cambios de marco o codones de parada prematuros .
La mayoría de los genomas no bacterianos contienen muchos pseudogenes, a menudo tantos como genes funcionales. Esto no es sorprendente, ya que se espera que varios procesos biológicos creen accidentalmente pseudogenes y no existen mecanismos especializados para eliminarlos de los genomas. Con el tiempo, los pseudogenes pueden eliminarse de sus genomas por casualidad en la replicación del ADN o por errores de reparación del ADN , o pueden acumular tantos cambios mutacionales que ya no son reconocibles como genes anteriores. El análisis de estos eventos de degeneración ayuda a aclarar los efectos de los procesos no selectivos en los genomas.
Las secuencias de pseudogenes pueden transcribirse en ARN a niveles bajos, debido a elementos promotores heredados del gen ancestral o que surgen por nuevas mutaciones. Aunque la mayoría de estas transcripciones no tendrán más importancia funcional que las transcripciones casuales de otras partes del genoma, algunas han dado lugar a ARN reguladores beneficiosos y nuevas proteínas.
Los pseudogenes se caracterizan habitualmente por una combinación de homología con un gen conocido y pérdida de alguna funcionalidad. Es decir, aunque cada pseudogen tiene una secuencia de ADN que es similar a algún gen funcional, generalmente no pueden producir productos proteicos finales funcionales. [1] A veces, los pseudogenes son difíciles de identificar y caracterizar en los genomas, porque los dos requisitos de homología y pérdida de funcionalidad generalmente están implícitos a través de alineamientos de secuencia en lugar de probados biológicamente.
Los pseudogenes para genes de ARN suelen ser más difíciles de descubrir, ya que no necesitan ser traducidos y, por lo tanto, no tienen "marcos de lectura".
Los pseudogenes pueden complicar los estudios de genética molecular. Por ejemplo, la amplificación de un gen por PCR puede amplificar simultáneamente un pseudogen que comparte secuencias similares. Esto se conoce como sesgo de PCR o sesgo de amplificación. De manera similar, los pseudogenes a veces se anotan como genes en las secuencias del genoma .





.jpg/440px-Drosophila_melanogaster_-_side_(aka).jpg)
