| Familia ubiquitina | ||||||||
|---|---|---|---|---|---|---|---|---|
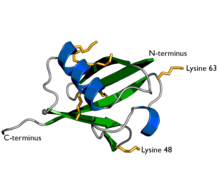 Un diagrama de ubiquitina . Las siete cadenas laterales de lisina se muestran en amarillo / naranja. | ||||||||
| Identificadores | ||||||||
| Símbolo | ubiquitina | |||||||
| Pfam | PF00240 | |||||||
| InterPro | IPR000626 | |||||||
| PROSITE | PDOC00271 | |||||||
| SCOP2 | 1aar / SCOPe / SUPFAM | |||||||
| ||||||||
La ubiquitina es una proteína reguladora pequeña (8,6 kDa ) que se encuentra en la mayoría de los tejidos de los organismos eucariotas , es decir, se encuentra de forma ubicua . Fue descubierto en 1975 [1] por Gideon Goldstein y caracterizado a finales de los años setenta y ochenta. [2] Cuatro genes en el genoma humano codifican la ubiquitina: UBB , UBC , UBA52 y RPS27A . [3]
La adición de ubiquitina a una proteína sustrato se llama ubiquitylation (o, alternativamente, ubiquitinación o ubiquitinylation ). La ubiquitilación afecta a las proteínas de muchas maneras: puede marcarlas para su degradación a través del proteasoma , alterar su ubicación celular , afectar su actividad y promover o prevenir interacciones de proteínas . [4] [5] [6] La ubiquitilación implica tres pasos principales: activación, conjugación y ligadura, realizada por enzimas activadoras de ubiquitina (E1), enzimas conjugadoras de ubiquitina (E2) y ubiquitina ligasas.(E3s), respectivamente. El resultado de esta secuencial cascada es enlazar ubiquitina a lisina residuos en el sustrato de proteína a través de un enlace isopeptídico , cisteína residuos a través de un tioéster de bonos , serina y treonina residuos a través de un enlace éster , o el grupo amino del de la proteína N-terminal a través de una enlace peptídico . [7] [8] [9]
Las modificaciones de la proteína pueden ser una sola proteína de ubiquitina (monoubiquitilación) o una cadena de ubiquitina (poliubiquitilación). Las moléculas de ubiquitina secundarias siempre están unidas a uno de los siete residuos de lisina o la metionina N-terminal de la molécula de ubiquitina anterior. Estos residuos de 'enlace' están representados por una "K" o "M" (la notación de aminoácidos de una letra de lisina y metionina, respectivamente) y un número, que se refiere a su posición en la molécula de ubiquitina como en K48, K29 o M1. . La primera molécula de ubiquitina se une covalentemente a través de su terminal Cgrupo carboxilato a una lisina, cisteína, serina, treonina o N-terminal particular de la proteína diana. La poliubiquitilación se produce cuando el extremo C de otra ubiquitina se une a uno de los siete residuos de lisina o la primera metionina de la molécula de ubiquitina añadida previamente, creando una cadena. Este proceso se repite varias veces, lo que lleva a la adición de varias ubiquitinas. Solo la poliubiquitilación en lisinas definidas, principalmente en K48 y K29, está relacionada con la degradación por parte del proteasoma (conocido como el "beso molecular de la muerte"), mientras que otras poliubiquitilaciones (por ejemplo, en K63, K11, K6 y M1) y monoubiquitilaciones pueden regular Procesos como el tráfico endocítico , la inflamación , la traducción y la reparación del ADN.. [10]
El descubrimiento de que las cadenas de ubiquitina se dirigen a las proteínas del proteasoma, que degrada y recicla las proteínas, fue galardonado con el Premio Nobel de Química en 2004. [8] [11] [12]
Identificación

La ubiquitina (originalmente, polipéptido inmunopoyético ubicuo ) se identificó por primera vez en 1975 [1] como una proteína de 8,6 kDa expresada en todas las células eucariotas . Las funciones básicas de la ubiquitina y los componentes de la vía de ubiquitilación fueron aclaradas a principios de la década de 1980 en el Technion por Aaron Ciechanover , Avram Hershko e Irwin Rose, por lo que se otorgó el Premio Nobel de Química en 2004. [11]
El sistema de ubiquitilación se caracterizó inicialmente como un sistema proteolítico dependiente de ATP presente en extractos celulares. Se encontró que un polipéptido termoestable presente en estos extractos, el factor de proteólisis dependiente de ATP 1 (APF-1), se unía covalentemente a la lisozima del sustrato de la proteína modelo en un proceso dependiente de ATP y Mg 2+ . [13] Varias moléculas de APF-1 se unieron a una única molécula de sustrato mediante un isopéptido.enlace, y se encontró que los conjugados se degradaban rápidamente con la liberación de APF-1 libre. Poco después de caracterizar la conjugación de APF-1-proteína, se identificó APF-1 como ubiquitina. El grupo carboxilo del residuo de glicina C-terminal de ubiquitina (Gly76) se identificó como el resto conjugado con los residuos de lisina del sustrato .
La proteína
| Número de residuos | 76 |
|---|---|
| Masa molecular | 8564.8448 Da |
| Punto isoeléctrico (pI) | 6,79 |
| Nombres de genes | RPS27A (UBA80, UBCEP1), UBA52 (UBCEP2), UBB , UBC |
| Secuencia (una sola letra ) | MQIFV K TLTG K TITLEVEPSDTIENV K A K IQD K EGIPPD QQRLIFAG K QLEDGRTLSDYNIQ K ESTLHLVLRLRGG |
La ubiquitina es una pequeña proteína que existe en todas las células eucariotas . Realiza sus innumerables funciones mediante la conjugación con una amplia gama de proteínas diana. Pueden ocurrir una variedad de modificaciones diferentes . La proteína ubiquitina en sí consta de 76 aminoácidos y tiene una masa molecular de aproximadamente 8,6 kDa. Las características clave incluyen su cola C-terminal y los 7 residuos de lisina . Está muy conservado a lo largo de la evolución eucariota; la ubiquitina humana y de levadura comparten una identidad de secuencia del 96% . [ cita requerida ]
Genes
La ubiquitina está codificada en mamíferos por 4 genes diferentes. Los genes UBA52 y RPS27A codifican una única copia de ubiquitina fusionada con las proteínas ribosómicas L40 y S27a, respectivamente. Los genes UBB y UBC codifican proteínas precursoras de poliubiquitina. [3]
Ubicuidad
La ubiquitilación (también conocida como ubiquitinación o ubiquitinilación) es una modificación enzimática postraduccional en la que una proteína de ubiquitina se une a una proteína sustrato . Este proceso generalmente une el último aminoácido de ubiquitina ( glicina 76) a un residuo de lisina en el sustrato. Se forma un enlace isopéptido entre el grupo carboxilo (COO - ) de la glicina de la ubiquitina y el grupo épsilon- amino (ε- NH+
3) de la lisina del sustrato. [14] La escisión con tripsina de un sustrato conjugado con ubiquitina deja un "remanente" de di-glicina que se utiliza para identificar el sitio de ubiquitilación. [15] [16] La ubiquitina también se puede unir a otros sitios en una proteína que son nucleófilos ricos en electrones , denominada " ubiquitilación no canónica". [9] Esto se observó por primera vez con el grupo amina del extremo N-terminal de una proteína que se utiliza para la ubiquitilación, en lugar de un residuo de lisina, en la proteína MyoD [17] y se ha observado desde entonces en otras 22 proteínas en múltiples especies, [18 ] [19] [20] [21] [22][23] [24] [25] [26] [27] [28] [29] [30] [31] [32] [33] [34] [35] [36] incluida la ubiquitina misma. [37] [38] También hay evidencia creciente de residuos que no son de lisina como objetivos de ubiquitilación utilizando grupos no amina, como el grupo sulfhidrilo en la cisteína, [33] [34] [39] [40] [41] [42] [ 43] [44] [45] [46] y elgrupo hidroxilo en treonina y serina. [33] [34] [39] [45] [46] [47][48] [49] [50] El resultado final de este proceso es la adición de una molécula de ubiquitina (monoubiquitilación) o una cadena de moléculas de ubiquitina (poliubiquitinación) a la proteína sustrato. [51]
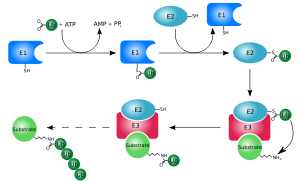
La ubiquitinación requiere tres tipos de enzima: las enzimas ubiquitina-activación , ubiquitina-conjugación de enzimas , y ubiquitina ligasas , conocidos como E1s, E2, y E3, respectivamente. El proceso consta de tres pasos principales:
- Activación: la ubiquitina se activa en una reacción de dos pasos por una enzima activadora de ubiquitina E1 , que depende de ATP . El paso inicial implica la producción de un intermedio de ubiquitina-adenilato. El E1 se une tanto a ATP como a ubiquitina y cataliza la acil-adenilación del extremo C-terminal de la molécula de ubiquitina. El segundo paso transfiere ubiquitina a un residuo de cisteína del sitio activo , con liberación de AMP . Este paso da como resultado un enlace tioéster entre el grupo carboxilo C-terminal de ubiquitina y el grupo sulfhidrilo de cisteína E1 . [14] [52]El genoma humano contiene dos genes que producen enzimas capaces de activar la ubiquitina: UBA1 y UBA6 . [53]
- Conjugación: las enzimas conjugadoras de ubiquitina E2 catalizan la transferencia de ubiquitina de E1 a la cisteína del sitio activo de E2 mediante una reacción de trans (tio) esterificación. Para realizar esta reacción, el E2 se une tanto a la ubiquitina activada como a la enzima E1. Los seres humanos poseen 35 enzimas E2 diferentes, mientras que otros organismos eucariotas tienen entre 16 y 35. Se caracterizan por su estructura altamente conservada, conocida como el pliegue catalítico de conjugación de ubiquitina (UBC). [54]
 Glicina y lisina unidas por un enlace isopéptido. El enlace isopéptido se resalta en amarillo.
Glicina y lisina unidas por un enlace isopéptido. El enlace isopéptido se resalta en amarillo. - Ligación: las ligasas de ubiquitina E3 catalizan el paso final de la cascada de ubiquitinación. Más comúnmente, crean un enlace isopéptido entre una lisina de la proteína diana y la glicina C-terminal de la ubiquitina. En general, este paso requiere la actividad de uno de los cientos de E3. Las enzimas E3 funcionan como módulos de reconocimiento de sustrato del sistema y son capaces de interactuar tanto con E2 como con el sustrato . Algunas enzimas E3 también activan las enzimas E2. Las enzimas E3 poseen uno de dos dominios : el homólogo al dominio del terminal carboxilo E6-AP ( HECT ) y el nuevo gen realmente interesante ( RING) dominio (o el dominio U-box estrechamente relacionado). Los dominios E3 de HECT se unen transitoriamente a ubiquitina en este proceso (se forma un intermedio tioéster obligado con la cisteína del sitio activo de E3), mientras que los dominios E3 de RING catalizan la transferencia directa de la enzima E2 al sustrato. [55] El complejo promotor de anafase (APC) y el complejo SCF (para el complejo proteico Skp1-Cullin-F-box) son dos ejemplos de E3 de múltiples subunidades involucradas en el reconocimiento y ubiquitinación de proteínas diana específicas para su degradación por el proteasoma . [56]
En la cascada de ubiquitinación, E1 puede unirse con muchos E2, que pueden unirse con cientos de E3 de forma jerárquica. Tener niveles dentro de la cascada permite una regulación estricta de la maquinaria de ubiquitinación. [7] Otras proteínas similares a la ubiquitina (UBL) también se modifican a través de la cascada E1-E2-E3, aunque existen variaciones en estos sistemas. [57]
Las enzimas E4, o factores de elongación de la cadena de ubiquitina, son capaces de agregar cadenas de poliubiquitina preformadas a las proteínas del sustrato. [58] Por ejemplo, la monoubiquitilación múltiple del supresor de tumores p53 por Mdm2 [59] puede ir seguida de la adición de una cadena de poliubiquitina usando p300 y CBP . [60] [61]
Tipos
La ubiquitinación afecta el proceso celular regulando la degradación de proteínas (a través del proteasoma y lisosoma ), coordinando la localización celular de proteínas, activando e inactivando proteínas y modulando las interacciones proteína-proteína . [4] [5] [6] Estos efectos están mediados por diferentes tipos de ubiquitinación de sustrato, por ejemplo, la adición de una sola molécula de ubiquitina (monoubiquitinación) o diferentes tipos de cadenas de ubiquitina (poliubiquitinación). [62]
Monoubiquitination
La monoubiquitinación es la adición de una molécula de ubiquitina a un residuo de proteína sustrato. La multi-monoubiquitinación es la adición de una molécula de ubiquitina a múltiples residuos de sustrato. La monoubiquitinación de una proteína puede tener efectos diferentes a la poliubiquitinación de la misma proteína. Se cree que es necesaria la adición de una sola molécula de ubiquitina antes de la formación de cadenas de poliubiquitina. [62] La monoubiquitinación afecta los procesos celulares como el tráfico de membranas , la endocitosis y la gemación viral . [10] [63]
Cadenas de poliubiquitina
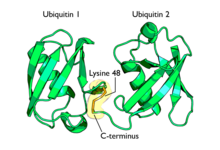

La poliubiquitinación es la formación de una cadena de ubiquitina en un solo residuo de lisina en la proteína sustrato. Después de la adición de un único resto de ubiquitina a un sustrato de proteína, se pueden agregar más moléculas de ubiquitina a la primera, produciendo una cadena de poliubiquitina. [62] Estas cadenas se forman uniendo el residuo de glicina de una molécula de ubiquitina a una lisina de ubiquitina unida a un sustrato. La ubiquitina tiene siete residuos de lisina y un extremo N que sirve como puntos de ubiquitinación; son K6, K11, K27, K29, K33, K48, K63 y M1, respectivamente. [8]Las cadenas unidas a lisina 48 fueron las primeras identificadas y son el tipo mejor caracterizado de cadena de ubiquitina. Las cadenas de K63 también se han caracterizado bien, mientras que la función de otras cadenas de lisina, cadenas mixtas, cadenas ramificadas, cadenas lineales unidas a M1 y cadenas heterólogas (mezclas de ubiquitina y otras proteínas similares a la ubiquitina) permanece menos clara. [16] [38] [62] [63] [64]
Las cadenas de poliubiquitina unidas a lisina 48 se dirigen a las proteínas para su destrucción, mediante un proceso conocido como proteólisis . Las cadenas de ubiquitina múltiple de al menos cuatro moléculas de ubiquitina de largo deben unirse a un residuo de lisina en la proteína condenada para que sea reconocida por el proteasoma 26S . [65] Se trata de una estructura en forma de barril que comprende un núcleo proteolítico central compuesto por cuatro estructuras de anillo, flanqueadas por dos cilindros que permiten selectivamente la entrada de proteínas ubiquitinadas. Una vez dentro, las proteínas se degradan rápidamente en pequeños péptidos (generalmente de 3 a 25 residuos de aminoácidos de longitud). Las moléculas de ubiquitina se escinden de la proteína inmediatamente antes de su destrucción y se reciclan para su uso posterior. [66]Aunque la mayoría de los sustratos proteicos están ubiquitinados, existen ejemplos de proteínas no ubiquitinadas dirigidas al proteasoma. [67] Las cadenas de poliubiquitina son reconocidas por una subunidad del proteasoma: S5a / Rpn10. Esto se logra mediante un motivo de interacción con ubiquitina (UIM) que se encuentra en un parche hidrofóbico en la región C-terminal de la unidad S5a / Rpn10. [4]
Las cadenas unidas a lisina 63 no están asociadas con la degradación proteasomal de la proteína sustrato. En cambio, permiten la coordinación de otros procesos como el tráfico endocítico , la inflamación , la traducción y la reparación del ADN . [10] En las células, las cadenas unidas a lisina 63 están unidas por el complejo ESCRT-0 , que evita su unión al proteasoma. Este complejo contiene dos proteínas, Hrs y STAM1, que contienen un UIM, que le permite unirse a cadenas unidas a lisina 63. [68] [69]
Se sabe menos acerca de las cadenas de ubiquitina atípicas (no unidas a lisina 48), pero la investigación está comenzando a sugerir funciones para estas cadenas. [63] Existe evidencia que sugiere que las cadenas atípicas unidas por lisina 6, 11, 27, 29 y metionina 1 pueden inducir la degradación proteasomal. [67] [70]
Se pueden formar cadenas de ubiquitina ramificadas que contienen múltiples tipos de enlaces. [71] Se desconoce la función de estas cadenas. [8]
Estructura
Las cadenas unidas de manera diferente tienen efectos específicos sobre la proteína a la que están unidas, causados por diferencias en las conformaciones de las cadenas de proteínas. Las cadenas unidas a K29, K33, [72] K63 y M1 tienen una conformación bastante lineal; se conocen como cadenas de conformación abierta. Las cadenas unidas a K6, K11 y K48 forman conformaciones cerradas. Las moléculas de ubiquitina en cadenas de conformación abierta no interactúan entre sí, excepto por los enlaces isopéptidos covalentes que las unen. Por el contrario, las cadenas de conformación cerradas tienen interfaces con residuos que interactúan. La alteración de las conformaciones de la cadena expone y oculta diferentes partes de la proteína ubiquitina, y los diferentes enlaces son reconocidos por proteínas que son específicas para las topologías únicas.que son intrínsecos al vínculo. Las proteínas pueden unirse específicamente a la ubiquitina a través de dominios de unión a ubiquitina (UBD). Las distancias entre las unidades de ubiquitina individuales en las cadenas difieren entre las cadenas de lisina 63 y 48 unidas. Los UBD explotan esto al tener pequeños espaciadores entre los motivos que interactúan con la ubiquitina que se unen a las cadenas unidas a lisina 48 (cadenas compactas de ubiquitina) y espaciadores más grandes para las cadenas unidas a la lisina 63. La maquinaria involucrada en el reconocimiento de cadenas de poliubiquitina también puede diferenciar entre cadenas unidas a K63 y cadenas unidas a M1, demostrado por el hecho de que estas últimas pueden inducir la degradación proteasomal del sustrato. [8] [10] [70]
Función
El sistema de ubiquitinación funciona en una amplia variedad de procesos celulares, que incluyen: [73]
- Procesamiento de antígenos
- Apoptosis
- Biogénesis de orgánulos
- Ciclo y división celular
- Transcripción y reparación de ADN
- Diferenciación y desarrollo
- Respuesta inmune e inflamación
- Degeneración neural y muscular.
- Mantenimiento de la pluripotencia [74]
- Morfogénesis de redes neuronales
- Modulación de los receptores de la superficie celular, los canales iónicos y la vía secretora.
- Respuesta al estrés y moduladores extracelulares.
- Biogénesis del ribosoma
- Infección viral
Proteínas de membrana
La multimonobiquitinación puede marcar proteínas transmembrana (por ejemplo, receptores ) para su eliminación de las membranas (internalización) y cumplir varias funciones de señalización dentro de la célula. Cuando las moléculas transmembrana de la superficie celular se marcan con ubiquitina, la localización subcelular de la proteína se altera, y a menudo se dirige a la proteína para su destrucción en los lisosomas. Esto sirve como un mecanismo de retroalimentación negativa, porque a menudo la estimulación de los receptores por ligandos aumenta su tasa de ubiquitinación e internalización. Al igual que la monoubiquitinación, las cadenas de poliubiquitina unidas a lisina 63 también tienen un papel en el tráfico de algunas proteínas de membrana. [10] [62] [65] [75]
Sistema Fougaro
El sistema de fougaro (griego; Fougaro, chimenea) es un sistema de suborganelos en el núcleo que puede ser un mecanismo para reciclar o eliminar moléculas de la célula al medio externo. Las moléculas o péptidos se ubiquitinan antes de ser liberados del núcleo de las células. Las moléculas ubiquitinadas se liberan de forma independiente o asociadas con proteínas endosomales como Beclin. [76]
Mantenimiento genómico
El antígeno nuclear de células proliferantes (PCNA) es una proteína involucrada en la síntesis de ADN . En condiciones fisiológicas normales, el PCNA está sumoilado (una modificación postraduccional similar a la ubiquitinación). Cuando el ADN se daña por radiación ultravioleta o sustancias químicas, la molécula SUMO que está unida a un residuo de lisina es reemplazada por ubiquitina. El PCNA monoubiquitinado recluta polimerasas que pueden llevar a cabo la síntesis de ADN con ADN dañado; pero esto es muy propenso a errores, lo que posiblemente resulte en la síntesis de ADN mutado. La poliubiquitinación de PCNA ligada a lisina 63 le permite realizar un bypass de mutación menos propenso a errores conocido por la ruta de cambio de plantilla. [6][77] [78]
La ubiquitinación de la histona H2AX está involucrada en el reconocimiento del daño del ADN de las roturas de la doble hebra del ADN. Las cadenas de poliubiquitina unidas a lisina 63 se forman en la histona H2AX por el par de ligasa E2 / E3 , Ubc13-Mms2 / RNF168. [79] [80] Esta cadena K63 parece reclutar RAP80, que contiene un UIM , y RAP80 luego ayuda a localizar BRCA1 . Esta vía eventualmente reclutará las proteínas necesarias para la reparación de la recombinación homóloga . [81]
Regulación transcripcional
Las histonas pueden ubiquitinarse y esto suele ser en forma de monoubiquitination (aunque sí ocurren formas poliubiquitinadas). La ubiquitinación de histonas altera la estructura de la cromatina y permite el acceso de enzimas involucradas en la transcripción. La ubiquitina en las histonas también actúa como un sitio de unión para proteínas que activan o inhiben la transcripción y también pueden inducir modificaciones posteriores a la traducción de la proteína. Todos estos efectos pueden modular la transcripción de genes. [82] [83]
Deubiquitinación
Las enzimas desubiquitinantes (DUB) se oponen al papel de la ubicuinación al eliminar la ubiquitina de las proteínas del sustrato. Son cisteína proteasas que rompen el enlace amida entre las dos proteínas. Son muy específicas, al igual que las ligasas E3 que unen la ubiquitina, con solo unos pocos sustratos por enzima. Pueden escindir tanto isopéptidos (entre ubiquitina y lisina) como enlaces peptídicos (entre ubiquitina y el extremo N-terminal). Además de eliminar la ubiquitina de las proteínas del sustrato, los DUB tienen muchas otras funciones dentro de la célula. La ubiquitina se expresa como múltiples copias unidas en una cadena (poliubiquitina) o unidas a subunidades ribosómicas. Los DUB escinden estas proteínas para producir ubiquitina activa. También reciclan la ubiquitina que se ha unido a pequeñas moléculas nucleofílicas durante el proceso de ubiquitinación. La monoubiquitina está formada por DUB que escinden la ubiquitina de las cadenas de poliubiquitina libres que se han eliminado previamente de las proteínas. [84] [85]
Dominios de unión a ubiquitina
| Dominio | Numero de Proteinas en el proteoma | Largo (aminoácidos) | Unión de ubiquitina Afinidad |
|---|---|---|---|
| SEÑAL | S. cerevisiae : 7 H. sapiens : 21 | 42–43 | ~ 2–160 μM |
| GATII | S. cerevisiae : 2 H. sapiens : 14 | 135 | ~ 180 μM |
| PEGAMENTO | S. cerevisiae :? H. sapiens :? | ~ 135 | ~ 460 μM |
| NZF | S. cerevisiae : 1 H. sapiens : 25 | ~ 35 | ~ 100–400 μM |
| PAZ | S. cerevisiae : 5 H. sapiens : 16 | ~ 58 | No conocida |
| UBA | S. cerevisiae : 10 H. sapiens : 98 | 45–55 | ~ 0.03–500 μM |
| UEV | S. cerevisiae : 2 H. sapiens :? | ~ 145 | ~ 100–500 μM |
| UIM | S. cerevisiae : 8 H. sapiens : 71 | ~ 20 | ~ 100–400 μM |
| VHS | S. cerevisiae : 4 H. sapiens : 28 | 150 | No conocida |
Los dominios de unión a ubiquitina (UBD) son dominios proteicos modulares que se unen de forma no covalente a la ubiquitina, estos motivos controlan varios eventos celulares. Se conocen estructuras moleculares detalladas para una serie de UBD, la especificidad de unión determina su mecanismo de acción y regulación, y cómo regula las proteínas y los procesos celulares. [86] [87]
Asociaciones de enfermedades
Patogenia
La vía de la ubiquitina se ha implicado en la patogenia de una amplia gama de enfermedades y trastornos que incluyen: [88]
- Neurodegeneración
- Infección e inmunidad
- Desordenes genéticos
- Cáncer
Neurodegeneración
La ubiquitina está implicada en enfermedades neurodegenerativas asociadas con la disfunción Proteostasis, incluyendo la enfermedad de Alzheimer , la enfermedad de la neurona motora , [89] la enfermedad de Huntington y la enfermedad de Parkinson . [90] Las variantes de la transcripción que codifican diferentes isoformas de ubiquilina-1 se encuentran en lesiones asociadas con la enfermedad de Alzheimer y Parkinson . [91] Se ha demostrado que niveles más altos de ubiquilina en el cerebro disminuyen la malformación de la proteína precursora amiloide (APP) , que desempeña un papel clave en el desencadenamiento de la enfermedad de Alzheimer. [92]Por el contrario, los niveles más bajos de ubiquilin-1 en el cerebro se han asociado con un aumento de la malformación de la APP . [92] Una mutación por desplazamiento del marco de lectura en la ubiquitina B puede resultar en un péptido truncado sin la glicina C-terminal . Se ha demostrado que este péptido anormal, conocido como UBB + 1 , se acumula selectivamente en la enfermedad de Alzheimer y otras tauopatías .
Infección e inmunidad
La ubiquitina y las moléculas similares a la ubiquitina regulan ampliamente las vías de transducción de señales inmunitarias en prácticamente todas las etapas, incluida la represión en estado estable, la activación durante la infección y la atenuación tras la eliminación. Sin esta regulación, la activación inmune contra patógenos puede ser defectuosa, lo que resulta en una enfermedad crónica o la muerte. Alternativamente, el sistema inmunológico puede volverse hiperactivo y los órganos y tejidos pueden estar sujetos a daño autoinmune .
Por otro lado, los virus deben bloquear o redirigir los procesos de la célula hospedadora, incluida la inmunidad, para replicarse de manera efectiva, sin embargo, muchos virus relevantes para la enfermedad tienen genomas limitados en cuanto a información . Debido a su gran número de funciones en la célula, la manipulación del sistema de ubiquitina representa una forma eficaz de que dichos virus bloqueen, subviertan o redirijan procesos críticos de la célula huésped para respaldar su propia replicación. [93]
La proteína del gen I inducible por ácido retinoico ( RIG-I ) es un sensor primario del sistema inmunológico para el ARN viral y otros ARN invasivos en células humanas. [94] La vía de señalización inmunitaria del receptor tipo RIG-I ( RLR ) es una de las más estudiadas en términos de la función de la ubiquitina en la regulación inmunitaria. [95]
Trastornos genéticos
- El síndrome de Angelman es causado por una interrupción de UBE3A , que codifica una enzima ubiquitina ligasa (E3) denominada E6-AP.
- El síndrome de Von Hippel-Lindau implica la interrupción de una ligasa de ubiquitina E3 denominada supresor de tumores VHL o gen VHL .
- Anemia de Fanconi : ocho de los trece genes identificados cuya alteración puede causar esta enfermedad codifican proteínas que forman un gran complejo de ubiquitina ligasa (E3).
- El síndrome 3-M es un trastorno de retraso del crecimiento autosómico recesivo asociado con mutaciones de la ubiquitina ligasa Cullin7 E3. [96]
Uso diagnóstico
La inmunohistoquímica que utiliza anticuerpos contra la ubiquitina puede identificar acumulaciones anormales de esta proteína dentro de las células, lo que indica un proceso patológico. Estas acumulaciones de proteínas se conocen como cuerpos de inclusión (que es un término general para cualquier colección microscópicamente visible de material anormal en una célula). Ejemplos incluyen:
- Enredos neurofibrilares en la enfermedad de Alzheimer
- Cuerpo de Lewy en la enfermedad de Parkinson
- Pick cadáveres en la enfermedad de Pick
- Inclusiones en la enfermedad de las neuronas motoras y la enfermedad de Huntington
- Cuerpos de Mallory en la enfermedad hepática alcohólica
- Fibras de Rosenthal en astrocitos
Enlace al cáncer
La modificación postraduccional de proteínas es un mecanismo generalmente utilizado en la señalización de células eucariotas . [97] La ubiquitinación, o conjugación de ubiquitina a proteínas , es un proceso crucial para la progresión del ciclo celular y la proliferación y desarrollo celular . Aunque la ubiquitinación generalmente sirve como una señal para la degradación de proteínas a través del proteasoma 26S , también podría servir para otros procesos celulares fundamentales, [97] por ejemplo, en endocitosis , [98] activación enzimática [99] y reparación del ADN. [100]Además, dado que la ubiquitinación funciona para regular estrechamente el nivel celular de ciclinas , se espera que su mala regulación tenga impactos severos. Se observó la primera evidencia de la importancia de la ruta ubiquitina / proteasoma en los procesos oncogénicos debido a la alta actividad antitumoral de los inhibidores del proteasoma. [101] [102] [103] Varios estudios han demostrado que los defectos o alteraciones en los procesos de ubiquitinación están comúnmente asociados o presentes en el carcinoma humano. [104] [105] [106] [107] [108] [109] [110] [111] Las neoplasias malignas se pueden desarrollar a través de la mutación de pérdida de función directamente en elgen supresor de tumores , aumento de la actividad de la ubiquitinación y / o atenuación indirecta de la ubiquitinación debido a la mutación en proteínas relacionadas. [112]
Pérdida directa de mutación de función de la ubiquitina ligasa E3
Carcinoma de células renales
El gen VHL ( Von Hippel-Lindau ) codifica un componente de una ubiquitina ligasa E3 . El complejo VHL se dirige a un miembro de la familia de factores de transcripción inducible por hipoxia (HIF) para su degradación al interactuar con el dominio de destrucción dependiente de oxígeno en condiciones normóxicas. HIF activa objetivos posteriores como el factor de crecimiento endotelial vascular (VEGF), promoviendo la angiogénesis . Las mutaciones en VHL previenen la degradación de HIF y, por tanto, conducen a la formación de lesiones hipervasculares y tumores renales. [104] [112]
Cáncer de mama
El gen BRCA1 es otro gen supresor de tumores en humanos que codifica la proteína BRCA1 que participa en la respuesta al daño del ADN. La proteína contiene un motivo RING con actividad de ubiquitina ligasa E3. BRCA1 podría formar dímeros con otras moléculas, como BARD1 y BAP1 , por su actividad de ubiquitinación. Las mutaciones que afectan la función de la ligasa a menudo se encuentran y se asocian con varios cánceres. [108] [112]
Ciclina E
Como los procesos en la progresión del ciclo celular son los procesos más fundamentales para el crecimiento y la diferenciación celular, y son los más comunes que se alteran en los carcinomas humanos, se espera que las proteínas reguladoras del ciclo celular estén bajo estricta regulación. El nivel de ciclinas, como su nombre indica, es alto solo en cierto momento durante el ciclo celular. Esto se logra mediante el control continuo de los niveles de ciclinas / CDK mediante ubiquitinación y degradación. Cuando la ciclina E se asocia con CDK2 y se fosforila, una proteína Fbw7 de caja F asociada a SCF reconoce el complejo y, por lo tanto, lo dirige para su degradación. Se han encontrado mutaciones en Fbw7 en más del 30% de los tumores humanos, caracterizándola como una proteína supresora de tumores. [111]
Aumento de la actividad de ubiquitinación
Cáncer de cuello uterino
Se sabe que los tipos oncogénicos del virus del papiloma humano (VPH) secuestran la vía celular ubiquitina- proteasoma para la infección viral y la replicación. Las proteínas E6 del VPH se unirán al extremo N de la ubiquitina ligasa celular E6-AP E3, redirigiendo el complejo para unirse a p53 , un gen supresor de tumores bien conocido cuya inactivación se encuentra en muchos tipos de cáncer. [106] Por lo tanto, p53 sufre ubiquitinación y degradación mediada por proteasoma. Mientras tanto, E7, otro de los genes del VPH de expresión temprana, se unirá a Rb , también un gen supresor de tumores, que mediará su degradación. [112] La pérdida de p53 y Rb en las células permite que se produzca una proliferación celular ilimitada.
Reglamento p53
La amplificación de genes a menudo ocurre en varios casos de tumores, incluido MDM2 , un gen que codifica una ubiquitina ligasa RING E3 responsable de la regulación a la baja de la actividad de p53. MDM2 se dirige a p53 para ubiquitinación y degradación proteasomal, manteniendo así su nivel apropiado para la condición celular normal. La sobreexpresión de MDM2 provoca la pérdida de la actividad de p53 y, por lo tanto, permite que las células tengan un potencial replicativo ilimitado. [107] [112]
p27
Otro gen que es un objetivo de la amplificación de genes es SKP2 . SKP2 es una proteína de caja F que participa en el reconocimiento de sustratos para la ubiquitinación y degradación. SKP2 se dirige a p27 Kip-1 , un inhibidor de las quinasas dependientes de ciclina ( CDK ). Las CDKs2 / 4 se asocian con las ciclinas E / D, respectivamente, familia de reguladores del ciclo celular para controlar la progresión del ciclo celular a través de la fase G1. El bajo nivel de proteína p27 Kip-1 se encuentra a menudo en varios cánceres y se debe a la sobreactivación de la proteólisis mediada por ubiquitina a través de la sobreexpresión de SKP2. [109] [112]
Efp
Efp , o proteína RING-finger inducible por estrógenos, es una ligasa de ubiquitina E3 cuya sobreexpresión ha demostrado ser la principal causa de cáncer de mama independiente de estrógenos . [103] [113] El sustrato de Efp es la proteína 14-3-3 que regula negativamente el ciclo celular.
Evasión de ubiquitinación
Cáncer colorrectal
El gen asociado con el cáncer colorrectal es la poliposis coli adenomatosa (APC), que es un gen supresor de tumores clásico . El producto del gen APC se dirige a la beta-catenina para su degradación a través de la ubiquitinación en el extremo N , regulando así su nivel celular. La mayoría de los casos de cáncer colorrectal se encuentran con mutaciones en el gen APC. Sin embargo, en los casos en los que el gen APC no está mutado, se encuentran mutaciones en el extremo N de la beta-catenina, lo que la hace libre de ubiquitinación y, por lo tanto, aumenta su actividad. [105] [112]
Glioblastoma
Como el cáncer más agresivo se originó en el cerebro, las mutaciones encontradas en pacientes con glioblastoma están relacionadas con la deleción de una parte del dominio extracelular del receptor del factor de crecimiento epidérmico (EGFR). Esta deleción provoca que la ligasa CBL E3 sea incapaz de unirse al receptor para su reciclaje y degradación a través de una vía ubiquitina-lisosomal. Por tanto, el EGFR es constitutivamente activo en la membrana celular y activa sus efectores posteriores que están implicados en la proliferación y migración celular. [110]
Ubiquitinación dependiente de la fosforilación
La interacción entre la ubiquitinación y la fosforilación ha sido un interés de investigación en curso, ya que la fosforilación a menudo sirve como un marcador donde la ubiquitinación conduce a la degradación. [97] Además, la ubiquitinación también puede actuar para activar o desactivar la actividad quinasa de una proteína. [114] El papel fundamental de la fosforilación se subraya en gran medida en la activación y eliminación de la autoinhibición en la proteína Cbl . [115] Cbl es una ubiquitina ligasa E3 con un dominio de dedo RING que interactúa con su dominio de unión a tirosina quinasa (TKB) , lo que evita la interacción del dominio RING con una enzima conjugadora de ubiquitina E2. Esta interacción intramolecular es una regulación de autoinhibición que evita su papel como regulador negativo de varios factores de crecimiento y de señalización de tirosina quinasa y activación de células T. [115] La fosforilación de Y363 alivia la autoinhibición y mejora la unión a E2. [115] Se ha demostrado que las mutaciones que hacen que la proteína Cbl sea disfuncional debido a la pérdida de su función ligasa / supresora de tumores y el mantenimiento de su función oncogénica / de señalización positiva provocan el desarrollo de cáncer. [116] [117]
Como objetivo de drogas
Detección de sustratos de ubiquitina ligasa
La identificación de los sustratos de la ligasa E3 es fundamental para comprender su implicación en las enfermedades humanas, ya que la desregulación de las interacciones entre el sustrato E3 y E3 suele ser una causa principal en muchos casos. Para superar la limitación del mecanismo utilizado para identificar los sustratos de la ubiquitina ligasa E3, en 2008 se desarrolló un sistema llamado 'Perfil de estabilidad global de proteínas (GPS)'. [118] Este sistema de alto rendimiento utilizó proteínas informadoras fusionadas con miles de sustratos potenciales de forma independiente. Mediante la inhibición de la actividad de la ligasa (a través de la fabricación de Cul1 dominante negativo, por lo tanto, hace que no se produzca la ubiquitinación), el aumento de la actividad informadora muestra que los sustratos identificados se están acumulando. Este enfoque agregó una gran cantidad de sustratos nuevos a la lista de sustratos de ligasa E3.
Posibles aplicaciones terapéuticas
Bloqueo del reconocimiento de sustrato específico por las ligasas E3, por ejemplo, Bortezomib . [113]
Desafío
Encontrar una molécula específica que inhiba selectivamente la actividad de una determinada ligasa E3 y / o las interacciones proteína-proteína implicadas en la enfermedad sigue siendo una de las áreas de investigación importantes y en expansión. Además, como la ubiquitinación es un proceso de varios pasos con varios actores y formas intermedias, es necesario tener muy en cuenta las interacciones muy complejas entre los componentes al diseñar los inhibidores de moléculas pequeñas. [103]
Proteínas similares
Aunque la ubiquitina es el modificador postraducción más comprendido, existe una familia creciente de proteínas similares a la ubiquitina (UBL) que modifican las dianas celulares en una vía paralela a la de la ubiquitina, pero distinta. Los UBL conocidos incluyen: pequeño modificador similar a ubiquitina ( SUMO ), proteína de reacción cruzada de ubiquitina (UCRP, también conocida como gen 15 estimulado por interferón ISG15 ), modificador 1 relacionado con ubiquitina ( URM1 ), expresado en células precursoras neuronales proteína-8 regulada negativamente en el desarrollo ( NEDD8 , también llamada Rub1 en S. cerevisiae ), antígeno leucocitario humano F asociado ( FAT10 ), autofagia-8 ( ATG8 ) y -12 ( ATG12), Pocas proteínas similares a la ubiquitina ( FUB1 ), MUB (UBL anclado a la membrana), [119] modificador-1 del pliegue de ubiquitina ( UFM1 ) y proteína similar a la ubiquitina-5 ( UBL5 , que se conoce como homóloga a la ubiquitina). 1 [Hub1] en S. pombe ). [120] [121] Si bien estas proteínas comparten solo una identidad de secuencia primaria modesta con la ubiquitina, están estrechamente relacionadas en tres dimensiones. Por ejemplo, SUMO comparte solo el 18% de identidad de secuencia, pero contienen el mismo pliegue estructural. Este pliegue se denomina "pliegue de ubiquitina". FAT10 y UCRP contienen dos. Este pliegue de agarre beta globular compacto se encuentra en ubiquitina, UBL y proteínas que comprenden un dominio similar a la ubiquitina, por ejemplo, S. cerevisiae La proteína de duplicación del cuerpo del polo del huso, Dsk2, y la proteína NER, Rad23, contienen dominios de ubiquitina N-terminales.
Estas moléculas relacionadas tienen funciones novedosas e influyen en diversos procesos biológicos. También existe una regulación cruzada entre las diversas vías de conjugación, ya que algunas proteínas pueden ser modificadas por más de un UBL y, a veces, incluso en el mismo residuo de lisina. Por ejemplo, la modificación SUMO a menudo actúa de manera antagónica a la de la ubiquitinación y sirve para estabilizar sustratos de proteínas. Las proteínas conjugadas con UBL no suelen ser objeto de degradación por parte del proteasoma, sino que funcionan en diversas actividades reguladoras. La unión de UBL podría alterar la conformación del sustrato, afectar la afinidad por los ligandos u otras moléculas que interactúan, alterar la localización del sustrato e influir en la estabilidad de la proteína.
Las UBL son estructuralmente similares a la ubiquitina y se procesan, activan, conjugan y liberan de los conjugados mediante pasos enzimáticos que son similares a los mecanismos correspondientes para la ubiquitina. Los UBL también se traducen con extensiones C-terminal que se procesan para exponer el invariante C-terminal LRGG. Estos modificadores tienen sus propias enzimas específicas E1 (activación), E2 (conjugación) y E3 (ligadura) que conjugan las UBL con dianas intracelulares. Estos conjugados pueden revertirse mediante isopeptidasas específicas de UBL que tienen mecanismos similares a los de las enzimas desubiquitinantes. [73]
Dentro de algunas especies, el reconocimiento y destrucción de las mitocondrias de los espermatozoides a través de un mecanismo que involucra a la ubiquitina es responsable de la eliminación de las mitocondrias de los espermatozoides después de que ocurre la fertilización. [122]
Orígenes procariotas
Se cree que la ubiquitina desciende de proteínas bacterianas similares a ThiS ( O32583 ) [123] o MoaD ( P30748 ). [124] Estas proteínas procarióticas, a pesar de tener poca identidad de secuencia (ThiS tiene un 14% de identidad con la ubiquitina), comparten el mismo pliegue proteico. Estas proteínas también comparten la química del azufre con la ubiquitina. MoaD, que participa en la biosíntesis de molibdopterina , interactúa con MoeB, que actúa como una enzima activadora de ubiquitina E1 para MoaD, fortaleciendo el vínculo entre estas proteínas procarióticas y el sistema de ubiquitina. Existe un sistema similar para ThiS, con su enzima ThiF similar a E1. También se cree que la proteína de Saccharomyces cerevisiae Urm-1, un modificador relacionado con la ubiquitina, es un "fósil molecular" que conecta la relación evolutiva con las moléculas procarióticas similares a la ubiquitina y la ubiquitina. [125]
Las arqueas tienen un homólogo funcionalmente más cercano del sistema de modificación de ubiquitina, en el que se realiza la "muestreo" con SAMP (pequeñas proteínas modificadoras de arqueas). El sistema de muestreo solo usa E1 para guiar proteínas al proteosoma . [126] Proteoarchaeota , que están relacionados con el antepasado de los eucariotas, poseen todas las enzimas E1, E2 y E3 más un sistema Rpn11 regulado. A diferencia de SAMP, que son más similares a ThiS o MoaD, la ubiquitina de Proteoarchaeota es más similar a los homólogos eucariotas. [127]
Proteína procariota similar a la ubiquitina (Pup) y ubiquitina bacteriana (UBact)
La proteína procariótica similar a la ubiquitina (Pup) es un análogo funcional de la ubiquitina que se ha encontrado en el filo bacteriano Gram-positivo Actinobacteria . Tiene la misma función (dirigirse a proteínas para degradaciones), aunque la enzimología de ubiquitinación y pupilación es diferente, y las dos familias no comparten homología. En contraste con la reacción de ubiquitinación de tres pasos, la pupilación requiere dos pasos, por lo tanto, solo dos enzimas están involucradas en la pupilación.
En 2017, se notificaron homólogos de Pup en cinco filos de bacterias gramnegativas , en siete filos de bacterias candidatas y en un arqueón [128] Las secuencias de los homólogos de Pup son muy diferentes de las secuencias de Pup en bacterias grampositivas y fueron denominada ubiquitina bacteriana (UBact), aunque aún no se ha demostrado que la distinción esté respaldada filogenéticamente por un origen evolutivo separado y no tiene evidencia experimental. [128]
El hallazgo del sistema Pup / UBact-proteasoma en bacterias grampositivas y gramnegativas sugiere que el sistema Pup / UBact-proteasoma evolucionó en bacterias antes de la división en clados gram positivos y negativos hace más de 3000 millones de años o, [129] que estos sistemas fueron adquiridos por diferentes linajes bacterianos a través de transferencia horizontal de genes de un tercer organismo, aún desconocido. En apoyo de la segunda posibilidad, se encontraron dos loci UBact en el genoma de un Archaeon metanotrófico anaeróbico sin cultivar (ANME-1; locus CBH38808.1 y locus CBH39258.1 ).
Proteínas humanas que contienen dominio de ubiquitina
Estos incluyen proteínas similares a la ubiquitina.
ANUBL1 ; BAG1 ; BAT3 / BAG6 ; C1orf131 ; DDI1 ; DDI2 ; FAU ; HERPUD1 ; HERPUD2 ; LÚPULO ; IKBKB ; ISG15 ; LOC391257 ; MIDN ; NEDD8 ; OASL ; PARK2 ; RAD23A ; RAD23B ; RPS27A ; SACS ; 8U SF3A1 ; SUMO1 ; SUMO2 ; SUMO3 ; SUMO4 ; TMUB1 ; TMUB2 ; UBA52 ; UBB ; UBC ; UBD ; UBFD1 ; UBL4 ; UBL4A ; UBL4B ; UBL7 ; UBLCP1 ; UBQLN1 ; UBQLN2 ; UBQLN3 ; UBQLN4 ; UBQLNL ; UBTD1 ; UBTD2 ; UHRF1 ; UHRF2;
Proteínas relacionadas
- Dominio proteico asociado a ubiquitina
Predicción de ubiquitinación
Los programas de predicción disponibles actualmente son:
- UbiPred es un servidor de predicción basado en SVM que utiliza 31 propiedades fisicoquímicas para predecir sitios de ubiquitinación. [130]
- UbPred es un predictor aleatorio basado en bosques de posibles sitios de ubiquitinación en proteínas. Se entrenó en un conjunto combinado de 266 sitios de ubiquitinación no redundantes verificados experimentalmente disponibles de nuestros experimentos y de dos estudios proteómicos a gran escala. [131]
- CKSAAP_UbSite es una predicción basada en SVM que emplea la composición de pares de aminoácidos espaciados k que rodean un sitio de consulta (es decir, cualquier lisina en una secuencia de consulta) como entrada, utiliza el mismo conjunto de datos que UbPred. [132]
Ver también
- Autofagia
- Autophagin
- ERAD
- JUNQ y IPOD
- Proteína procariota similar a la ubiquitina
- Enzimas SUMO
Referencias
- ^ a b Goldstein G, Scheid M, Hammerling U, Schlesinger DH, Niall HD, Boyse EA (enero de 1975). "Aislamiento de un polipéptido que tiene propiedades diferenciadoras de linfocitos y probablemente esté representado universalmente en células vivas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 72 (1): 11–5. Código Bibliográfico : 1975PNAS ... 72 ... 11G . doi : 10.1073 / pnas.72.1.11 . PMC 432229 . PMID 1078892 .
- ^ Wilkinson KD (octubre de 2005). "El descubrimiento de la proteólisis dependiente de ubiquitina" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (43): 15280–2. Código de Bibliografía : 2005PNAS..10215280W . doi : 10.1073 / pnas.0504842102 . PMC 1266097 . PMID 16230621 .
- ↑ a b Kimura Y, Tanaka K (junio de 2010). "Mecanismos reguladores implicados en el control de la homeostasis de ubiquitina" . Revista de bioquímica . 147 (6): 793–8. doi : 10.1093 / jb / mvq044 . PMID 20418328 .
- ↑ a b c Glickman MH, Ciechanover A (abril de 2002). "La vía proteolítica de ubiquitina-proteasoma: destrucción en aras de la construcción". Revisiones fisiológicas . 82 (2): 373–428. doi : 10.1152 / physrev.00027.2001 . PMID 11917093 .
- ↑ a b Mukhopadhyay D, Riezman H (enero de 2007). "Funciones independientes del proteasoma de ubiquitina en endocitosis y señalización". Ciencia . 315 (5809): 201–5. Código Bibliográfico : 2007Sci ... 315..201M . doi : 10.1126 / science.1127085 . PMID 17218518 . S2CID 35434448 .
- ↑ a b c Schnell JD, Hicke L (septiembre de 2003). "Funciones no tradicionales de ubiquitina y proteínas de unión a ubiquitina" . La revista de química biológica . 278 (38): 35857–60. doi : 10.1074 / jbc.R300018200 . PMID 12860974 .
- ↑ a b Pickart CM, Eddins MJ (noviembre de 2004). "Ubiquitina: estructuras, funciones, mecanismos" . Biochimica et Biophysica Acta (BBA) - Investigación de células moleculares . 1695 (1–3): 55–72. doi : 10.1016 / j.bbamcr.2004.09.019 . PMID 15571809 .
- ↑ a b c d e Komander D, Violación M (2012). "El código de ubiquitina". Revisión anual de bioquímica . 81 : 203-29. doi : 10.1146 / annurev-biochem-060310-170328 . PMID 22524316 .
- ↑ a b McDowell GS, Philpott A (agosto de 2013). "Ubiquitilación no canónica: mecanismos y consecuencias" . La Revista Internacional de Bioquímica y Biología Celular . 45 (8): 1833–42. doi : 10.1016 / j.biocel.2013.05.026 . PMID 23732108 .
- ↑ a b c d e Miranda M, Sorkin A (junio de 2007). "Regulación de receptores y transportadores por ubiquitinación: nuevos conocimientos sobre mecanismos sorprendentemente similares". Intervenciones moleculares . 7 (3): 157–67. doi : 10.1124 / mi.7.3.7 . PMID 17609522 .
- ^ a b "El Premio Nobel de Química 2004" . Nobelprize.org . Consultado el 16 de octubre de 2010 .
- ^ "El premio Nobel de química 2004: información popular" . Nobelprize.org . Consultado el 14 de diciembre de 2013 .
- ^ Ciechanover A, Hod Y, Hershko A (agosto de 2012). "Un componente polipeptídico termoestable de un sistema proteolítico dependiente de ATP de reticulocitos. 1978". Comunicaciones de investigación bioquímica y biofísica . 425 (3): 565–70. doi : 10.1016 / j.bbrc.2012.08.025 . PMID 22925675 .
- ↑ a b Pickart CM (2001). "Mecanismos subyacentes a la ubiquitilación". Revisión anual de bioquímica . 70 : 503–33. doi : 10.1146 / annurev.biochem.70.1.503 . PMID 11395416 .
- ^ Marotti LA, Newitt R, Wang Y, Aebersold R, Dohlman HG (abril de 2002). "Identificación directa de un sitio de ubiquitilación de proteína G por espectrometría de masas". Bioquímica . 41 (16): 5067–74. doi : 10.1021 / bi015940q . PMID 11955054 .
- ^ a b Peng J, Schwartz D, Elias JE, Thoreen CC, Cheng D, Marsischky G, Roelofs J, Finley D, Gygi SP (agosto de 2003). "Un enfoque proteómico para comprender la ubiquitilación de proteínas". Biotecnología de la naturaleza . 21 (8): 921–6. doi : 10.1038 / nbt849 . PMID 12872131 . S2CID 11992443 .
- ^ Breitschopf K, Bengala E, Ziv T, Admon A, Ciechanover A (octubre de 1998). "Un sitio novedoso para la ubiquitilación: el residuo N-terminal, y no las lisinas internas de MyoD, es esencial para la conjugación y degradación de la proteína" . El diario EMBO . 17 (20): 5964–73. doi : 10.1093 / emboj / 17.20.5964 . PMC 1170923 . PMID 9774340 .
- ^ Bloom J, Amador V, Bartolini F, DeMartino G, Pagano M (octubre de 2003). "Degradación mediada por proteasoma de p21 a través de ubiquitinilación N-terminal" . Celular . 115 (1): 71–82. doi : 10.1016 / S0092-8674 (03) 00755-4 . PMID 14532004 . S2CID 15114828 .
- ^ Scaglione KM, Basrur V, Ashraf NS, Konen JR, Elenitoba-Johnson KS, Todi SV, Paulson HL (junio de 2013). "La enzima conjugadora de ubiquitina (E2) Ube2w ubiquitina el extremo N de los sustratos" . La revista de química biológica . 288 (26): 18784–8. doi : 10.1074 / jbc.C113.477596 . PMC 3696654 . PMID 23696636 .
- ^ Sadeh R, Breitschopf K, Bercovich B, Zoabi M, Kravtsova-Ivantsiv Y, Kornitzer D, Schwartz A, Ciechanover A (octubre de 2008). "El dominio N-terminal de MyoD es necesario y suficiente para su degradación dependiente de la localización nuclear por el sistema de ubiquitina" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (41): 15690–5. Código Bibliográfico : 2008PNAS..10515690S . doi : 10.1073 / pnas.0808373105 . PMC 2560994 . PMID 18836078 .
- ^ Coulombe P, Rodier G, Bonneil E, Thibault P, Meloche S (julio de 2004). "La ubiquitilación N-terminal de la quinasa 3 regulada por señal extracelular y p21 dirige su degradación por el proteasoma" . Biología Molecular y Celular . 24 (14): 6140–50. doi : 10.1128 / MCB.24.14.6140-6150.2004 . PMC 434260 . PMID 15226418 .
- ^ Kuo ML, den Besten W, Bertwistle D, Roussel MF, Sherr CJ (agosto de 2004). "Polubiquitilación N-terminal y degradación del supresor de tumores Arf" . Genes y desarrollo . 18 (15): 1862–74. doi : 10.1101 / gad.1213904 . PMC 517406 . PMID 15289458 .
- ^ Ben-Saadon R, Fajerman I, Ziv T, Hellman U, Schwartz AL, Ciechanover A (octubre de 2004). "La proteína supresora de tumores p16 (INK4a) y la oncoproteína 58 E7 del virus del papiloma humano son proteínas sin lisina de origen natural que son degradadas por el sistema de ubiquitina. Evidencia directa de ubiquitilación en el residuo N-terminal" . La revista de química biológica . 279 (40): 41414–21. doi : 10.1074 / jbc.M407201200 . PMID 15254040 .
- ^ Li H, Okamoto K, Peart MJ, Prives C (febrero de 2009). "El recambio de ciclina G1 independiente de lisina puede estabilizarse mediante subunidades B'alpha de la proteína fosfatasa 2A" . Biología Molecular y Celular . 29 (3): 919-28. doi : 10.1128 / MCB.00907-08 . PMC 2630686 . PMID 18981217 .
- ^ Reinstein E, Scheffner M, Oren M, Ciechanover A, Schwartz A (noviembre de 2000). "Degradación de la oncoproteína del virus del papiloma humano E7 por el sistema ubiquitina-proteasoma: direccionamiento a través de ubiquitilación del residuo N-terminal" . Oncogén . 19 (51): 5944–50. doi : 10.1038 / sj.onc.1203989 . PMID 11127826 .
- ^ Aviel S, Winberg G, Massucci M, Ciechanover A (agosto de 2000). "Degradación de la proteína 1 de la membrana latente del virus epstein-barr (LMP1) por la vía de la ubiquitina-proteasoma. Orientación a través de la ubiquitilación del residuo N-terminal" . La revista de química biológica . 275 (31): 23491–9. doi : 10.1074 / jbc.M002052200 . PMID 10807912 .
- ^ Ikeda M, Ikeda A, Longnecker R (agosto de 2002). "Ubiquitilación independiente de lisina del virus de Epstein-Barr LMP2A" . Virología . 300 (1): 153–9. doi : 10.1006 / viro.2002.1562 . PMID 12202215 .
- ^ Yang J, Hong Y, Wang W, Wu W, Chi Y, Zong H, Kong X, Wei Y, Yun X, Cheng C, Chen K, Gu J (mayo de 2009). "HSP70 protege BCL2L12 y BCL2L12A de la degradación proteasomal mediada por ubiquitilación N-terminal" . Cartas FEBS . 583 (9): 1409-14. doi : 10.1016 / j.febslet.2009.04.011 . PMID 19376117 . S2CID 32330510 .
- ^ Wang Y, Shao Q, Yu X, Kong W, Hildreth JE, Liu B (mayo de 2011). "La etiqueta de hemaglutinina N-terminal hace que APOBEC3G deficiente en lisina sea resistente a la degradación inducida por Vif del VIH-1 por poliubiquitilación reducida" . Revista de Virología . 85 (9): 4510–9. doi : 10.1128 / JVI.01925-10 . PMC 3126286 . PMID 21345952 .
- ^ Trausch-Azar JS, Lingbeck J, Ciechanover A, Schwartz AL (julio de 2004). "La degradación de Id1 mediada por ubiquitina-proteasoma está modulada por MyoD" . La revista de química biológica . 279 (31): 32614–9. doi : 10.1074 / jbc.M403794200 . PMID 15163661 .
- ^ Trausch-Azar J, Leone TC, Kelly DP, Schwartz AL (diciembre de 2010). "Degradación dependiente del proteasoma de ubiquitina del coactivador transcripcional PGC-1 {alfa} a través de la vía N-terminal" . La revista de química biológica . 285 (51): 40192–200. doi : 10.1074 / jbc.M110.131615 . PMC 3001001 . PMID 20713359 .
- ^ Fajerman I, Schwartz AL, Ciechanover A (febrero de 2004). "Degradación del regulador de desarrollo Id2: orientación a través de ubiquitilación N-terminal". Comunicaciones de investigación bioquímica y biofísica . 314 (2): 505-12. doi : 10.1016 / j.bbrc.2003.12.116 . PMID 14733935 .
- ^ a b c Vosper JM, McDowell GS, Hindley CJ, Fiore-Heriche CS, Kucerova R, Horan I, Philpott A (junio de 2009). "La ubiquitilación en sitios canónicos y no canónicos se dirige al factor de transcripción neurogenina para la proteólisis mediada por ubiquitina" . La revista de química biológica . 284 (23): 15458–68. doi : 10.1074 / jbc.M809366200 . PMC 2708843 . PMID 19336407 .
- ^ a b c McDowell GS, Kucerova R, Philpott A (octubre de 2010). "La ubiquitilación no canónica de la proteína proneural Ngn2 ocurre tanto en embriones de Xenopus como en células de mamíferos". Comunicaciones de investigación bioquímica y biofísica . 400 (4): 655–60. doi : 10.1016 / j.bbrc.2010.08.122 . PMID 20807509 .
- ^ Tatham MH, Plechanovová A, Jaffray EG, Salmen H, Hay RT (julio de 2013). "Ube2W conjuga ubiquitina a grupos α-amino de la proteína N-terminal" . La revista bioquímica . 453 (1): 137–45. doi : 10.1042 / BJ20130244 . PMC 3778709 . PMID 23560854 .
- ^ Vittal V, Shi L, Wenzel DM, Scaglione KM, Duncan ED, Basrur V, Elenitoba-Johnson KS, Baker D, Paulson HL, Brzovic PS, Klevit RE (enero de 2015). "El trastorno intrínseco impulsa la ubiquitilación N-terminal por Ube2w" . Biología química de la naturaleza . 11 (1): 83–9. doi : 10.1038 / nchembio.1700 . PMC 4270946 . PMID 25436519 .
- ^ Johnson ES, Ma PC, Ota IM, Varshavsky A (julio de 1995). "Una vía proteolítica que reconoce la ubiquitina como una señal de degradación" . La revista de química biológica . 270 (29): 17442–56. doi : 10.1074 / jbc.270.29.17442 . PMID 7615550 .
- ↑ a b Kirisako T, Kamei K, Murata S, Kato M, Fukumoto H, Kanie M, Sano S, Tokunaga F, Tanaka K, Iwai K (octubre de 2006). "Un complejo de ubiquitina ligasa ensambla cadenas lineales de poliubiquitina" . El diario EMBO . 25 (20): 4877–87. doi : 10.1038 / sj.emboj.7601360 . PMC 1618115 . PMID 17006537 .
- ^ a b Wang X, Herr RA, Chua WJ, Lybarger L, Wiertz EJ, Hansen TH (mayo de 2007). "La ubiquitinación de residuos de serina, treonina o lisina en la cola citoplasmática puede inducir ERAD de MHC-I por la ligasa viral E3 mK3" . The Journal of Cell Biology . 177 (4): 613–24. doi : 10.1083 / jcb.200611063 . PMC 2064207 . PMID 17502423 .
- ^ Cadwell K, Coscoy L (julio de 2005). "Ubiquitinación en residuos de nonlysine por una ligasa de ubiquitina E3 viral" . Ciencia . 309 (5731): 127–30. Código bibliográfico : 2005Sci ... 309..127C . doi : 10.1126 / science.1110340 . PMID 15994556 .
- ^ Cadwell K, Coscoy L (abril de 2008). "Las especificidades de las ligasas de ubiquitina E3 codificadas por herpesvirus asociado al sarcoma de Kaposi están determinadas por las posiciones de los residuos de lisina o cisteína dentro de los dominios intracitoplasmáticos de sus objetivos" . Revista de Virología . 82 (8): 4184–9. doi : 10.1128 / JVI.02264-07 . PMC 2293015 . PMID 18272573 .
- ^ Williams C, van den Berg M, Sprenger RR, Distel B (agosto de 2007). "Una cisteína conservada es esencial para la ubiquitinación dependiente de Pex4p del receptor de importación peroxisomal Pex5p" . La revista de química biológica . 282 (31): 22534–43. doi : 10.1074 / jbc.M702038200 . PMID 17550898 .
- ^ Carvalho AF, Pinto MP, Grou CP, Alencastre IS, Fransen M, Sá-Miranda C, Azevedo JE (octubre de 2007). "Ubiquitinación de mamíferos Pex5p, el receptor de importación peroxisomal" . La revista de química biológica . 282 (43): 31267–72. doi : 10.1074 / jbc.M706325200 . PMID 17726030 .
- ^ Léon S, Subramani S (marzo de 2007). "Un residuo de cisteína conservado de Pichia pastoris Pex20p es esencial para su reciclaje del peroxisoma al citosol" . La revista de química biológica . 282 (10): 7424-30. doi : 10.1074 / jbc.M611627200 . PMC 3682499 . PMID 17209040 .
- ^ a b Tait SW, de Vries E, Maas C, Keller AM, D'Santos CS, Borst J (diciembre de 2007). "La inducción de la apoptosis por Bid requiere ubiquitinación no convencional y degradación de su fragmento N-terminal" . The Journal of Cell Biology . 179 (7): 1453–66. doi : 10.1083 / jcb.200707063 . PMC 2373500 . PMID 18166654 .
- ↑ a b Roark R, Itzhaki L, Philpott A (diciembre de 2012). "La regulación compleja controla la proteólisis de Neurogenin3" . Biología Abierta . 1 (12): 1264–72. doi : 10.1242 / bio.20121750 . PMC 3522888 . PMID 23259061 .
- ^ Magadán JG, Pérez-Victoria FJ, Sougrat R, Ye Y, Strebel K, Bonifacino JS (abril de 2010). "Mecanismo multicapa de regulación a la baja de CD4 por Vpu de VIH-1 que implica distintos pasos de retención de ER y dirección de ERAD" . PLOS Patógenos . 6 (4): e1000869. doi : 10.1371 / journal.ppat.1000869 . PMC 2861688 . PMID 20442859 .
- ^ Tokarev AA, Munguia J, Guatelli JC (enero de 2011). "La ubiquitinación de serina-treonina media la regulación a la baja de BST-2 / tetherin y el alivio de la liberación de virión restringida por Vpu de VIH-1" . Revista de Virología . 85 (1): 51–63. doi : 10.1128 / JVI.01795-10 . PMC 3014196 . PMID 20980512 .
- ^ Ishikura S, Weissman AM, Bonifacino JS (julio de 2010). "Los residuos de serina en la cola citosólica de la cadena alfa del receptor de antígeno de células T median la ubiquitinación y la degradación asociada al retículo endoplásmico de la proteína no ensamblada" . La revista de química biológica . 285 (31): 23916–24. doi : 10.1074 / jbc.M110.127936 . PMC 2911338 . PMID 20519503 .
- ^ Shimizu Y, Okuda-Shimizu Y, Hendershot LM (diciembre de 2010). "La ubiquitilación de un sustrato ERAD se produce en múltiples tipos de aminoácidos" . Célula molecular . 40 (6): 917–26. doi : 10.1016 / j.molcel.2010.11.033 . PMC 3031134 . PMID 21172657 .
- ^ Dikic I, Robertson M (marzo de 2012). "Ligasas de ubiquitina y más allá" . Biología BMC . 10 : 22. doi : 10.1186 / 1741-7007-10-22 . PMC 3305657 . PMID 22420755 .
- ^ Schulman BA, Harper JW (mayo de 2009). "Activación de proteínas similares a ubiquitina por enzimas E1: el ápice de las vías de señalización aguas abajo" . Nature Reviews Biología celular molecular . 10 (5): 319–31. doi : 10.1038 / nrm2673 . PMC 2712597 . PMID 19352404 .
- ^ Groettrup M, Pelzer C, Schmidtke G, Hofmann K (mayo de 2008). "Activando la familia de ubiquitina: UBA6 desafía el campo" . Tendencias en Ciencias Bioquímicas . 33 (5): 230–7. doi : 10.1016 / j.tibs.2008.01.005 . PMID 18353650 .
- ^ van Wijk SJ, Timmers HT (abril de 2010). "La familia de enzimas conjugadoras de ubiquitina (E2): decidir entre la vida y la muerte de las proteínas". Revista FASEB . 24 (4): 981–93. doi : 10.1096 / fj.09-136259 . PMID 19940261 . S2CID 21280193 .
- ^ Metzger MB, Hristova VA, Weissman AM (febrero de 2012). "Familias de dedos HECT y RING de ligasas de ubiquitina E3 de un vistazo" . Revista de ciencia celular . 125 (Pt 3): 531–7. doi : 10.1242 / jcs.091777 . PMC 3381717 . PMID 22389392 .
- ^ Skaar JR, Pagano M (diciembre de 2009). "Control del crecimiento celular por las ligasas de ubiquitina SCF y APC / C" . Opinión actual en biología celular . 21 (6): 816–24. doi : 10.1016 / j.ceb.2009.08.004 . PMC 2805079 . PMID 19775879 .
- ^ Kerscher O, Felberbaum R, Hochstrasser M (2006). "Modificación de proteínas por ubiquitina y proteínas similares a ubiquitina". Revisión anual de biología celular y del desarrollo . 22 : 159–80. doi : 10.1146 / annurev.cellbio.22.010605.093503 . PMID 16753028 .
- ^ Koegl M, Hoppe T, Schlenker S, Ulrich HD, Mayer TU, Jentsch S (marzo de 1999). "Un factor de ubiquitinación novedoso, E4, está involucrado en el ensamblaje de la cadena de multiubiquitina" . Celular . 96 (5): 635–44. doi : 10.1016 / S0092-8674 (00) 80574-7 . PMID 10089879 .
- ^ Lai Z, Ferry KV, Diamond MA, Wee KE, Kim YB, Ma J, Yang T, Benfield PA, Copeland RA, Auger KR (agosto de 2001). "El mdm2 humano media la mono-ubiquitinación múltiple de p53 por un mecanismo que requiere la isomerización de la enzima" . La revista de química biológica . 276 (33): 31357–67. doi : 10.1074 / jbc.M011517200 . PMID 11397792 .
- ^ Grossman SR, Deato ME, Brignone C, Chan HM, Kung AL, Tagami H, Nakatani Y, Livingston DM (abril de 2003). "Polubiquitinación de p53 por una actividad ligasa de ubiquitina de p300". Ciencia . 300 (5617): 342–4. Código Bibliográfico : 2003Sci ... 300..342G . doi : 10.1126 / science.1080386 . PMID 12690203 . S2CID 11526100 .
- ^ Shi D, Pop MS, Kulikov R, Love IM, Kung AL, Kung A, Grossman SR (septiembre de 2009). "CBP y p300 son ligasas de poliubiquitina E4 citoplasmáticas para p53" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 106 (38): 16275–80. Código bibliográfico : 2009PNAS..10616275S . doi : 10.1073 / pnas.0904305106 . PMC 2752525 . PMID 19805293 .
- ↑ a b c d e Komander D (octubre de 2009). "La complejidad emergente de la ubiquitinación de proteínas". Transacciones de la sociedad bioquímica . 37 (Pt 5): 937–53. doi : 10.1042 / BST0370937 . PMID 19754430 .
- ↑ a b c Ikeda F, Dikic I (junio de 2008). "Cadenas de ubiquitina atípicas: nuevas señales moleculares. Modificaciones de proteínas: más allá de la serie de revisiones de sospechosos habituales" . Informes EMBO . 9 (6): 536–42. doi : 10.1038 / embor.2008.93 . PMC 2427391 . PMID 18516089 .
- ^ Xu P, Peng J (mayo de 2008). "Caracterización de la estructura de la cadena de poliubiquitina por espectrometría de masas media-baja" . Química analítica . 80 (9): 3438–44. doi : 10.1021 / ac800016w . PMC 2663523 . PMID 18351785 .
- ↑ a b Hicke L (marzo de 2001). "Regulación de proteínas por monoubiquitin". Nature Reviews Biología celular molecular . 2 (3): 195–201. doi : 10.1038 / 35056583 . PMID 11265249 . S2CID 205013847 .
- ^ Lecker SH, Goldberg AL, Mitch WE (julio de 2006). "Degradación de proteínas por la vía ubiquitina-proteasoma en estados normales y patológicos" . Revista de la Sociedad Americana de Nefrología . 17 (7): 1807-19. doi : 10.1681 / ASN.2006010083 . PMID 16738015 .
- ↑ a b Kravtsova-Ivantsiv Y, Ciechanover A (febrero de 2012). "Señales basadas en ubiquitina no canónicas para la degradación proteasomal" . Revista de ciencia celular . 125 (Pt 3): 539–48. doi : 10.1242 / jcs.093567 . PMID 22389393 .
- ^ Nathan JA, Kim HT, Ting L, Gygi SP, Goldberg AL (febrero de 2013). "¿Por qué las proteínas celulares unidas a las cadenas de poliubiquitina K63 no se asocian con los proteasomas?" . El diario EMBO . 32 (4): 552–65. doi : 10.1038 / emboj.2012.354 . PMC 3579138 . PMID 23314748 .
- ^ Bache KG, Raiborg C, Mehlum A, Stenmark H (abril de 2003). "STAM y Hrs son subunidades de un complejo de unión a ubiquitina multivalente en endosomas tempranos" . La revista de química biológica . 278 (14): 12513–21. doi : 10.1074 / jbc.M210843200 . PMID 12551915 .
- ^ a b Zhao S, Ulrich HD (abril de 2010). "Distintas consecuencias de la modificación postraduccional por cadenas de poliubiquitina lineales frente a K63 enlazadas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 107 (17): 7704–9. Código Bibliográfico : 2010PNAS..107.7704Z . doi : 10.1073 / pnas.0908764107 . PMC 2867854 . PMID 20385835 .
- ^ Kim HT, Kim KP, Lledias F, Kisselev AF, Scaglione KM, Skowyra D, Gygi SP, Goldberg AL (junio de 2007). "Ciertos pares de enzimas conjugadoras de ubiquitina (E2) y ligasas de ubiquitina-proteína (E3) sintetizan cadenas de ubiquitina bifurcadas no degradables que contienen todos los enlaces isopéptidos posibles" . La revista de química biológica . 282 (24): 17375–86. doi : 10.1074 / jbc.M609659200 . PMID 17426036 .
- ^ Michel MA, Elliott PR, Swatek KN, Simicek M, Pruneda JN, Wagstaff JL, Freund SM, Komander D (abril de 2015). "Ensamblaje y reconocimiento específico de poliubiquitina unida a k29 y k33" . Célula molecular . 58 (1): 95–109. doi : 10.1016 / j.molcel.2015.01.042 . PMC 4386031 . PMID 25752577 .
- ^ a b "Descripción general de la vía del proteasoma de ubiquitina" . Archivado desde el original el 30 de marzo de 2008 . Consultado el 30 de abril de 2008 .
- ^ Bax, M (junio de 2019). "El sistema de proteasoma de ubiquitina es un regulador clave de la supervivencia de las células madre pluripotentes y la diferenciación de las neuronas motoras" . Celdas . 8 (6): 581. doi : 10.3390 / cells8060581 . PMC 6627164 . PMID 31200561 .
- ^ Soni D, Wang DM, Regmi SC, Mittal M, Vogel SM, Schlüter D, Tiruppathi C (mayo de 2018). "La función deubiquitinasa de A20 mantiene y repara la barrera endotelial después de una lesión vascular pulmonar" . Descubrimiento de la muerte celular . 4 (60): 60. doi : 10.1038 / s41420-018-0056-3 . PMC 5955943 . PMID 29796309 .
- ^ Thomas S, Hoxha K, Alexander W, Gilligan J, Dilbarova R, Whittaker K, Kossenkov A, Prendergast GC, Mullin JM. Apriete de la barrera intestinal por un anticuerpo que penetra en las células contra Bin1, un objetivo candidato para la inmunoterapia de la colitis ulcerosa. J Cell Biochem. 2019 Mar; 120 (3): 4225-4237.
- ^ Shaheen M, Shanmugam I, Hromas R (agosto de 2010). "El papel de las modificaciones postraduccionales de PCNA en la síntesis de translesiones" . Revista de ácidos nucleicos . 2010 : 1–8. doi : 10.4061 / 2010/761217 . PMC 2935186 . PMID 20847899 .
- ^ Jackson SP, Durocher D (marzo de 2013). "Regulación de las respuestas al daño del ADN por ubiquitina y SUMO" . Célula molecular . 49 (5): 795–807. doi : 10.1016 / j.molcel.2013.01.017 . PMID 23416108 .
- ^ Campbell SJ, Edwards RA, Leung CC, Neculai D, Hodge CD, Dhe-Paganon S, Glover JN (julio de 2012). "Conocimientos moleculares sobre la función de las proteínas que contienen RING finger (RNF) hRNF8 y hRNF168 en ubiquitilación dependiente de Ubc13 / Mms2" . La revista de química biológica . 287 (28): 23900–10. doi : 10.1074 / jbc.M112.359653 . PMC 3390666 . PMID 22589545 .
- ^ Ikura T, Tashiro S, Kakino A, Shima H, Jacob N, Amunugama R, Yoder K, Izumi S, Kuraoka I, Tanaka K, Kimura H, Ikura M, Nishikubo S, Ito T, Muto A, Miyagawa K, Takeda S, Fishel R, Igarashi K, Kamiya K (octubre de 2007). "La acetilación dependiente del daño del ADN y la ubiquitinación de H2AX mejora la dinámica de la cromatina" . Biología Molecular y Celular . 27 (20): 7028–40. doi : 10.1128 / MCB.00579-07 . PMC 2168918 . PMID 17709392 .
- ^ Kim H, Chen J, Yu X (mayo de 2007). "La proteína de unión a ubiquitina RAP80 media la respuesta al daño del ADN dependiente de BRCA1". Ciencia . 316 (5828): 1202–5. Código Bibliográfico : 2007Sci ... 316.1202K . doi : 10.1126 / science.1139621 . PMID 17525342 . S2CID 31636419 .
- ^ Hofmann K (abril de 2009). "Dominios de unión a ubiquitina y su papel en la respuesta al daño del ADN". Reparación de ADN . 8 (4): 544–56. doi : 10.1016 / j.dnarep.2009.01.003 . PMID 19213613 .
- ^ Hammond-Martel I, Yu H, Affar el B (febrero de 2012). "Funciones de la señalización de ubiquitina en la regulación de la transcripción". Señalización celular . 24 (2): 410-21. doi : 10.1016 / j.cellsig.2011.10.009 . PMID 22033037 .
- ^ Reyes-Turcu FE, Ventii KH, Wilkinson KD (2009). "Regulación y roles celulares de enzimas deubiquitinantes específicas de ubiquitina" . Revisión anual de bioquímica . 78 : 363–97. doi : 10.1146 / annurev.biochem.78.082307.091526 . PMC 2734102 . PMID 19489724 .
- ^ Nijman SM, Luna-Vargas MP, Velds A, Brummelkamp TR, Dirac AM, Sixma TK, Bernards R (diciembre de 2005). "Un inventario genómico y funcional de enzimas desubiquitinantes". Celular . 123 (5): 773–86. doi : 10.1016 / j.cell.2005.11.007 . hdl : 1874/20959 . PMID 16325574 . S2CID 15575576 .
- ↑ a b Hicke L, Schubert HL, Hill CP (agosto de 2005). "Dominios de unión a ubiquitina". Nature Reviews Biología celular molecular . 6 (8): 610-21. doi : 10.1038 / nrm1701 . PMID 16064137 . S2CID 3056635 .
- ↑ Husnjak K, Dikic I (1 de enero de 2012). "Proteínas de unión a ubiquitina: decodificadores de funciones celulares mediadas por ubiquitina". Revisión anual de bioquímica . 81 : 291–322. doi : 10.1146 / annurev-biochem-051810-094654 . PMID 22482907 .
- ^ Popovic, D (noviembre de 2014). "Ubiquitinación en la patogenia y el tratamiento de enfermedades" . Medicina de la naturaleza . 20 (11): 1242-1253. doi : 10.1038 / nm.3739 . PMID 25375928 . S2CID 205394130 .
- ^ Yerbury, Justin (mayo de 2020). "Disfunción de la homeostasis del proteoma: un principio unificador en la patogénesis de la ELA" . Tendencias en neurociencias . 43 (5): 274‐284. doi : 10.1016 / j.tins.2020.03.002 . PMID 32353332 . S2CID 216095994 .
- ^ Popovic, D (noviembre de 2014). "Ubiquitinación en la patogenia y el tratamiento de enfermedades" . Medicina de la naturaleza . 20 (11): 1242-1253. doi : 10.1038 / nm.3739 . PMID 25375928 . S2CID 205394130 .
- ^ "UBQLN1 ubiquilin 1 [Homo sapiens]" . Gene . Centro Nacional de Información Biotecnológica . Consultado el 9 de mayo de 2012 .
- ^ a b Stieren ES, El Ayadi A, Xiao Y, Siller E, Landsverk ML, Oberhauser AF, Barral JM, Boehning D (octubre de 2011). "Ubiquilin-1 es un acompañante molecular de la proteína precursora amiloide" . La revista de química biológica . 286 (41): 35689–98. doi : 10.1074 / jbc.M111.243147 . PMC 3195644 . PMID 21852239 . Resumen de Lay - Science Daily .
- ^ Heaton SM, Borg NA, Dixit VM (enero de 2016). "Ubiquitina en la activación y atenuación de la inmunidad antiviral innata" . La Revista de Medicina Experimental . 213 (1): 1–13. doi : 10.1084 / jem.20151531 . PMC 4710203 . PMID 26712804 .
- ^ Takeuchi O, Akira S (marzo de 2010). "Receptores de reconocimiento de patrones e inflamación" . Celular . 140 (6): 805-20. doi : 10.1016 / j.cell.2010.01.022 . PMID 20303872 . S2CID 223338 .
- ↑ Okamoto M, Kouwaki T, Fukushima Y, Oshiumi H (2018). "Regulación de la activación de RIG-I por poliubiquitinación ligada a K63" . Fronteras en inmunología . 8 : 1942. doi : 10.3389 / fimmu.2017.01942 . PMC 5760545 . PMID 29354136 .
- ^ Huber C, Dias-Santagata D, Glaser A, O'Sullivan J, Brauner R, Wu K, Xu X, Pearce K, Wang R, Uzielli ML, Dagoneau N, Chemaitilly W, Superti-Furga A, Dos Santos H, Mégarbané A, Morin G, Gillessen-Kaesbach G, Hennekam R, Van der Burgt I, Black GC, Clayton PE, Read A, Le Merrer M, Scambler PJ, Munnich A, Pan ZQ, Winter R, Cormier-Daire V (octubre 2005). "Identificación de mutaciones en CUL7 en el síndrome 3-M". Genética de la naturaleza . 37 (10): 1119–24. doi : 10.1038 / ng1628 . PMID 16142236 . S2CID 44003147 .
- ↑ a b c Nguyen LK, Kolch W, Kholodenko BN (julio de 2013). "Cuando la ubiquitinación se encuentra con la fosforilación: una perspectiva de biología de sistemas de señalización EGFR / MAPK" . Comunicación y señalización celular . 11 : 52. doi : 10.1186 / 1478-811X-11-52 . PMC 3734146 . PMID 23902637 .
- ^ Sorkin A, Goh LK (octubre de 2008). "Endocitosis y tráfico intracelular de ErbBs" . Investigación celular experimental . 314 (17): 3093–106. doi : 10.1016 / j.yexcr.2008.07.029 . PMC 2605728 . PMID 18793634 .
- ^ Nguyen LK, Muñoz-García J, Maccario H, Ciechanover A, Kolch W, Kholodenko BN (diciembre de 2011). "Interruptores, respuestas excitables y oscilaciones en el sistema de ubiquitinación Ring1B / Bmi1" . PLOS Biología Computacional . 7 (12): e1002317. Código bibliográfico : 2011PLSCB ... 7E2317N . doi : 10.1371 / journal.pcbi.1002317 . PMC 3240587 . PMID 22194680 .
- ^ Zhou W, Wang X, Rosenfeld MG (enero de 2009). "Ubiquitinación de histona H2A en la regulación transcripcional y reparación de daños en el ADN". La Revista Internacional de Bioquímica y Biología Celular . 41 (1): 12–5. doi : 10.1016 / j.biocel.2008.09.016 . PMID 18929679 .
- ^ Dou QP, Li B (agosto de 1999). "Inhibidores del proteasoma como posibles nuevos agentes anticancerígenos". Actualizaciones sobre resistencia a los medicamentos . 2 (4): 215-223. doi : 10.1054 / drup.1999.0095 . PMID 11504494 .
- ^ Vries EG, Verweij J (2000). "Investigación del cáncer clínico 2000: nuevos agentes y terapias". Actualizaciones sobre resistencia a los medicamentos . 3 (4): 197–201. doi : 10.1054 / drup.2000.0153 . PMID 11498385 .
- ^ a b c Pray TR, Parlati F, Huang J, Wong BR, Payan DG, Bennett MK, Issakani SD, Molineaux S, Demo SD (diciembre de 2002). "Ligasas de ubiquitina E3 reguladoras del ciclo celular como dianas contra el cáncer". Actualizaciones sobre resistencia a los medicamentos . 5 (6): 249–58. doi : 10.1016 / s1368-7646 (02) 00121-8 . PMID 12531181 .
- ^ a b Clifford SC, Cockman ME, Smallwood AC, Mole DR, Woodward ER, Maxwell PH, Ratcliffe PJ, Maher ER (2001). "Los efectos contrastantes sobre la regulación de HIF-1alpha por mutaciones de pVHL que causan enfermedades se correlacionan con patrones de tumorigénesis en la enfermedad de von Hippel-Lindau" . Genética molecular humana . 10 (10): 1029–38. doi : 10.1093 / hmg / 10.10.1029 . PMID 11331613 .
- ↑ a b Sparks AB, Morin PJ, Vogelstein B, Kinzler KW (marzo de 1998). "Análisis mutacional de la vía APC / beta-catenina / Tcf en cáncer colorrectal". Investigación del cáncer . 58 (6): 1130–4. PMID 9515795 .
- ↑ a b Scheffner M, Huibregtse JM, Vierstra RD, Howley PM (noviembre de 1993). "El complejo HPV-16 E6 y E6-AP funciona como una ubiquitina-proteína ligasa en la ubiquitinación de p53". Celular . 75 (3): 495–505. doi : 10.1016 / 0092-8674 (93) 90384-3 . PMID 8221889 . S2CID 27437768 .
- ↑ a b Momand J, Jung D, Wilczynski S, Niland J (agosto de 1998). "La base de datos de amplificación del gen MDM2" . Investigación de ácidos nucleicos . 26 (15): 3453–9. doi : 10.1093 / nar / 26.15.3453 . PMC 147746 . PMID 9671804 .
- ^ a b Hashizume R, Fukuda M, Maeda I, Nishikawa H, Oyake D, Yabuki Y, Ogata H, Ohta T (mayo de 2001). "El heterodímero RING BRCA1-BARD1 es una ubiquitina ligasa inactivada por una mutación derivada del cáncer de mama" . La revista de química biológica . 276 (18): 14537–40. doi : 10.1074 / jbc.C000881200 . PMID 11278247 .
- ↑ a b Zhu CQ, Blackhall FH, Pintilie M, Iyengar P, Liu N, Ho J, Chomiak T, Lau D, Winton T, Shepherd FA, Tsao MS (2004). "Las aberraciones del número de copias del gen Skp2 son comunes en el carcinoma de pulmón de células no pequeñas, y su sobreexpresión en tumores con mutación ras es un marcador de mal pronóstico" . Investigación clínica del cáncer . 10 (6): 1984-1991. doi : 10.1158 / 1078-0432.ccr-03-0470 . PMID 15041716 .
- ↑ a b Schmidt MH, Furnari FB, Cavenee WK, Bögler O (mayo de 2003). "La intensidad de señalización del receptor del factor de crecimiento epidérmico determina las interacciones de las proteínas intracelulares, la ubiquitinación y la internalización" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 100 (11): 6505–10. Código Bibliográfico : 2003PNAS..100.6505S . doi : 10.1073 / pnas.1031790100 . PMC 164476 . PMID 12734385 .
- ^ a b Knuutila S, Aalto Y, Autio K, Björkqvist AM, El-Rifai W, Hemmer S, Huhta T, Kettunen E, Kiuru-Kuhlefelt S, Larramendy ML, Lushnikova T, Monni O, Pere H, Tapper J, Tarkkanen M, Varis A, Wasenius VM, Wolf M, Zhu Y (septiembre de 1999). "Pérdidas de número de copias de ADN en neoplasias humanas" . La Revista Estadounidense de Patología . 155 (3): 683–94. doi : 10.1016 / S0002-9440 (10) 65166-8 . PMC 1866903 . PMID 10487825 .
- ↑ a b c d e f g Mani A, Gelmann EP (julio de 2005). "La vía de la ubiquitina-proteasoma y su papel en el cáncer". Revista de Oncología Clínica . 23 (21): 4776–89. doi : 10.1200 / JCO.2005.05.081 . PMID 16034054 .
- ↑ a b Nalepa G, Wade Harper J (mayo de 2003). "Dianas terapéuticas contra el cáncer aguas arriba del proteasoma". Reseñas de tratamientos contra el cáncer . 29 Supl. 1: 49–57. doi : 10.1016 / s0305-7372 (03) 00083-5 . PMID 12738243 .
- ^ Witowsky JA, Johnson GL (enero de 2003). "La ubiquitilación de MEKK1 inhibe su fosforilación de MKK1 y MKK4 y la activación de las vías ERK1 / 2 y JNK" . La revista de química biológica . 278 (3): 1403–6. doi : 10.1074 / jbc.C200616200 . PMID 12456688 .
- ^ a b c Kobashigawa Y, Tomitaka A, Kumeta H, Noda NN, Yamaguchi M, Inagaki F (diciembre de 2011). "Mecanismos de activación inducidos por fosforilación y autoinhibición del cáncer humano y la proteína E3 relacionada con enfermedades autoinmunes Cbl-b" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (51): 20579–84. Código bibliográfico : 2011PNAS..10820579K . doi : 10.1073 / pnas.1110712108 . PMC 3251137 . PMID 22158902 .
- ^ Niemeyer CM, Kang MW, Shin DH, Furlan I, Erlacher M, Bunin NJ, Bunda S, Finklestein JZ, Sakamoto KM, Gorr TA, Mehta P, Schmid I, Kropshofer G, Corbacioglu S, Lang PJ, Klein C, Schlegel PG, Heinzmann A, Schneider M, Starý J, van den Heuvel-Eibrink MM, Hasle H, Locatelli F, Sakai D, Archambeault S, Chen L, Russell RC, Sybingco SS, Ohh M, Braun BS, Flotho C, Loh ML (Septiembre de 2010). "Las mutaciones de CBL de la línea germinal causan anomalías en el desarrollo y predisponen a la leucemia mielomonocítica juvenil" . Genética de la naturaleza . 42 (9): 794–800. doi : 10.1038 / ng.641 . PMC 4297285 . PMID 20694012 .
- ^ Kales SC, Ryan PE, Nau MM, Lipkowitz S (junio de 2010). "Cbl y neoplasias mieloides humanas: el oncogén Cbl llega a la mayoría de edad" . Investigación del cáncer . 70 (12): 4789–94. doi : 10.1158 / 0008-5472.CAN-10-0610 . PMC 2888780 . PMID 20501843 .
- ^ Yen HC, Elledge SJ (2008). "Identificación de sustratos de ligasa de ubiquitina SCF por perfil de estabilidad de proteínas global". Ciencia . 322 (5903): 923–9. Código Bibliográfico : 2008Sci ... 322..923Y . doi : 10.1126 / science.1160462 . PMID 18988848 . S2CID 23586705 .
- ^ Downes BP, Saracco SA, Lee SS, Crowell DN, Vierstra RD (septiembre de 2006). "MUB, una familia de proteínas de pliegue de ubiquitina que están ancladas a la membrana plasmática por prenilación" . La revista de química biológica . 281 (37): 27145–57. doi : 10.1074 / jbc.M602283200 . PMID 16831869 .
- ^ Welchman RL, Gordon C, Mayer RJ (agosto de 2005). "Ubiquitina y proteínas similares a ubiquitina como señales multifuncionales". Nature Reviews Biología celular molecular . 6 (8): 599–609. doi : 10.1038 / nrm1700 . PMID 16064136 . S2CID 7373421 .
- ^ Grabbe C, Dikic I (abril de 2009). "Funciones funcionales de las proteínas que contienen dominio similar a ubiquitina (ULD) y dominio de unión a ubiquitina (UBD)". Revisiones químicas . 109 (4): 1481–94. doi : 10.1021 / cr800413p . PMID 19253967 .
- ^ Sutovsky P, Moreno RD, Ramalho-Santos J, Dominko T, Simerly C, Schatten G (agosto de 2000). "Mitocondrias de espermatozoides ubiquitinadas, proteólisis selectiva y regulación de la herencia mitocondrial en embriones de mamíferos" . Biología de la reproducción . 63 (2): 582–90. doi : 10.1095 / biolreprod63.2.582 . PMID 10906068 .
- ^ Wang C, Xi J, Begley TP, Nicholson LK (enero de 2001). "Estructura de la solución de ThiS e implicaciones para las raíces evolutivas de la ubiquitina". Biología estructural de la naturaleza . 8 (1): 47–51. doi : 10.1038 / 83041 . PMID 11135670 . S2CID 29632248 .
- ^ Lago MW, Wuebbens MM, Rajagopalan KV, Schindelin H (noviembre de 2001). "Mecanismo de activación de ubiquitina revelado por la estructura de un complejo bacteriano MoeB-MoaD". Naturaleza . 414 (6861): 325–9. Código Bibliográfico : 2001Natur.414..325L . doi : 10.1038 / 35104586 . PMID 11713534 . S2CID 3224437 .
- ^ Hochstrasser M (marzo de 2009). "Origen y función de proteínas similares a la ubiquitina" . Naturaleza . 458 (7237): 422–9. Código bibliográfico : 2009Natur.458..422H . doi : 10.1038 / nature07958 . PMC 2819001 . PMID 19325621 .
- ^ Maupin-Furlow JA (2013). "Proteasomas de Archaeal y sampylation" . Bioquímica subcelular . 66 : 297–327. doi : 10.1007 / 978-94-007-5940-4_11 . ISBN 978-94-007-5939-8. PMC 3936409 . PMID 23479445 .
- ^ Fuchs AC, Maldoner L, Wojtynek M, Hartmann MD, Martin J (julio de 2018). "Procesamiento de ubiquitina mediada por Rpn11 en un sistema de ubiquitinación de arquea ancestral" . Comunicaciones de la naturaleza . 9 (1): 2696. Bibcode : 2018NatCo ... 9.2696F . doi : 10.1038 / s41467-018-05198-1 . PMC 6043591 . PMID 30002364 .
- ↑ a b Lehmann G, Udasin RG, Livneh I, Ciechanover A (febrero de 2017). "Identificación de UBact, una proteína similar a la ubiquitina, junto con otros componentes homólogos de un sistema de conjugación y el proteasoma en diferentes bacterias gramnegativas". Comunicaciones de investigación bioquímica y biofísica . 483 (3): 946–950. doi : 10.1016 / j.bbrc.2017.01.037 . PMID 28087277 .
- ^ Marin J, Battistuzzi FU, Brown AC, Hedges SB (febrero de 2017). "El árbol del tiempo de los procariontes: nuevos conocimientos sobre su evolución y especiación" . Biología Molecular y Evolución . 34 (2): 437–446. doi : 10.1093 / molbev / msw245 . PMID 27965376 .
- ^ Tung CW, Ho SY (julio de 2008). "Identificación computacional de sitios de ubiquitilación de secuencias de proteínas" . BMC Bioinformática . 9 : 310. doi : 10.1186 / 1471-2105-9-310 . PMC 2488362 . PMID 18625080 .
- ^ Radivojac P, Vacic V, Haynes C, Cocklin RR, Mohan A, Heyen JW, Goebl MG, Iakoucheva LM (febrero de 2010). "Identificación, análisis y predicción de sitios de ubiquitinación de proteínas" . Las proteínas . 78 (2): 365–80. doi : 10.1002 / prot.22555 . PMC 3006176 . PMID 19722269 .
- ↑ Chen Z, Chen YZ, Wang XF, Wang C, Yan RX, Zhang Z (2011). Fraternali F (ed.). "Predicción de sitios de ubiquitinación mediante el uso de la composición de pares de aminoácidos espaciados k" . PLOS ONE . 6 (7): e22930. Código Bibliográfico : 2011PLoSO ... 622930C . doi : 10.1371 / journal.pone.0022930 . PMC 3146527 . PMID 21829559 .
Enlaces externos
- Entrada de GeneReviews / NCBI / NIH / UW sobre el síndrome de Angelman
- Entradas de OMIM sobre el síndrome de Angelman
- Entrada UniProt para ubiquitina
- "7.340 ubiquitinación: el proteasoma y la enfermedad humana" . MIT OpenCourseWare . 2004. Apuntes del curso MIT.
- Ubiquitina en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .
