Metabolismo de las purinas
Este artículo incluye una lista de referencias generales , pero permanece en gran parte sin verificar porque carece de suficientes citas en línea correspondientes . ( Julio de 2014 ) |
El metabolismo de las purinas se refiere a las vías metabólicas para sintetizar y descomponer las purinas que están presentes en muchos organismos.
Biosíntesis
Las purinas se sintetizan biológicamente como nucleótidos y, en particular, como ribótidos, es decir, bases unidas a la ribosa 5-fosfato . Tanto la adenina como la guanina se derivan del nucleótido inosina monofosfato (IMP), que es el primer compuesto de la ruta que tiene un sistema de anillo de purina completamente formado.
DIABLILLO
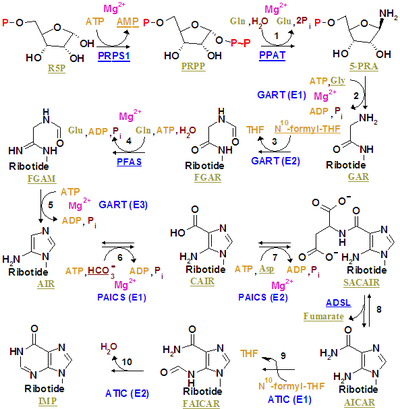
El monofosfato de inosina se sintetiza en una ribosa-fosfato preexistente a través de una vía compleja (como se muestra en la figura de la derecha). La fuente de los átomos de carbono y nitrógeno del anillo de purina, 5 y 4 respectivamente, proviene de múltiples fuentes. El aminoácido glicina aporta todos sus átomos de carbono (2) y nitrógeno (1), con átomos de nitrógeno adicionales de glutamina (2) y ácido aspártico (1), y átomos de carbono adicionales de grupos formilo (2), que se transfieren desde el tetrahidrofolato de coenzima como 10-formiltetrahidrofolato y un átomo de carbono de bicarbonato(1). Los grupos formilo forman carbono-2 y carbono-8 en el sistema de anillos de purina, que son los que actúan como puentes entre dos átomos de nitrógeno.
Un paso regulador clave es la producción de 5-fosfo-α- D- ribosil 1-pirofosfato ( PRPP ) por la ribosa fosfato pirofosfoquinasa, que es activada por fosfato inorgánico e inactivada por ribonucleótidos de purina. No es el paso comprometido para la síntesis de purina porque el PRPP también se usa en la síntesis de pirimidina y en las rutas de rescate.
El primer paso comprometido es la reacción de PRPP, glutamina y agua a 5'-fosforribosilamina (PRA), glutamato y pirofosfato , catalizada por amidofosforribosiltransferasa , que es activada por PRPP e inhibida por AMP , GMP e IMP .
PRPP + L-glutamina + H 2 O → PRA + L-glutamato + PPi
En el segundo paso, reaccione PRA , glicina y ATP para crear GAR , ADP y pirofosfato, catalizados por fosforribosilamina, glicina ligasa (GAR sintetasa). Debido a la labilidad química de PRA, que tiene una vida media de 38 segundos a pH 7,5 y 37 ° C, los investigadores han sugerido que el compuesto se canaliza de amidofosforribosiltransferasa a GAR sintetasa in vivo. [1]
PRA + Glicina + ATP → GAR + ADP + Pi
El tercero es catalizado por fosforribosilglicinamida formiltransferasa .
GAR + fTHF → fGAR + THF
El cuarto está catalizado por la fosforribosilformilglicinamidina sintasa .
fGAR + L-Glutamina + ATP → fGAM + L-Glutamato + ADP + Pi
El quinto es catalizado por AIR sintetasa (FGAM ciclasa) .
fGAM + ATP → AIRE + ADP + Pi + H 2 O
El sexto está catalizado por la fosforribosilaminoimidazol carboxilasa .
AIRE + CO 2 → CAIR + 2H +
El séptimo está catalizado por la fosforribosilaminoimidazoles succinocarboxamida sintasa .
CAIR + L-Aspartato + ATP → SAICAR + ADP + Pi
El ocho está catalizado por adenilosuccinato liasa .
SAICAR → AICAR + Fumarato
Los productos AICAR y fumarato avanzan por dos vías diferentes. AICAR sirve como reactivo para el noveno paso, mientras que el fumarato se transporta al ciclo del ácido cítrico, que luego puede omitir los pasos de evolución del dióxido de carbono para producir malato. La conversión de fumarato en malato es catalizada por fumarasa. De esta manera, el fumarato conecta la síntesis de purinas con el ciclo del ácido cítrico. [2]
El noveno está catalizado por la fosforribosilaminoimidazolcarboxamida formiltransferasa .
AICAR + fTHF → FAICAR + THF
El último paso es catalizado por la inosina monofosfato sintasa .
FAICAR → IMP + H 2 O
En eucariotas, el segundo, tercer y quinto paso son catalizados por la proteína biosintética de purina trifuncional adenosina-3 , que está codificada por el gen GART.
Tanto el noveno como el décimo paso se logran mediante una sola proteína llamada proteína biosíntesis de purina bifuncional PURH, codificada por el gen ATIC.
GMP
- IMP deshidrogenasa (IMPDH) convierte IMP en XMP
- GMP sintasa convierte XMP en GMP
- GMP reductasa convierte GMP de nuevo en IMP
AMPERIO
- la adenilosuccinato sintasa convierte IMP en adenilosuccinato
- La adenilosuccinato liasa convierte el adenilosuccinato en AMP
- AMP desaminasa convierte AMP nuevamente en IMP
Degradación
Las purinas son metabolizadas por varias enzimas :
Guanina
- Una nucleasa libera el nucleótido
- Una nucleotidasa crea guanosina
- La purina nucleósido fosforilasa convierte la guanosina en guanina
- La guanasa convierte la guanina en xantina
- La xantina oxidasa (una forma de xantina oxidorreductasa) cataliza la oxidación de la xantina a ácido úrico
Adenina
- Una nucleasa libera el nucleótido
- Una nucleotidasa crea adenosina , luego la adenosina desaminasa crea inosina
- Alternativamente, AMP desaminasa crea ácido inosínico , luego una nucleotidasa crea inosina
- La purina nucleósido fosforilasa actúa sobre la inosina para crear hipoxantina.
- La xantina oxidasa cataliza la biotransformación de hipoxantina en xantina
- La xantina oxidasa actúa sobre la xantina para crear ácido úrico
Regulaciones de la biosíntesis de nucleótidos de purina
La formación de 5'-fosforribosilamina a partir de glutamina y PRPP catalizada por PRPP aminotransferasa es el punto de regulación para la síntesis de purinas. La enzima es una enzima alostérica, por lo que se puede convertir de IMP, GMP y AMP en alta concentración. Se une a la enzima para ejercer inhibición, mientras que PRPP se une en gran cantidad a la enzima que causa la activación. Entonces, IMP, GMP y AMP son inhibidores, mientras que PRPP es un activador. Entre la formación de 5'-fosforribosilo, aminoimidazol e IMP, no se conoce ningún paso de regulación.
Salvar
Las purinas del recambio de ácidos nucleicos celulares (o de los alimentos) también se pueden recuperar y reutilizar en nuevos nucleótidos.
- La enzima adenina fosforribosiltransferasa (APRT) recupera la adenina .
- La enzima hipoxantina-guanina fosforribosiltransferasa (HGPRT) recupera la guanina y la hipoxantina . [3] (La deficiencia genética de HGPRT causa el síndrome de Lesch-Nyhan ).
Trastornos
Cuando un gen defectuoso hace que aparezcan brechas en el proceso de reciclaje metabólico de purinas y pirimidinas, estas sustancias químicas no se metabolizan correctamente y los adultos o los niños pueden sufrir cualquiera de los veintiocho trastornos hereditarios, posiblemente algunos más aún desconocidos. Los síntomas pueden incluir gota , anemia, epilepsia, retraso en el desarrollo, sordera, mordeduras compulsivas, insuficiencia renal o cálculos, o pérdida de inmunidad.
El metabolismo de las purinas puede tener desequilibrios que pueden surgir de la incorporación de nucleótidos trifosfatos nocivos en el ADN y el ARN, lo que conduce a alteraciones genéticas y mutaciones y, como resultado, dan lugar a varios tipos de enfermedades. Algunas de las enfermedades son:
- Inmunodeficiencia grave por pérdida de adenosina desaminasa.
- Hiperuricemia y síndrome de Lesch-Nyhan por pérdida de hipoxantina-guanina fosforribosiltransferasa.
- Diferentes tipos de cáncer por un aumento de la actividad de enzimas como la IMP deshidrogenasa. [4]
Farmacoterapia
La modulación del metabolismo de las purinas tiene valor farmacoterapéutico.
Los inhibidores de la síntesis de purinas inhiben la proliferación de células, especialmente leucocitos . Estos inhibidores incluyen azatioprina , un inmunosupresor utilizado en el trasplante de órganos , enfermedades autoinmunes como la artritis reumatoide o enfermedad inflamatoria intestinal como la enfermedad de Crohn y la colitis ulcerosa .
El micofenolato de mofetilo es un fármaco inmunosupresor que se utiliza para prevenir el rechazo en el trasplante de órganos; inhibe la síntesis de purinas bloqueando la inositol monofosfato deshidrogenasa. Además, el metotrexato inhibe indirectamente la síntesis de purinas al bloquear el metabolismo del ácido fólico (es un inhibidor de la dihidrofolato reductasa ).
El alopurinol es un fármaco que inhibe la enzima xantina oxidorreductasa y, por tanto, reduce el nivel de ácido úrico en el organismo. Esto puede ser útil en el tratamiento de la gota, que es una enfermedad causada por el exceso de ácido úrico, que forma cristales en las articulaciones.
Síntesis prebiótica de ribonucleósidos de purina
Para comprender cómo surgió la vida , se requiere conocimiento de las vías químicas que permiten la formación de los componentes básicos de la vida en condiciones prebióticas plausibles . Nam y col. [5] demostró la condensación directa de nucleobases de purina y pirimidina con ribosa para dar ribonucleósidos en microgotas acuosas, un paso clave que conduce a la formación de ARN. Además, Becker et al. Presentaron un proceso prebiótico plausible para sintetizar ribonucleósidos de purina. [6]
Biosíntesis de purinas en los tres dominios de la vida
Los organismos de los tres dominios de la vida, eucariotas , bacterias y arqueas , pueden realizar la biosíntesis de novo de purinas. Esta capacidad refleja la esencialidad de las purinas para la vida. La vía bioquímica de síntesis es muy similar en eucariotas y especies bacterianas, pero es más variable entre especies de arqueas. [7] Se determinó que un conjunto casi completo, o completo, de genes requeridos para la biosíntesis de purinas está presente en 58 de las 65 especies de arqueas estudiadas. [7] Sin embargo, también se identificaron siete especies de arqueas con genes que codifican purina total o casi completamente ausentes. Aparentemente, las especies de arqueas que no pueden sintetizar purinas pueden adquirir purinas exógenas para crecer. [7] , y por lo tanto son similares a los mutantes de purina de eucariotas, por ejemplo, mutantes de purina del hongo Ascomycete Neurospora crassa , [8] que también requieren purinas exógenas para su crecimiento.
Ver también
- Señalización purinérgica
- Fármaco antirreumático modificador de la enfermedad (FAME)
Referencias
- ^ Antle VD, Liu D, McKellar BR, Caperelli CA, Hua M, Vince R (abril de 1996). "Especificidad de sustrato de sintetasa de ribonucleótido de glicinamida de hígado de pollo" . La revista de química biológica . 271 (14): 8192–5. doi : 10.1074 / jbc.271.14.8192 . PMID 8626510 .
- ↑ Garrett RH, Grisham CM (11 de febrero de 2016). Bioquímica (Sexta ed.). Boston, MA. págs. 666 y 934. ISBN 9781305577206. OCLC 914290655 .
- ^ Ansari MY, Equbal A, Dikhit MR, Mansuri R, Rana S, Ali V, et al. (Febrero de 2016). "Establecimiento de correlación entre análisis de prueba in-silico e in-vitro contra Leishmania HGPRT a inhibidores". Revista Internacional de Macromoléculas Biológicas . 83 : 78–96. doi : 10.1016 / j.ijbiomac.2015.11.051 . PMID 26616453 .
- ^ Pang B, McFaline JL, Burgis NE, Dong M, Taghizadeh K, Sullivan MR, et al. (Febrero de 2012). "Los defectos en el metabolismo de los nucleótidos de purina conducen a una incorporación sustancial de xantina e hipoxantina en el ADN y el ARN" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (7): 2319–24. Código Bibliográfico : 2012PNAS..109.2319P . doi : 10.1073 / pnas.1118455109 . JSTOR 41477470 . PMC 3289290 . PMID 22308425 .
- ^ Nam I, Nam HG, Zare RN. Síntesis abiótica de ribonucleósidos de purina y pirimidina en microgotas acuosas. Proc Natl Acad Sci US A. 2 de enero de 2018; 115 (1): 36-40. doi: 10.1073 / pnas.1718559115. Publicación electrónica del 18 de diciembre de 2017 PMID: 29255025; PMCID: PMC5776833
- ^ Becker S, Thoma I, Deutsch A, Gehrke T, Mayer P, Zipse H, Carell T. Una vía de formación de nucleósidos de purina prebiótica estrictamente regioselectiva de alto rendimiento. Ciencia. 2016 13 de mayo; 352 (6287): 833-6. doi: 10.1126 / science.aad2808. PMID: 27174989.
- ^ a b c Marrón AM, Hoopes SL, Blanco RH, Sarisky CA. Biosíntesis de purinas en arqueas: variaciones sobre un tema. Biol Direct. 14 de diciembre de 2011; 6:63. doi: 10.1186 / 1745-6150-6-63. PMID: 22168471; PMCID: PMC3261824
- ^ Bernstein, H. compuestos de imidazol acumulados por mutantes de purina de Neurospora crassa J. Gen. Microbiol. 5: 41-46 (1961)
enlaces externos
- La página de bioquímica médica
- Metabolismo de las purinas - Vía de referencia
- PUMPA: Asociación de pacientes metabólicos de purina
- Purinas
- Vías metabólicas
- Bioquímica