Código de barras de ADN de algas
El código de barras de ADN de las algas se usa comúnmente para la identificación de especies y estudios filogenéticos . Las algas forman un grupo filogenéticamente heterogéneo, lo que significa que la aplicación de un único código de barras / marcador universal para la delimitación de especies es inviable, por lo que se aplican diferentes marcadores / códigos de barras para este fin en diferentes grupos de algas.
El código de barras de ADN de diatomeas es un método para la identificación taxonómica de diatomeas incluso a nivel de especie . Se lleva a cabo utilizando ADN o ARN seguido de amplificación y secuenciación de regiones conservadas específicas en el genoma de la diatomea seguida de asignación taxonómica.
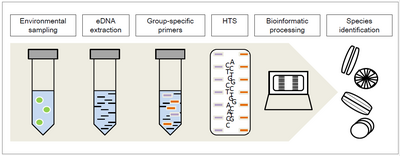
Uno de los principales desafíos de la identificación de diatomeas es que a menudo se recolecta como una mezcla de diatomeas de varias especies. La codificación de metabarras de ADN es el proceso de identificación de especies individuales a partir de una muestra mixta de ADN ambiental (también llamado eDNA) que es ADN extraído directamente del medio ambiente, como en muestras de suelo o agua.
Un método recientemente aplicado es la codificación de metabarras de ADN de diatomeas, que se utiliza para la evaluación de la calidad ecológica de ríos y arroyos debido a la respuesta específica de las diatomeas a condiciones ecológicas particulares. Como la identificación de especies a través de la morfología es relativamente difícil y requiere mucho tiempo y experiencia, [1] [2] la metabarcoding de ADN de secuenciación de alto rendimiento (HTS) permite la asignación taxonómica y, por lo tanto, la identificación de la muestra completa con respecto a los cebadores específicos del grupo elegidos para el amplificación previa del ADN .
Hasta ahora, ya se han desarrollado varios marcadores de ADN , dirigidos principalmente al ARNr 18S . [3] Utilizando la región hipervariable V4 del ADN de subunidad pequeña ribosomal (SSU rADN), se encontró que la identificación basada en ADN era más eficiente que el enfoque clásico basado en la morfología. [4] Otras regiones conservadas en los genomas que se utilizan con frecuencia como genes marcadores son la ribulosa-1-5-bisfosfato carboxilasa (rbcL), la citocromo oxidasa I (cox1, COI), [5] ITS [6] y 28S . [7]Se ha demostrado repetidamente que los datos moleculares obtenidos mediante la codificación de metabarras de eDNA de diatomeas reflejan con bastante fidelidad los índices de diatomeas bióticas basados en la morfología y, por lo tanto, proporcionan una evaluación similar del estado del ecosistema. [8] [9] Mientras tanto, las diatomeas se utilizan habitualmente para la evaluación de la calidad ecológica en otros ecosistemas de agua dulce. [7] Junto con los invertebrados acuáticos , se consideran los mejores indicadores de perturbaciones relacionadas con las condiciones físicas, químicas o biológicas de los cursos de agua. Numerosos estudios están utilizando diatomeas bentónicas para el biomonitoreo. [10] [11] [12] [13]Debido a que no se encontró un código de barras de ADN de diatomeas ideal, se ha propuesto que se usan diferentes marcadores para diferentes propósitos. De hecho, los genes cox1, ITS y 28S, altamente variables, se consideraron más adecuados para estudios taxonómicos, mientras que los genes 18S y rbcL más conservados parecen más apropiados para la biomonitorización.
La aplicación del concepto de códigos de barras de ADN a las diatomeas promete un gran potencial para resolver el problema de la identificación inexacta de especies y así facilitar los análisis de la biodiversidad de las muestras ambientales. [14]
.jpg/440px-Naviculoid_diatom_(3074442495).jpg)