La hibridación in situ (ISH) es un tipo de hibridación que utiliza unacadena de ADN , ARN o ácidos nucleicos modificados complementarios marcados(es decir, una sonda ) para localizar una secuencia de ADN o ARN específica en una porción o sección de tejido ( in situ ) o si el tejido es lo suficientemente pequeño (p. ej., semillas de plantas,embriones de Drosophila ), en todo el tejido (montaje completo de ISH), en las células y en las células tumorales circulantes (CTC). Esto es distinto de la inmunohistoquímica , que generalmente localiza proteínas en secciones de tejido.
La hibridación in situ se utiliza para revelar la ubicación de secuencias específicas de ácidos nucleicos en cromosomas o tejidos, un paso crucial para comprender la organización, regulación y función de los genes. Las técnicas clave actualmente en uso incluyen la hibridación in situ a ARNm con oligonucleótidos y sondas de ARN (tanto radiomarcadas como con hapteno), análisis con microscopios de luz y electrónicos, hibridación in situ de montaje completo , doble detección de ARN y ARN más proteína, e hibridación in situ fluorescente para detectar secuencias cromosómicas. El ADN ISH se puede utilizar para determinar la estructura de los cromosomas. ISH de ADN fluorescente(FISH) se puede utilizar, por ejemplo, en diagnósticos médicos para evaluar la integridad cromosómica. El ARN ISH ( hibridación in situ de ARN ) se utiliza para medir y localizar ARN (ARNm, ARN lnc y miARN) dentro de secciones de tejido, células, montajes completos y células tumorales circulantes (CTC). La hibridación in situ fue inventada por la bióloga francesa Mary-Lou Pardue y Joseph G. Gall . [1] [2] [3]
Desafíos de la hibridación in situ [ editar ]
La hibridación in situ es una técnica poderosa para identificar especies de ARNm específicas dentro de células individuales en secciones de tejido, lo que proporciona información sobre los procesos fisiológicos y la patogénesis de la enfermedad. Sin embargo, la hibridación in situ requiere que se tomen muchos pasos con una optimización precisa para cada tejido examinado y para cada sonda utilizada. Para preservar el ARNm diana dentro de los tejidos, a menudo se requiere que se utilicen fijadores de reticulación (como formaldehído ). [4]
Además, la hibridación in situ en cortes de tejido requiere que los cortes de tejido sean muy delgados, normalmente de 3 µm a 7 µm de espesor. Los métodos comunes de preparación de secciones de tejido para el procesamiento de hibridación in situ incluyen cortar muestras con un criostato o un cortador de tejido Compresstome . Un criostato toma tejido fresco o fijo y lo sumerge en nitrógeno líquido para congelación instantánea. Luego, el tejido se incrusta en un medio congelado llamado OCT y se cortan secciones delgadas. Los obstáculos incluyen la aparición de artefactos de congelación en el tejido que pueden interferir con la tinción adecuada del ARNm. El Compresstomecorta el tejido en rodajas finas sin un proceso de congelación; Las secciones que flotan libremente se cortan después de ser incluidas en agarosa para mayor estabilidad. Este método evita la congelación del tejido y, por tanto, los artefactos de congelación asociados. El proceso es permanente e irreversible una vez que se completa. [5]
Proceso [ editar ]
Para la histoquímica de hibridación , las células y los tejidos de la muestra se tratan generalmente para fijar las transcripciones diana en su lugar y aumentar el acceso a la sonda. Como se señaló anteriormente, la sonda es un ADN complementario marcado o, ahora más comúnmente, un ARN complementario ( ribosonda). La sonda se hibrida con la secuencia diana a temperatura elevada, y luego se lava el exceso de sonda (después de la hidrólisis previa usando RNasa en el caso de la sonda de ARN en exceso no hibridada). Los parámetros de la solución, como la temperatura, la sal y / o la concentración de detergente, pueden manipularse para eliminar cualquier interacción no idéntica (es decir, solo quedarán unidas las coincidencias de secuencia exacta). Luego, la sonda que se marcó con bases marcadas con radio, fluorescencia o antígeno (p. Ej., Digoxigenina ) se localiza y cuantifica en el tejido mediante autorradiografía , microscopía de fluorescencia o inmunohistoquímica., respectivamente. ISH también puede usar dos o más sondas, marcadas con radiactividad u otras etiquetas no radiactivas, para detectar simultáneamente dos o más transcripciones.
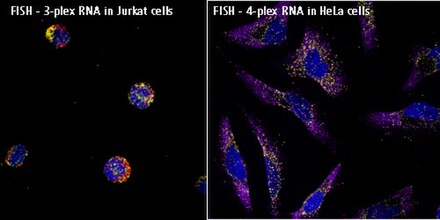
Se puede usar una tecnología alternativa, el ensayo de ADN ramificado , para ensayos de hibridación in situ de ARN (ARNm, ARNlnc y miARN) con sensibilidad de una sola molécula sin el uso de radiactividad. Este enfoque (por ejemplo, ensayos ViewRNA) se puede utilizar para visualizar hasta cuatro dianas en un ensayo, y utiliza un diseño de sonda patentado y amplificación de la señal de bDNA para generar señales sensibles y específicas. Las muestras (células, tejidos y CTC) se fijan y luego se tratan para permitir la accesibilidad del objetivo del ARN (desenmascaramiento del ARN). Las sondas específicas de la diana se hibridan con cada ARN diana. La posterior amplificación de la señal se basa en la hibridación específica de sondas adyacentes ( oligonucleótidos individuales[oligos] que se unen uno al lado del otro en los objetivos de ARN). Una sonda específica de diana típica contendrá 40 oligonucleótidos, lo que dará como resultado 20 pares de oligo que se unen uno al lado del otro en la diana para la detección de ARNm y ARNln, y 2 oligonucleótidos o un solo par para la detección de miARN. La amplificación de la señal se logra mediante una serie de pasos de hibridación secuenciales. Una molécula de preamplificador se hibrida con cada par de oligo en el ARN específico de la diana, luego múltiples moléculas de amplificador se hibridan con cada preamplificador. A continuación, múltiples oligonucleótidos de sonda marcadora (conjugados con fosfatasa alcalina o directamente con fluoróforos) se hibridan con cada molécula amplificadora. Una estructura de amplificación de señal completamente ensamblada "Árbol" tiene 400 sitios de unión para las sondas marcadoras. Cuando todas las sondas específicas de la diana se unen a la transcripción de ARNm diana, un 8,Se produce una amplificación de señal de 000 veces para esa transcripción. Los sistemas de amplificación de señales separados pero compatibles permiten los ensayos multiplex. La señal se puede visualizar usando un microscopio de fluorescencia o de campo claro.
Pasos básicos para las sondas marcadas con digoxigenina [ editar ]
- permeabilización de células con proteinasa K para abrir las membranas celulares (alrededor de 25 minutos, no es necesario para secciones de tejido o algunos embriones en etapa temprana)
- Unión de ARNm a sonda de ARN marcada (generalmente durante la noche)
- Unión de anticuerpo-fosfatasa a sonda de ARN (algunas horas)
- tinción de anticuerpos (p. ej., con fosfatasa alcalina )
El protocolo tarda entre 2 y 3 días y su configuración lleva un tiempo. Algunas empresas venden robots para automatizar el proceso (por ejemplo, Intavis InsituPro VSi ). Como resultado, se han realizado exámenes a gran escala en laboratorios de miles de genes. Por lo general, se puede acceder a los resultados a través de sitios web (ver enlaces externos). [6]
Ver también [ editar ]
- Hibridación cromogénica in situ (CISH)
- Hibridación in situ fluorescente
Referencias [ editar ]
- ^ O'Connor, Clare. "Hibridación de fluorescencia in situ (FISH)" . Educación en la naturaleza.
- ^ Gall, JG ; Pardue, ML (junio de 1969). "Formación y detección de moléculas híbridas ARN-ADN en preparaciones citológicas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 63 (2): 378–83. Código Bibliográfico : 1969PNAS ... 63..378G . doi : 10.1073 / pnas.63.2.378 . PMC 223575 . PMID 4895535 .
- ^ Gall, Joe . "Premio Albert Lasker por logros especiales en ciencia médica" . Fundación Lasker.
- ^ Lehmann, Ruth; Tautz, Diethard (1 de enero de 1994), Goldstein, Lawrence SB; Fyrberg, Eric A. (eds.), "Chapter 30 In Situ Hybridization to RNA" , Methods in Cell Biology , Academic Press, 44 , págs. 575–598, doi : 10.1016 / s0091-679x (08) 60933-4 , recuperado 2021-02-21
- ^ Brown, Lindsay A .; Cazador, David (1 de mayo de 2007). "Hibridación fluorescente in situ en micromatrices de tejidos: desafíos y soluciones" . Revista de Histología Molecular . 38 (2): 151-157. doi : 10.1007 / s10735-006-9069-y . ISSN 1567-2387 .
- ↑ Panoskaltsis-Mortari, A .; Bucy, RP (febrero de 1995). "Hibridación in situ con sondas de ARN marcadas con digoxigenina: hechos y artefactos" . BioTechniques . 18 (2): 300–307. ISSN 0736-6205 . PMID 7727134 .
- Jin, L; Lloyd, RV (1997). "Hibridación in situ: métodos y aplicaciones" . Revista de análisis de laboratorio clínico . 11 (1): 2–9. doi : 10.1002 / (SICI) 1098-2825 (1997) 11: 1 <2 :: AID-JCLA2> 3.0.CO; 2-F . PMC 6760707 . PMID 9021518 .
- Histoquímica de hibridación in situ completa y anotada
- La secuenciación del ARN de las células tumorales circulantes pancreáticas implica la señalización de WNT en la metástasis
- El transcriptoma local en el neurópilo sináptico revelado por secuenciación profunda e imágenes de alta resolución
Enlaces externos [ editar ]
- In + Situ + Hybridization en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .
- Hibridación in situ de sondas de ARN y miARN en células, CTC y tejidos
- Hibridación in situ de montaje completo de sondas de ARN para tejidos vegetales
- Preparación de conjuntos de sondas de ADN complejas para FISH 3D con hasta seis fluorocromos diferentes
- Hibridación de transcripción in situ de embriones de montaje completo para análisis fenotípico de Drosophila tratada con ARNi
- Bases de datos in situ:
- Factores de transcripción de Ghost, C. intestinalis
- Expresión genética del pez cebra
- Expresión del gen MGI GXD de ratón
- eurexpress