Este artículo puede ser demasiado técnico para que la mayoría de los lectores lo comprendan . ( Octubre de 2018 ) ( Obtenga información sobre cómo y cuándo eliminar este mensaje de plantilla ) |
La exhibición de fagos es una técnica de laboratorio para el estudio de interacciones proteína-proteína , proteína - péptido y proteína- ADN que utiliza bacteriófagos ( virus que infectan bacterias ) para conectar proteínas con la información genética que las codifica . [1] En esta técnica, un gen que codifica una proteína de interés se inserta en un gen de la proteína de la cubierta del fago , lo que hace que el fago "muestre" la proteína en su exterior mientras contiene el gen de la proteína en su interior, lo que resulta en una conexión entre genotipo yfenotipo . A continuación, estos fagos de presentación pueden cribarse frente a otras proteínas, péptidos o secuencias de ADN, para detectar la interacción entre la proteína presentada y esas otras moléculas. De esta manera, se pueden cribar y amplificar grandes bibliotecas de proteínas en un proceso llamado selección in vitro , que es análogo a la selección natural .
Los bacteriófagos más comunes utilizados en la presentación en fagos son M13 y fd fago filamentoso , [2] [3] aunque T4 , [4] T7 , y λ fago también se han utilizado.
Historia [ editar ]
La presentación de fagos fue descrita por primera vez por George P. Smith en 1985, cuando demostró la presentación de péptidos en fagos filamentosos (virus largos y delgados que infectan bacterias) fusionando la proteína de la cápside del virus con un péptido de una colección de secuencias de péptidos. [1] Esto mostró los diferentes péptidos en las superficies externas de la colección de clones virales, donde el paso de selección del proceso aisló los péptidos con la mayor afinidad de unión. En 1988, Stephen Parmley y George Smith describieron el biopanning para la selección por afinidad y demostraron que las rondas recursivas de selección podrían enriquecer los clones presentes en 1 en mil millones o menos.[5] En 1990, Jamie Scott y George Smith describieron la creación de grandes bibliotecas de péptidos aleatorios mostradas en fagos filamentosos. [6] La tecnología de visualización de fagos fue desarrollada y mejorada por grupos en el Laboratorio de Biología Molecular con Greg Winter y John McCafferty , el Instituto de Investigación Scripps con Richard Lerner y Carlos Barbas y el Centro Alemán de Investigación del Cáncer con Frank Breitling y Stefan Dübel para exhibición. de proteínas como anticuerpos para la ingeniería de proteínas terapéuticas . Smith y Winter recibieron la mitad del premio Nobel de química 2018 por su contribución al desarrollo de la exhibición de fagos. [7] Una patente de George Pieczenik que reivindica la prioridad de 1985 también describe la generación de bibliotecas de péptidos. [8]
Principio [ editar ]
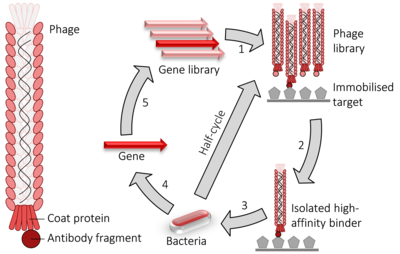
Al igual que el sistema de dos híbridos , la presentación de fagos se utiliza para el cribado de alto rendimiento de interacciones de proteínas. En el caso de la presentación de fagos filamentosos M13 , el ADN que codifica la proteína o péptido de interés se liga en el gen pIII o pVIII, que codifica la proteína de la cubierta menor o mayor , respectivamente. A veces se utilizan múltiples sitios de clonación para asegurar que los fragmentos se inserten en los tres posibles marcos de lectura de modo que el fragmento de ADNc se traduzca en el marco adecuado. El gen del fago y el inserto de ADN híbrido se insertan (un proceso conocido como " transducción ") enCélulas bacterianas de E. coli como TG1, SS320, ER2738 o XL1-Blue E. coli . Sise utiliza un vector " fagémido "(un vector de construcción de presentación simplificada), las partículas de fago no se liberarán de lascélulas de E. coli hasta que se infecten con el fago auxiliar , lo que permite el empaquetamiento del ADN del fago y el ensamblaje de los viriones maduroscon el fragmento de proteína relevante como parte de su recubrimiento externo en la proteína de recubrimiento menor (pIII) o mayor (pVIII). Inmovilizando un objetivo de ADN o proteína relevante en la superficie de una placa de microtitulaciónbueno, un fago que muestra una proteína que se une a uno de esos objetivos en su superficie permanecerá mientras otros se eliminan mediante el lavado. Los que quedan pueden eluirse , usarse para producir más fagos (por infección bacteriana con fagos auxiliares) y para producir una mezcla de fagos enriquecida con fagos relevantes (es decir, de unión). El ciclo repetido de estos pasos se conoce como "cribado" , en referencia al enriquecimiento de una muestra de oro mediante la eliminación de materiales indeseables. El fago eluido en el paso final puede usarse para infectar un huésped bacteriano adecuado, del cual se pueden recolectar los fagémidos y la secuencia de ADN relevante escindida y secuenciada para identificar las proteínas o fragmentos de proteína que interactúan relevantes.
El uso de un fago auxiliar puede eliminarse utilizando la tecnología de "línea celular de empaquetamiento bacteriano". [9]
La elución se puede realizar combinando tampón de elución de pH bajo con sonificación, que, además de aflojar la interacción péptido-diana, también sirve para separar la molécula diana de la superficie de inmovilización. Este método basado en ultrasonidos permite la selección en un solo paso de un péptido de alta afinidad. [10]
Aplicaciones [ editar ]
Las aplicaciones de la tecnología de presentación de fagos incluyen la determinación de los compañeros de interacción de una proteína (que se utilizaría como el "cebo" del fago inmovilizado con una biblioteca de ADN que consta de todas las secuencias codificantes de una célula, tejido u organismo) de modo que la función o el mecanismo de se puede determinar la función de esa proteína. [11] La exhibición de fagos es también un método ampliamente utilizado para la evolución de proteínas in vitro (también llamado ingeniería de proteínas ). Como tal, la presentación de fagos es una herramienta útil en el descubrimiento de fármacos . Se utiliza para encontrar nuevos ligandos (inhibidores de enzimas, agonistas de receptores y antagonistas) para las proteínas diana. [12] [13] [14]La técnica también se utiliza para determinar antígenos tumorales (para su uso en el diagnóstico y la orientación terapéutica) [15] y en la búsqueda de interacciones proteína-ADN [16] utilizando bibliotecas de ADN especialmente construidas con segmentos aleatorizados. Recientemente, la exhibición de fagos también se ha utilizado en el contexto de tratamientos contra el cáncer, como el enfoque de transferencia celular adoptiva. [17] En estos casos, la presentación de fagos se utiliza para crear y seleccionar anticuerpos sintéticos que se dirigen a las proteínas de la superficie del tumor. [17] Estos se convierten en receptores sintéticos para células T recolectadas del paciente que se utilizan para combatir la enfermedad. [18]
Métodos que compiten por in vitro evolución de proteínas incluyen presentación en levadura , pantalla bacteriana , presentación de ribosomas , y pantalla mRNA .
Maduración de anticuerpos in vitro [ editar ]
La invención de la presentación de anticuerpos en fagos revolucionó el descubrimiento de fármacos de anticuerpos. El trabajo inicial fue realizado por los laboratorios del Laboratorio de Biología Molecular del MRC ( Greg Winter y John McCafferty ), el Instituto de Investigación Scripps (Richard Lerner y Carlos F. Barbas) y el Centro Alemán de Investigación del Cáncer (Frank Breitling y Stefan Dübel). [19] [20] [21] En 1991, el grupo Scripps informó sobre la primera presentación y selección de anticuerpos humanos en fagos. [22] Este estudio inicial describió el rápido aislamiento del anticuerpo humano Fab que se une a la toxina del tétanosy el método se extendió luego para clonar rápidamente anticuerpos humanos anti-VIH-1 para el diseño y la terapia de vacunas. [23] [24] [25] [26] [27]
La exhibición de fagos de bibliotecas de anticuerpos se ha convertido en un método poderoso tanto para estudiar la respuesta inmune como para seleccionar y desarrollar rápidamente anticuerpos humanos para la terapia. La presentación de anticuerpos en fagos fue utilizada más tarde por Carlos F.Barbas en el Instituto de Investigación Scripps para crear bibliotecas de anticuerpos humanos sintéticos, un principio patentado por primera vez en 1990 por Breitling y colaboradores (patente CA 2035384), lo que permite la creación in vitro de anticuerpos humanos a partir de anticuerpos sintéticos. elementos de diversidad. [28] [29] [30] [31]
Las bibliotecas de anticuerpos que muestran millones de anticuerpos diferentes en fagos se utilizan a menudo en la industria farmacéutica para aislar conductores de anticuerpos terapéuticos altamente específicos, para su desarrollo en fármacos de anticuerpos principalmente como agentes terapéuticos contra el cáncer o antiinflamatorios. Uno de los más exitosos fue el adalimumab , descubierto por Cambridge Antibody Technology como D2E7 y desarrollado y comercializado por Abbott Laboratories . El adalimumab, un anticuerpo contra el TNF alfa , fue el primer anticuerpo completamente humano del mundo, [32] que logró ventas anuales superiores a los mil millones de dólares. [33]
Protocolo general [ editar ]
A continuación se muestra la secuencia de eventos que se siguen en el cribado de presentación de fagos para identificar polipéptidos que se unen con alta afinidad a la proteína o secuencia de ADN deseada:
- Las proteínas diana o las secuencias de ADN se inmovilizan en los pocillos de una placa de microvaloración .
- Muchas secuencias genéticas se expresan en una biblioteca de bacteriófagos en forma de fusiones con la proteína de la cubierta del bacteriófago, de modo que se muestran en la superficie de la partícula viral. La proteína mostrada corresponde a la secuencia genética dentro del fago.
- Esta biblioteca de presentación de fagos se agrega a la placa y, después de permitir que el fago se una, se lava la placa.
- Las proteínas que muestran los fagos que interactúan con las moléculas diana permanecen adheridas a la placa, mientras que todas las demás se eliminan por lavado.
- Los fagos unidos pueden eluirse y usarse para crear más fagos mediante la infección de huéspedes bacterianos adecuados. El nuevo fago constituye una mezcla enriquecida que contiene considerablemente menos fagos irrelevantes (es decir, que no se unen) que los presentes en la mezcla inicial.
- Los pasos 3 a 5 se repiten opcionalmente una o más veces, enriqueciendo aún más la biblioteca de fagos en proteínas de unión.
- Después de una mayor amplificación basada en bacterias, el ADN dentro del fago que interactúa se secuencia para identificar las proteínas o fragmentos de proteínas que interactúan.
Selección de la proteína de la cubierta [ editar ]
Fagos filamentosos [ editar ]
pIII [ editar ]
pIII es la proteína que determina la infectividad del virión. pIII se compone de tres dominios (N1, N2 y CT) conectados por enlazadores ricos en glicina. [34] El dominio N2 se une al pilus F durante la infección por virión, liberando el dominio N1 que luego interactúa con una proteína TolA en la superficie de la bacteria. [34] Las inserciones dentro de esta proteína generalmente se agregan en la posición 249 (dentro de una región enlazadora entre CT y N2), en la posición 198 (dentro del dominio N2) y en el N-terminal (insertado entre la secuencia de secreción N-terminal y la N -terminus de pIII). [34]Sin embargo, cuando se usa el sitio BamHI ubicado en la posición 198, se debe tener cuidado con el residuo de cisteína no apareado (C201) que podría causar problemas durante la presentación de fagos si se usa una versión no truncada de pIII. [34]
Una ventaja de usar pIII en lugar de pVIII es que pIII permite la presentación monovalente cuando se usa un fagémido (plásmido derivado del fago Ff) combinado con un fago auxiliar. Además, pIII permite la inserción de secuencias de proteínas más grandes (> 100 aminoácidos) [35] y es más tolerante que pVIII. Sin embargo, el uso de pIII como socio de fusión puede conducir a una disminución de la infectividad del fago que conduce a problemas como el sesgo de selección causado por la diferencia en la tasa de crecimiento del fago [36] o, lo que es peor, la incapacidad del fago para infectar a su huésped. [34] La pérdida de la infectividad del fago puede evitarse utilizando un plásmido fagémido y un fago auxiliar de modo que el fago resultante contenga tanto el pIII de tipo salvaje como el de fusión. [34]
El cDNA también se ha analizado utilizando pIII mediante un sistema de dos cremalleras de leucina complementarias, [37] Direct Interaction Rescue [38] o mediante la adición de un enlazador de 8-10 aminoácidos entre el cDNA y pIII en el C-terminal. [39]
pVIII [ editar ]
pVIII es la principal proteína de la cubierta de los fagos Ff. Los péptidos generalmente se fusionan al extremo N-terminal de pVIII. [34] Por lo general, los péptidos que pueden fusionarse con pVIII tienen entre 6 y 8 aminoácidos de longitud. [34] La restricción de tamaño parece tener menos que ver con el impedimento estructural causado por la sección agregada [40] y más con la exclusión de tamaño causada por pIV durante la exportación de proteína de cubierta. [40] Dado que hay alrededor de 2700 copias de la proteína en un fago típico, es más probable que la proteína de interés se exprese polivalentemente incluso si se usa un fagémido. [34] Esto hace que el uso de esta proteína sea desfavorable para el descubrimiento de socios de unión de alta afinidad. [34]
Para superar el problema del tamaño de pVIII, se han diseñado proteínas de cubierta artificiales. [41] Un ejemplo es la proteína de cubierta artificial invertida (ACP) de Weiss y Sidhu, que permite la visualización de proteínas grandes en el extremo C-terminal. [41] Los ACP podrían mostrar una proteína de 20 kDa, sin embargo, solo a niveles bajos (en su mayoría solo monovalentemente). [41]
pVI [ editar ]
pVI se ha utilizado ampliamente para la visualización de bibliotecas de ADNc. [34] La presentación de bibliotecas de ADNc a través de la presentación de fagos es una alternativa atractiva al método de levadura-2-híbrido para el descubrimiento de proteínas y péptidos que interactúan debido a su alta capacidad de rendimiento. [34] pVI se ha utilizado preferentemente para pVIII y pIII para la expresión de bibliotecas de ADNc porque se puede añadir la proteína de interés al extremo C-terminal de pVI sin afectar en gran medida el papel de pVI en el ensamblaje de fagos. Esto significa que el codón de parada en el ADNc ya no es un problema. [42] Sin embargo, la presentación de ADNc en fagos siempre está limitada por la incapacidad de la mayoría de los procariotas para producir modificaciones postraduccionales presentes en las células eucariotas o por el plegamiento incorrecto de proteínas de múltiples dominios.
Si bien pVI ha sido útil para el análisis de bibliotecas de ADNc, pIII y pVIII siguen siendo las proteínas de cubierta más utilizadas para la presentación de fagos. [34]
pVII y pIX [ editar ]
En un experimento en 1995, se intentó la visualización de glutatión S-transferasa tanto en pVII como en pIX y fracasó. [43] Sin embargo, la presentación en fagos de esta proteína se completó con éxito después de la adición de una secuencia señal periplásmica (pelB u ompA) en el extremo N-terminal. [44] En un estudio reciente, se ha demostrado que AviTag, FLAG e His pueden mostrarse en pVII sin la necesidad de una secuencia de señal. Luego, la expresión de los Fv de cadena sencilla (scFv) y los receptores de células T de cadena única (scTCR) se expresaron tanto con como sin la secuencia señal. [45]
PelB (una secuencia señal de aminoácidos que dirige la proteína al periplasma donde una peptidasa señal luego escinde PelB) mejoró el nivel de presentación de fagos en comparación con las fusiones pVII y pIX sin la secuencia señal. Sin embargo, esto condujo a la incorporación de más genomas de fagos auxiliares en lugar de genomas de fagémidos. En todos los casos, los niveles de presentación de fagos fueron más bajos que con la fusión pIII. Sin embargo, la visualización inferior podría ser más favorable para la selección de aglutinantes debido a que la visualización inferior está más cerca de la visualización monovalente real. En cinco de seis ocasiones, las fusiones pVII y pIX sin pelB fueron más eficaces que las fusiones pIII en los ensayos de selección por afinidad. El documento incluso continúa afirmando que las plataformas de visualización pVII y pIX pueden superar a pIII a largo plazo. [45]
El uso de pVII y pIX en lugar de pIII también podría ser una ventaja porque el rescate del virión puede realizarse sin romper el enlace virión-antígeno si el pIII usado es de tipo salvaje. En cambio, se podría escindir en una sección entre la perla y el antígeno para eluir. Dado que el pIII está intacto, no importa si el antígeno permanece unido al fago. [45]
Fagos T7 [ editar ]
El problema de usar fagos Ff para la presentación de fagos es que requieren que la proteína de interés se transloque a través de la membrana interna bacteriana antes de ensamblarse en el fago. [46] Algunas proteínas no pueden someterse a este proceso y, por lo tanto, no pueden mostrarse en la superficie de los fagos Ff. En estos casos, se utiliza en su lugar la visualización de fagos T7. [46] En la presentación del fago T7, la proteína que se mostrará se une al extremo C-terminal de la proteína de la cápside del gen 10 de T7. [46]
La desventaja de usar T7 es que el tamaño de la proteína que se puede expresar en la superficie se limita a péptidos más cortos porque los grandes cambios en el genoma de T7 no se pueden acomodar como en M13 donde el fago solo alarga su capa para adaptarse a la genoma más grande dentro de él. Sin embargo, puede ser útil para la producción de una gran biblioteca de proteínas para la selección de scFV donde el scFV se expresa en un fago M13 y los antígenos se expresan en la superficie del fago T7. [47]
Recursos y herramientas de bioinformática [ editar ]
Las bases de datos y las herramientas computacionales para mimotopos han sido una parte importante del estudio de presentación de fagos. [48] Bases de datos, [49] programas y servidores web [50] se han utilizado ampliamente para excluir péptidos no relacionados con el objetivo, [51] caracterizar las interacciones entre moléculas pequeñas y proteínas y mapear las interacciones entre proteínas y proteínas. Los usuarios pueden usar la estructura tridimensional de una proteína y los péptidos seleccionados del experimento de presentación de fagos para mapear epítopos conformacionales. Algunos de los métodos computacionales rápidos y eficientes están disponibles en línea. [50]
Ver también [ editar ]
- Evolución dirigida
- interacciones proteína-proteína
- Secuencia líder PelB
Técnicas competitivas:
- Sistema de dos híbridos
- visualización de ARNm
- Pantalla de ribosoma
Referencias [ editar ]
- ↑ a b Smith GP (junio de 1985). "Fago de fusión filamentoso: vectores de expresión novedosos que muestran antígenos clonados en la superficie del virión". Ciencia . 228 (4705): 1315–7. Código Bibliográfico : 1985Sci ... 228.1315S . doi : 10.1126 / science.4001944 . PMID 4001944 .
- ^ Smith GP, Petrenko VA (abril de 1997). "Visualización de fagos". Chem. Rev . 97 (2): 391–410. doi : 10.1021 / cr960065d . PMID 11848876 .
- ^ Kehoe JW, Kay BK (noviembre de 2005). "Visualización de fagos filamentosos en el nuevo milenio". Chem. Rev . 105 (11): 4056–72. doi : 10.1021 / cr000261r . PMID 16277371 .
- ↑ Malys N, Chang DY, Baumann RG, Xie D, Black LW (2002). "Una biblioteca de presentación de péptidos aleatorizados de bacteriófago T4 SOC y HOC bipartito: detección y análisis de la interacción del fago T4 terminasa (gp17) y factor sigma tardío (gp55)". J Mol Biol . 319 (2): 289-304. doi : 10.1016 / S0022-2836 (02) 00298-X . PMID 12051907 .
- ^ Parmley SF, Smith GP (1988). "Vectores de fagos fd filamentosos seleccionables por anticuerpos: purificación por afinidad de genes diana". Gene . 73 (2): 305–318. doi : 10.1016 / 0378-1119 (88) 90495-7 . PMID 3149606 .
- ^ Scott, J .; Smith, G. (1990). "Búsqueda de ligandos peptídicos con una biblioteca de epítopos". Ciencia . 249 (4967): 386–390. Código Bibliográfico : 1990Sci ... 249..386S . doi : 10.1126 / science.1696028 . PMID 1696028 .
- ^ "El Premio Nobel de Química 2018" . NobelPrize.org . Consultado el 3 de octubre de 2018 .
- ^ Patente estadounidense 5866363 , Pieczenik G, "Método y medios para clasificar e identificar información biológica", publicada 1999-02-02
- ^ Chasteen L, Ayriss J, Pavlik P, Bradbury AR (2006). "Eliminación de fagos auxiliares de la visualización de fagos" . Ácidos nucleicos Res . 34 (21): e145. doi : 10.1093 / nar / gkl772 . PMC 1693883 . PMID 17088290 .
- ^ Lunder M, Bratkovic T, Urleb U, Kreft S, Strukelj B (junio de 2008). "Ultrasonido en pantalla de fagos: un nuevo enfoque para la elución inespecífica" . BioTechniques . 44 (7): 893–900. doi : 10.2144 / 000112759 . PMID 18533899 .
- ^ Explicación del "mapeo de interacción de proteínas" de The Wellcome Trust
- ^ Lunder M, Bratkovic T, Doljak B, Kreft S, Urleb U, Strukelj B, Plazar N (noviembre de 2005). "Comparación de bibliotecas de péptidos de presentación de fagos y bacterias en busca de motivo de unión a la diana". Apl. Biochem. Biotechnol . 127 (2): 125–31. doi : 10.1385 / ABAB: 127: 2: 125 . PMID 16258189 . S2CID 45243314 .
- ^ Bratkovic T, Lunder M, Popovic T, Kreft S, Turk B, Strukelj B, Urleb U (julio de 2005). "La selección de afinidad por la papaína produce potentes inhibidores de péptidos de las catepsinas L, B, H y K". Biochem. Biophys. Res. Comun . 332 (3): 897–903. doi : 10.1016 / j.bbrc.2005.05.028 . PMID 15913550 .
- ^ Lunder M, Bratkovic T, Kreft S, Strukelj B (julio de 2005). "Inhibidor peptídico de la lipasa pancreática seleccionada por presentación de fagos utilizando diferentes estrategias de elución" . J. Lipid Res . 46 (7): 1512–6. doi : 10.1194 / jlr.M500048-JLR200 . PMID 15863836 .
- ^ Hufton SE, Moerkerk PT, Meulemans EV, de Bruïne A, Arends JW, Hoogenboom HR (diciembre de 1999). "Presentación de fagos de repertorios de ADNc: el sistema de presentación de pVI y sus aplicaciones para la selección de ligandos inmunogénicos". J. Immunol. Métodos . 231 (1–2): 39–51. doi : 10.1016 / S0022-1759 (99) 00139-8 . PMID 10648926 .
- ^ Gommans WM, Haisma HJ, Rots MG (diciembre de 2005). "Ingeniería de factores de transcripción de proteínas de dedos de zinc: la relevancia terapéutica de activar o desactivar la expresión génica endógena a la orden". J. Mol. Biol . 354 (3): 507-19. doi : 10.1016 / j.jmb.2005.06.082 . PMID 16253273 .
- ^ a b "Células T de CAR: células inmunes de pacientes de ingeniería para tratar sus cánceres" . Instituto Nacional del Cáncer . 2013-12-06 . Consultado el 9 de febrero de 2018 .
- ^ Løset GÅ, Berntzen G, Frigstad T, Pollmann S, Gunnarsen KS, Sandlie I (12 de enero de 2015). "Receptores de células T diseñados con visualización de fagos como herramientas para el estudio de interacciones entre péptidos tumorales y MHC" . Fronteras en Oncología . 4 (378): 378. doi : 10.3389 / fonc.2014.00378 . PMC 4290511 . PMID 25629004 .
- ^ McCafferty J , Griffiths AD, Winter G , Chiswell DJ (diciembre de 1990). "Anticuerpos fagos: fagos filamentosos que muestran dominios variables de anticuerpos". Naturaleza . 348 (6301): 552–4. Código bibliográfico : 1990Natur.348..552M . doi : 10.1038 / 348552a0 . PMID 2247164 . S2CID 4258014 .
- ^ Scott JS, Barbas CF III, Burton DA (2001). Visualización de fagos: un manual de laboratorio . Plainview, Nueva York: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-740-2.
- ^ Breitling F, Dübel S, Seehaus T, Klewinghaus I, Little M (agosto de 1991). "Un vector de expresión de superficie para el cribado de anticuerpos". Gene . 104 (2): 147–53. doi : 10.1016 / 0378-1119 (91) 90244-6 . PMID 1916287 .
- ^ Barbas CF, Kang AS, Lerner RA, Benkovic SJ (septiembre de 1991). "Ensamblaje de bibliotecas de anticuerpos combinatorios en superficies de fagos: el sitio del gen III" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 88 (18): 7978–82. Código Bibliográfico : 1991PNAS ... 88.7978B . doi : 10.1073 / pnas.88.18.7978 . PMC 52428 . PMID 1896445 .
- ^ Burton DR, Barbas CF, Persson MA, Koenig S, Chanock RM, Lerner RA (noviembre de 1991). "Una gran variedad de anticuerpos monoclonales humanos contra el virus de inmunodeficiencia humana tipo 1 de bibliotecas combinatorias de individuos seropositivos asintomáticos" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 88 (22): 10134–7. Código Bibliográfico : 1991PNAS ... 8810134B . doi : 10.1073 / pnas.88.22.10134 . PMC 52882 . PMID 1719545 .
- ^ Barbas CF, Björling E, Chiodi F, Dunlop N, Cababa D, Jones TM, Zebedee SL, Persson MA, Nara PL, Norrby E (octubre de 1992). "Fragmentos Fab humanos recombinantes neutralizan el virus de la inmunodeficiencia humana tipo 1 in vitro" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 89 (19): 9339–43. Código Bibliográfico : 1992PNAS ... 89.9339B . doi : 10.1073 / pnas.89.19.9339 . PMC 50122 . PMID 1384050 .
- ^ Burton DR, Pyati J, Koduri R, Sharp SJ, Thornton GB, Parren PW, Sawyer LS, Hendry RM, Dunlop N, Nara PL (noviembre de 1994). "Neutralización eficaz de aislados primarios de VIH-1 por un anticuerpo monoclonal humano recombinante". Ciencia . 266 (5187): 1024–7. Bibcode : 1994Sci ... 266.1024B . doi : 10.1126 / science.7973652 . PMID 7973652 .
- ^ Yang WP, Green K, Pinz-Sweeney S, Briones AT, Burton DR, Barbas CF (diciembre de 1995). "CDR mutagénesis andante para la maduración por afinidad de un potente anticuerpo anti-VIH-1 humano en el rango picomolar". Revista de Biología Molecular . 254 (3): 392–403. doi : 10.1006 / jmbi.1995.0626 . PMID 7490758 .
- ^ Barbas CF, Hu D, Dunlop N, Sawyer L, Cababa D, Hendry RM, Nara PL, Burton DR (abril de 1994). "Evolución in vitro de un anticuerpo humano neutralizante al virus de inmunodeficiencia humana tipo 1 para mejorar la afinidad y ampliar la reactividad cruzada de cepas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (9): 3809-13. Código Bibliográfico : 1994PNAS ... 91.3809B . doi : 10.1073 / pnas.91.9.3809 . PMC 43671 . PMID 8170992 .
- ^ Barbas CF, Bain JD, Hoekstra DM, Lerner RA (mayo de 1992). "Bibliotecas de anticuerpos combinatorios semisintéticos: una solución química al problema de la diversidad" . Proc. Natl. Acad. Sci. USA . 89 (10): 4457–61. Código Bibliográfico : 1992PNAS ... 89.4457B . doi : 10.1073 / pnas.89.10.4457 . PMC 49101 . PMID 1584777 .
- ^ Barbas CF, Languino LR, Smith JW (noviembre de 1993). "Anticuerpos humanos autorreactivos de alta afinidad por diseño y selección: dirigidos al sitio de unión del ligando de la integrina" . Proc. Natl. Acad. Sci. USA . 90 (21): 10003–7. Código Bibliográfico : 1993PNAS ... 9010003B . doi : 10.1073 / pnas.90.21.10003 . PMC 47701 . PMID 7694276 .
- ^ Barbas CF, Wagner J (octubre de 1995). "Anticuerpos humanos sintéticos: selección y evolución de proteínas funcionales". Métodos . 8 (2): 94-103. doi : 10.1006 / meth.1995.9997 .
- ^ Barbas CF (agosto de 1995). "Anticuerpos humanos sintéticos". Nat. Med . 1 (8): 837–9. doi : 10.1038 / nm0895-837 . PMID 7585190 . S2CID 6983649 .
- ^ Lawrence S (abril de 2007). "Bebés de mil millones de dólares - medicamentos biotecnológicos como éxitos de taquilla". Nat. Biotechnol . 25 (4): 380–2. doi : 10.1038 / nbt0407-380 . PMID 17420735 . S2CID 205266758 .
- ^ Cambridge Anticuerpo: actualización de ventas | Anuncios de la empresa | Telégrafo
- ↑ a b c d e f g h i j k l m Lowman HB, Clackson T (2004). "1.3". Visualización de fagos: un enfoque práctico . Oxford [Oxfordshire]: Oxford University Press. págs. 10-11. ISBN 978-0-19-963873-4.
- ^ Sidhu SS, Weiss GA, Wells JA (febrero de 2000). "Visualización de alta copia de proteínas grandes en fagos para selecciones funcionales". J. Mol. Biol . 296 (2): 487–95. doi : 10.1006 / jmbi.1999.3465 . PMID 10669603 .
- ^ Derda R, Tang SK, Whitesides GM (julio de 2010). "Amplificación uniforme de fagos con diferentes características de crecimiento en compartimentos individuales que consisten en gotas monodispersas" . Angew. Chem. En t. Ed. Engl . 49 (31): 5301–4. doi : 10.1002 / anie.201001143 . PMC 2963104 . PMID 20583018 .
- ^ Crameri R, Jaussi R, Menz G, Blaser K (noviembre de 1994). "Visualización de productos de expresión de bibliotecas de ADNc en superficies de fagos. Un sistema de cribado versátil para el aislamiento selectivo de genes por interacción gen-producto / ligando específico". EUR. J. Biochem . 226 (1): 53–8. doi : 10.1111 / j.1432-1033.1994.tb20025.x . PMID 7957259 .
- ^ Gramatikoff K, Georgiev O, Schaffner W (diciembre de 1994). "Rescate de interacción directa, una nueva técnica de fagos filamentosos para estudiar interacciones proteína-proteína" . Ácidos nucleicos Res . 22 (25): 5761–2. doi : 10.1093 / nar / 22.25.5761 . PMC 310144 . PMID 7838733 .
- ^ Fuh G, Sidhu SS (septiembre de 2000). "Exposición eficaz en fagos de polipéptidos fusionados al extremo carboxi de la proteína de cubierta menor del gen-3 M13" . FEBS Lett . 480 (2–3): 231–4. doi : 10.1016 / s0014-5793 (00) 01946-3 . PMID 11034335 . S2CID 23009887 .
- ↑ a b Malik P, Terry TD, Bellintani F, Perham RN (octubre de 1998). "Factores que limitan la visualización de péptidos extraños en la principal proteína de la cubierta de las cápsides de bacteriófagos filamentosos y un papel potencial para la peptidasa líder". FEBS Lett . 436 (2): 263–6. doi : 10.1016 / s0014-5793 (98) 01140-5 . PMID 9781692 . S2CID 19331069 .
- ↑ a b c Weiss GA, Sidhu SS (junio de 2000). "Diseño y evolución de proteínas de recubrimiento artificiales M13". J. Mol. Biol . 300 (1): 213–9. doi : 10.1006 / jmbi.2000.3845 . PMID 10864510 .
- ^ Jespers LS, Messens JH, De Keyser A, Eeckhout D, Van den Brande I, Gansemans YG, Lauwereys MJ, Vlasuk GP, Stanssens PE (abril de 1995). "Expresión de superficie y selección basada en ligandos de ADNc fusionados al gen VI del fago filamentoso". Bio / Tecnología . 13 (4): 378–82. doi : 10.1038 / nbt0495-378 . PMID 9634780 . S2CID 6171262 .
- ^ Endemann H, Model P (julio de 1995). "Ubicación de las proteínas de la cubierta menor del fago filamentoso en el fago y en las células infectadas". J. Mol. Biol . 250 (4): 496–506. doi : 10.1006 / jmbi.1995.0393 . PMID 7616570 .
- ^ Gao C, Mao S, Lo CH, Wirsching P, Lerner RA, Janda KD (mayo de 1999). "Fabricación de anticuerpos artificiales: un formato para la presentación de fagos de matrices heterodiméricas combinatorias" . Proc. Natl. Acad. Sci. USA . 96 (11): 6025-30. Código bibliográfico : 1999PNAS ... 96.6025G . doi : 10.1073 / pnas.96.11.6025 . PMC 26829 . PMID 10339535 .
- ↑ a b c Løset GÅ, Roos N, Bogen B, Sandlie I (2011). "Expandiendo la versatilidad de la presentación de fagos II: selección de afinidad mejorada de dominios plegados en la proteína VII y IX del fago filamentoso" . PLOS ONE . 6 (2): e17433. Código bibliográfico : 2011PLoSO ... 617433L . doi : 10.1371 / journal.pone.0017433 . PMC 3044770 . PMID 21390283 .
- ↑ a b c Danner S, Belasco JG (noviembre de 2001). "Presentación del fago T7: un nuevo sistema de selección genética para clonar proteínas de unión a ARN de bibliotecas de ADNc" . Proc. Natl. Acad. Sci. USA . 98 (23): 12954–9. Código Bibliográfico : 2001PNAS ... 9812954D . doi : 10.1073 / pnas.211439598 . PMC 60806 . PMID 11606722 .
- ^ Castillo J, Goodson B, Winter J (noviembre de 2001). "T7 presentó péptidos como dianas para seleccionar scFv específicos de péptidos de bibliotecas de presentación de scFv de M13". J. Immunol. Métodos . 257 (1–2): 117–22. doi : 10.1016 / s0022-1759 (01) 00454-9 . PMID 11687245 .
- ^ Huang J, Ru B, Dai P (2011). "Recursos y herramientas bioinformáticas para la visualización de fagos" . Moléculas . 16 (1): 694–709. doi : 10,3390 / moléculas16010694 . PMC 6259106 . PMID 21245805 .
- ^ Huang J, Ru B, Zhu P, Nie F, Yang J, Wang X, Dai P, Lin H, Guo FB, Rao N (enero de 2012). "MimoDB 2.0: una base de datos de mimotopos y más" . Ácidos nucleicos Res . 40 (Problema de la base de datos): D271–7. doi : 10.1093 / nar / gkr922 . PMC 3245166 . PMID 22053087 .
- ↑ a b Negi SS, Braun W (2009). "Detección automatizada de epítopos conformacionales utilizando secuencias de péptidos de visualización de fagos" . Bioinform Biol Insights . 3 : 71–81. doi : 10.4137 / BBI.S2745 . PMC 2808184 . PMID 20140073 .
- ^ Huang J, Ru B, Li S, Lin H, Guo FB (2010). "SAROTUP: escáner y reportero de péptidos no relacionados con el objetivo" . J. Biomed. Biotechnol . 2010 : 101932. doi : 10.1155 / 2010/101932 . PMC 2842971 . PMID 20339521 .
Lectura adicional [ editar ]
- Ledsgaard L, Kilstrup M, Karatt-Vellatt A, McCafferty J, Laustsen AH (2018). "Conceptos básicos de la tecnología de presentación de fagos de anticuerpos" (PDF) . Toxinas . 10 (6): 236. doi : 10,3390 / toxins10060236 . PMC 6024766 . PMID 29890762 .
- Selección versus diseño en ingeniería química
- La biblioteca de fagos de anticuerpos humanos ETH-2
- Sidhu SS, Lowman HB, Cunningham BC, Wells JA (2000). "Presentación de fagos para la selección de péptidos de unión novedosos". Meth. Enzymol . Métodos en enzimología. 328 : 333–63. doi : 10.1016 / S0076-6879 (00) 28406-1 . ISBN 9780121822293. PMID 11075354 .
Enlaces externos [ editar ]
| Recursos de la biblioteca sobre la visualización de fagos |
|