Metiltransferasa
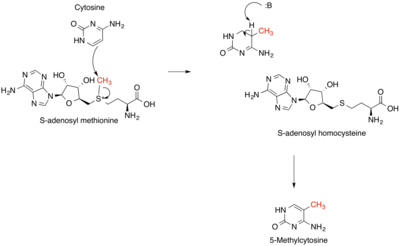
Las metiltransferasas son un gran grupo de enzimas que todas metilan sus sustratos, pero se pueden dividir en varias subclases según sus características estructurales. La clase más común de metiltransferasas es la clase I, todas las cuales contienen un pliegue de Rossmann para unirse a S- adenosil metionina (SAM). Las metiltransferasas de clase II contienen un dominio SET, que se ejemplifica mediante metiltransferasas de histonas de dominio SET , y metiltransferasas de clase III, que están asociadas a la membrana. [1] Las metiltransferasas también se pueden agrupar como diferentes tipos utilizando diferentes sustratos en las reacciones de transferencia de metilo. Estos tipos incluyen proteínas metiltransferasas, ADN / ARN metiltransferasas,metiltransferasas de productos naturales y metiltransferasas no dependientes de SAM. SAM es el donante de metilo clásico para metiltrasferasas, sin embargo, se ven ejemplos de otros donantes de metilo en la naturaleza. El mecanismo general para la transferencia de metilo es un ataque nucleofílico similar al S N 2 donde el azufre de metionina sirve como grupo saliente y el grupo metilo unido a él actúa como el electrófilo que transfiere el grupo metilo al sustrato de la enzima. SAM se convierte en homocisteína S -adenosil(SAH) durante este proceso. La ruptura del enlace SAM-metilo y la formación del enlace sustrato-metilo ocurren casi simultáneamente. Estas reacciones enzimáticas se encuentran en muchas vías y están implicadas en enfermedades genéticas, cáncer y enfermedades metabólicas. Otro tipo de transferencia de metilo es el radical S-adenosil metionina (SAM), que es la metilación de átomos de carbono inactivados en metabolitos primarios, proteínas, lípidos y ARN.
La metilación, así como otras modificaciones epigenéticas , afecta la transcripción , la estabilidad genética y la impronta parental . [2] Impacta directamente en la estructura de la cromatina y puede modular la transcripción de genes, o incluso silenciar o activar completamente los genes, sin mutar el gen en sí. Aunque los mecanismos de este control genético son complejos, la hipo e hipermetilación del ADN está implicada en muchas enfermedades.
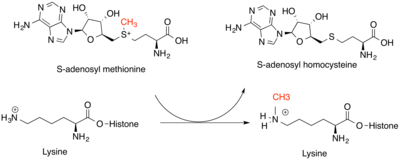
La metilación de proteínas tiene una función reguladora en las interacciones proteína-proteína , las interacciones proteína-ADN y la activación de proteínas.
Ejemplos: RCC1 , una proteína mitótica importante , está metilada para que pueda interactuar con los centrómeros de los cromosomas. Este es un ejemplo de regulación de la interacción proteína-proteína, ya que la metilación regula la unión de RCC1 a las proteínas histonas H2A y H2B . La interacción RCC1-cromatina también es un ejemplo de una interacción proteína-ADN, ya que otro dominio de RCC1 interactúa directamente con el ADN cuando esta proteína está metilada. Cuando RCC1 no está metilado, las células en división tienen múltiples polos de huso y, por lo general, no pueden sobrevivir.
p53 metilado en lisina para regular su activación e interacción con otras proteínas en la respuesta al daño del ADN. Este es un ejemplo de regulación de las interacciones proteína-proteína y activación de proteínas. p53 es un conocido supresor de tumores que activa las vías de reparación del ADN , inicia la apoptosis y detiene el ciclo celular . En general, responde a mutaciones en el ADN, enviando señales a la célula para que las arregle o inicie la muerte celular para que estas mutaciones no contribuyan al cáncer.
NF-κB (una proteína involucrada en la inflamación) es un conocido objetivo de metilación de la metiltransferasa SETD6 , que desactiva la señalización de NF-κB al inhibir una de sus subunidades, RelA . Esto reduce la activación transcripcional y la respuesta inflamatoria , lo que hace que la metilación de NF-κB sea un proceso regulador por el cual se reduce la señalización celular a través de esta vía. [3]