| Cascade (complejo asociado a CRISPR para defensa antiviral) | |
|---|---|
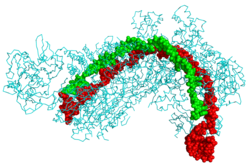 Proteína CRISPR Cascade (cian) unida al ARN CRISPR (verde) y al ADN del fago (rojo) [1] | |
| Identificadores | |
| Organismo | |
| Símbolo | CRISPR |
| PDB | 4QYZ |
| Parte de una serie sobre |
| Ingeniería genética |
|---|
 |
| Organismos genéticamente modificados |
| Historia y regulación |
| Proceso |
| Aplicaciones |
| Controversias |
CRISPR ( / k r ɪ s p ər / ) (que es un acrónimo de agrupadas repeticiones palindrómicas cortas espaciadas regularmente ) es una familia de ADN secuencias encontradas en los genomas de procariotas organismos tales como bacterias y arqueas . [2] Estas secuencias se derivan de fragmentos de ADN de bacteriófagos.que había infectado previamente al procariota. Se utilizan para detectar y destruir el ADN de bacteriófagos similares durante infecciones posteriores. Por tanto, estas secuencias juegan un papel clave en el sistema de defensa antivírico (es decir, anti-fagos) de los procariotas y proporcionan una forma de inmunidad adquirida . [2] [3] [4] [5] CRISPR se encuentran en aproximadamente el 50% de los genomas bacterianos secuenciados y casi el 90% de las arqueas secuenciadas. [6]
Cas9 (o "proteína 9 asociada a CRISPR") es una enzima que utiliza secuencias CRISPR como guía para reconocer y escindir hebras específicas de ADN que son complementarias a la secuencia CRISPR. Las enzimas Cas9 junto con las secuencias CRISPR forman la base de una tecnología conocida como CRISPR-Cas9 que se puede utilizar para editar genes dentro de organismos. [8] Este proceso de edición tiene una amplia variedad de aplicaciones, incluida la investigación biológica básica, el desarrollo de productos biotecnológicos y el tratamiento de enfermedades. [9] [10] La técnica de edición del genoma CRISPR-Cas9 contribuyó significativamente al premio Nobel de Química en 2020 otorgado a Emmanuelle Charpentiery Jennifer Doudna . [11] [12]
Historia [ editar ]
Secuencias repetidas [ editar ]
El descubrimiento de repeticiones de ADN agrupadas se produjo de forma independiente en tres partes del mundo. La primera descripción de lo que más tarde se llamaría CRISPR es del investigador de la Universidad de Osaka Yoshizumi Ishino y sus colegas en 1987. Clonaron accidentalmente parte de una secuencia CRISPR junto con el gen " iap" (conversión de isoenzimas de fosfatasa alcalina) [13] que estaba su objetivo. La organización de las repeticiones fue inusual. Las secuencias repetidas se organizan típicamente de forma consecutiva, sin secuencias diferentes intercaladas. [13] [10] No conocían la función de las repeticiones agrupadas interrumpidas.
En 1993, investigadores de Mycobacterium tuberculosis en los Países Bajos publicaron dos artículos sobre un grupo de repeticiones directas interrumpidas (RD) en esa bacteria. Reconocieron la diversidad de las secuencias que intervienen en las repeticiones directas entre diferentes cepas de M. tuberculosis [14] y utilizaron esta propiedad para diseñar un método de tipificación que se denominó spoligotyping , que todavía se utiliza en la actualidad. [15] [16]
Francisco Mojica de la Universidad de Alicante en España estudió las repeticiones observadas en los organismos arqueales de las especies Haloferax y Haloarcula , y su función. El supervisor de Mojica supuso en ese momento que las repeticiones agrupadas tenían un papel en la segregación correcta del ADN replicado en las células hijas durante la división celular porque los plásmidos y cromosomas con matrices de repeticiones idénticas no podían coexistir en Haloferax volcanii . También se observó por primera vez la transcripción de las repeticiones interrumpidas; esta fue la primera caracterización completa de CRISPR. [16] [17]Para el año 2000, Mojica realizó una encuesta de literatura científica y uno de sus estudiantes realizó una búsqueda en genomas publicados con un programa ideado por él mismo. Identificaron repeticiones interrumpidas en 20 especies de microbios como pertenecientes a la misma familia. [18] En 2001, Mojica y Ruud Jansen , que buscaban repeticiones interrumpidas adicionales, propusieron el acrónimo CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) para aliviar la confusión derivada de los numerosos acrónimos utilizados para describir las secuencias en la literatura científica. [17] [19] En 2002, Tang, et al. mostró evidencia de que CRISPR repite regiones del genoma de Archaeoglobus fulgidusse transcribieron en moléculas de ARN largas que posteriormente se procesaron en ARN pequeños de longitud unitaria, además de algunas formas más largas de 2, 3 o más unidades espaciadoras-repetidas. [20] [21]
En 2005, el investigador de yogur Rodolphe Barrangou , descubrió que Streptococcus thermophilus , después de desafíos iterativos de fagos, desarrolla una mayor resistencia a los fagos, y esta mayor resistencia se debe a la incorporación de secuencias espaciadoras CRISPR adicionales. [22] La empresa de alimentos danesa Danisco, para la que en ese momento trabajaba Barrangou, desarrolló luego cepas de S. thermophilus resistentes a los fagos para su uso en la producción de yogur. Más tarde, Danisco fue comprada por DuPont , que "posee alrededor del 50 por ciento del mercado mundial de cultivos lácteos" y la tecnología se generalizó. [23]
Sistemas asociados a CRISPR [ editar ]
Una importante adición a la comprensión de CRISPR vino con la observación de Jansen de que el grupo de repeticiones de procariotas estaba acompañado por un conjunto de genes homólogos que componen los sistemas asociados a CRISPR o genes cas . Inicialmente se reconocieron cuatro genes cas ( cas 1-4). Las proteínas Cas mostraron motivos de helicasa y nucleasa , lo que sugiere un papel en la estructura dinámica de los loci CRISPR. [24] En esta publicación se utilizó el acrónimo CRISPR como el nombre universal de este patrón. Sin embargo, la función CRISPR siguió siendo enigmática.
En 2005, tres grupos de investigación independientes demostraron que algunos espaciadores CRISPR se derivan del ADN del fago y del ADN extracromosómico , como los plásmidos . [28] [29] [30] En efecto, los espaciadores son fragmentos de ADN recolectados de virus que previamente intentaron atacar la célula. La fuente de los espaciadores fue una señal de que el sistema CRISPR / cas podría tener un papel en la inmunidad adaptativa en las bacterias . [25] [31] Los tres estudios que proponen esta idea fueron inicialmente rechazados por revistas de alto perfil, pero finalmente aparecieron en otras revistas. [32]
La primera publicación [29] que propone un papel de CRISPR-Cas en la inmunidad microbiana, de Mojica y colaboradores de la Universidad de Alicante , predijo un papel para la transcripción de ARN de los espaciadores en el reconocimiento de blancos en un mecanismo que podría ser análogo a la interferencia del ARN. sistema utilizado por las células eucariotas. Koonin y sus colegas ampliaron esta hipótesis de interferencia de ARN al proponer mecanismos de acción para los diferentes subtipos CRISPR-Cas de acuerdo con la función predicha de sus proteínas. [33]
El trabajo experimental de varios grupos reveló los mecanismos básicos de la inmunidad CRISPR-Cas. En 2007, se publicó la primera evidencia experimental de que CRISPR era un sistema inmunológico adaptativo. [10] [4] Una región CRISPR en Streptococcus thermophilus adquirió espaciadores del ADN de un bacteriófago infectante . Los investigadores manipularon la resistencia de S. thermophilus a diferentes tipos de fagos agregando y eliminando espaciadores cuya secuencia coincidía con las encontradas en los fagos probados. [34] [35] En 2008, Brouns y Van der Oost identificaron un complejo de proteínas Cas (llamado Cascade) que en E. colicortar el precursor de ARN CRISPR dentro de las repeticiones en moléculas de ARN maduras que contienen espaciador llamadas ARN CRISPR (ARNcr), que permaneció unido al complejo proteico. [36] Además, se descubrió que se requería Cascade, crRNA y una helicasa / nucleasa ( Cas3 ) para proporcionar a un huésped bacteriano inmunidad contra la infección por un virus de ADN . Al diseñar un CRISPR antivirus, demostraron que dos orientaciones del crRNA (sentido / antisentido) proporcionaban inmunidad, lo que indica que las guías del crRNA estaban dirigidas al dsDNA. Ese año Marraffini y Sontheimer confirmaron que una secuencia CRISPR de S. epidermidis apuntaba al ADN y no al ARN para evitar la conjugación.. Este hallazgo estaba en desacuerdo con el mecanismo propuesto similar a la interferencia de ARN de la inmunidad CRISPR-Cas, aunque más tarde se encontró un sistema CRISPR-Cas que se dirige al ARN extraño en Pyrococcus furiosus . [10] [34] Un estudio de 2010 mostró que CRISPR-Cas corta ambas cadenas de ADN de fago y plásmido en S. thermophilus . [37]
Cas9 [ editar ]
Los investigadores estudiaron un sistema CRISPR más simple de Streptococcus pyogenes que se basa en la proteína Cas9 . La endonucleasa Cas9 es un sistema de cuatro componentes que incluye dos pequeñas moléculas de crRNA y ARN CRISPR trans-activador (tracrRNA). [38] [39] Jennifer Doudna y Emmanuelle Charpentier rediseñaron la endonucleasa Cas9 en un sistema de dos componentes más manejable fusionando las dos moléculas de ARN en un "ARN de guía única" que, cuando se combinaba con Cas9, podía encontrar y cortar el ADN diana especificado por el ARN guía. Esta contribución fue tan significativa que fue reconocida por el Premio Nobel de Química.en 2020. Al manipular la secuencia de nucleótidos del ARN guía, el sistema artificial Cas9 podría programarse para apuntar a cualquier secuencia de ADN para la escisión. [40] Otro grupo de colaboradores que comprende Virginijus Šikšnys junto con Gasiūnas, Barrangou y Horvath mostró que Cas9 del sistema CRISPR de S. thermophilus también se puede reprogramar para apuntar a un sitio de su elección cambiando la secuencia de su crRNA. Estos avances impulsaron los esfuerzos para editar genomas con el sistema CRISPR-Cas9 modificado. [dieciséis]
Los grupos liderados por Feng Zhang y George Church publicaron simultáneamente descripciones de la edición del genoma en cultivos de células humanas utilizando CRISPR-Cas9 por primera vez. [10] [41] [42] Desde entonces se ha utilizado en una amplia gama de organismos, incluida la levadura de panadería ( Saccharomyces cerevisiae ), [43] [44] [45] el patógeno oportunista Candida albicans , [46] [47] pez cebra ( Danio rerio ), [48] moscas de la fruta ( Drosophila melanogaster ), [49] [50] hormigas ( Harpegnathos saltator[51] y Ooceraea biroi [52] ), mosquitos ( Aedes aegypti [53] ), nematodos ( Caenorhabditis elegans ), [54] plantas, [55] ratones, [56] [57] monos [58] y embriones humanos. [59]
CRISPR se ha modificado para producir factores de transcripción programables que permiten a los científicos apuntar y activar o silenciar genes específicos. [60]
El sistema CRISPR-Cas9 ha demostrado realizar ediciones genéticas efectivas en cigotos tripronucleares humanos descritos por primera vez en un artículo de 2015 de los científicos chinos P. Liang e Y. Xu. El sistema realizó una escisión exitosa de la beta-hemoglobina mutante (HBB) en 28 de 54 embriones. 4 de los 28 embriones se recombinaron con éxito utilizando una plantilla de donante proporcionada por los científicos. Los científicos demostraron que durante la recombinación del ADN de la hebra escindida, la secuencia endógena homóloga HBD compite con la plantilla del donante exógeno. La reparación del ADN en embriones humanos es mucho más complicada y particular que en las células madre derivadas. [61]
Cas12a (anteriormente Cpf1) [ editar ]
En 2015, la nucleasa Cas12a (antes conocida como Cpf1 [62] ) se caracterizó en el sistema CRISPR / Cpf1 de la bacteria Francisella novicida . [63] [64] Su nombre original, de una definición de familia de proteínas TIGRFAM construida en 2012, refleja la prevalencia de su subtipo CRISPR-Cas en los linajes Prevotella y Francisella . Cas12a mostró varias diferencias clave de Cas9, que incluyen: causar un corte 'escalonado' en el ADN de doble hebra en lugar del corte 'contundente' producido por Cas9, que se basa en un PAM 'T rico'(que proporciona sitios de orientación alternativos a Cas9) y requiere solo un ARN CRISPR (crRNA) para una orientación exitosa. Por el contrario, Cas9 requiere tanto crRNA como un crRNA transactivante (tracrRNA).
Estas diferencias pueden darle a Cas12a algunas ventajas sobre Cas9. Por ejemplo, los pequeños crRNA de Cas12a son ideales para la edición del genoma multiplexado, ya que se pueden empaquetar más en un vector que los sgRNA de Cas9. Además, los salientes pegajosos 5 'que deja Cas12a se pueden usar para el ensamblaje de ADN que es mucho más específico de la diana que la clonación tradicional de enzimas de restricción. [65] Por último, Cas12a escinde el ADN de 18 a 23 pares de bases aguas abajo del sitio PAM. Esto significa que no hay interrupción de la secuencia de reconocimiento después de la reparación, por lo que Cas12a permite múltiples rondas de escisión del ADN. Por el contrario, dado que Cas9 corta solo 3 pares de bases aguas arriba del sitio PAM, la vía NHEJ da como resultado indelmutaciones que destruyen la secuencia de reconocimiento, evitando así más rondas de corte. En teoría, las rondas repetidas de escisión del ADN deberían aumentar la oportunidad de que se produzca la edición genómica deseada. [66] Una característica distintiva de Cas12a, en comparación con Cas9, es que después de cortar su objetivo, Cas12a permanece unido al objetivo y luego escinde otras moléculas de ssDNA de forma no discriminatoria. [67] Esta propiedad se denomina actividad de "escisión colateral" o "trans-escisión" y se ha aprovechado para el desarrollo de diversas tecnologías de diagnóstico. [68] [69]
Cas13 (anteriormente C2c2) [ editar ]
En 2016 se caracterizó la nucleasa Cas13a (antes conocida como C2c2) de la bacteria Leptotrichia shahii . Cas13 es una endonucleasa de ARN guiada por ARN, lo que significa que no escinde el ADN, sino solo el ARN monocatenario. Cas13 es guiado por su crRNA a un objetivo de ssRNA y se une y escinde el objetivo. De manera similar a Cas12a, el Cas13 permanece unido al objetivo y luego escinde otras moléculas de ssRNA de forma no discriminatoria. [70] Esta propiedad de escisión colateral se ha aprovechado para el desarrollo de diversas tecnologías de diagnóstico. [71] [72] [73]
Estructura del lugar [ editar ]
Repeticiones y espaciadores [ editar ]
La matriz CRISPR se compone de una secuencia líder rica en AT seguida de repeticiones cortas que están separadas por espaciadores únicos. [74] Las repeticiones CRISPR suelen tener un tamaño de 28 a 37 pares de bases (bps), aunque puede haber tan solo 23 pb y hasta 55 pb. [75] Algunos muestran simetría de díadas , lo que implica la formación de una estructura secundaria como un tallo-bucle ('horquilla') en el ARN, mientras que otros están diseñados para no estar estructurados. El tamaño de los espaciadores en diferentes matrices CRISPR es típicamente de 32 a 38 pb (rango de 21 a 72 pb). [75] Pueden aparecer rápidamente nuevos espaciadores como parte de la respuesta inmunitaria a la infección por fagos. [76]Por lo general, hay menos de 50 unidades de la secuencia del espaciador repetido en una matriz CRISPR. [75]
Estructuras de ARN CRISPR [ editar ]
- CRISPR-DR2
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01315 .
- CRISPR-DR5
- Estructura secundaria extraída de la base de datos Rfam . Familia RF011318 .
- CRISPR-DR6
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01319 .
- CRISPR-DR8
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01321 .
- CRISPR-DR9
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01322 .
- CRISPR-DR19
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01332 .
- CRISPR-DR41
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01350 .
- CRISPR-DR52
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01365 .
- CRISPR-DR57
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01370 .
- CRISPR-DR65
- Estructura secundaria extraída de la base de datos Rfam . Familia RF01378 .
Genes cas y subtipos CRISPR [ editar ]
Los pequeños grupos de genes cas a menudo se encuentran junto a las matrices de espaciadores de repetición CRISPR. En conjunto, los 93 genes cas se agrupan en 35 familias basándose en la similitud de secuencia de las proteínas codificadas. 11 de las 35 familias forman el núcleo cas , que incluye las familias de proteínas Cas1 a Cas9. Un locus CRISPR-Cas completo tiene al menos un gen que pertenece al núcleo cas . [77]
Los sistemas CRISPR-Cas se dividen en dos clases. Los sistemas de clase 1 utilizan un complejo de múltiples proteínas Cas para degradar los ácidos nucleicos extraños. Los sistemas de clase 2 utilizan una única proteína Cas grande para el mismo propósito. La clase 1 se divide en tipos I, III y IV; la clase 2 se divide en los tipos II, V y VI. [78] Los 6 tipos de sistemas se dividen en 19 subtipos. [79] Cada tipo y la mayoría de los subtipos se caracterizan por un "gen característico" que se encuentra casi exclusivamente en la categoría. La clasificación también se basa en el complemento de genes cas que están presentes. La mayoría de los sistemas CRISPR-Cas tienen una proteína Cas1. La filogenia de las proteínas Cas1 generalmente concuerda con el sistema de clasificación. [77]Muchos organismos contienen múltiples sistemas CRISPR-Cas, lo que sugiere que son compatibles y pueden compartir componentes. [80] [81] La distribución esporádica de los subtipos CRISPR / Cas sugiere que el sistema CRISPR / Cas está sujeto a la transferencia horizontal de genes durante la evolución microbiana .
En esta tabla falta información sobre la referencia cruzada de UniProt e InterPro. ( Octubre de 2020 ) |
| Clase | Tipo cas | Subtipo cas | Proteína de firma | Función | Referencia |
|---|---|---|---|---|---|
| 1 | I | - | Cas3 | ADN nucleasa monocatenaria (dominio HD) y helicasa dependiente de ATP | [82] [83] |
| I A | Cas8a, Cas5 | Cas8 es una subunidad del módulo de interferencia que es importante en el direccionamiento del ADN invasor mediante el reconocimiento de la secuencia PAM . Se requiere Cas5 para el procesamiento y la estabilidad de los crRNA | [77] [84] | ||
| IB | Cas8b | ||||
| IC | Cas8c | ||||
| IDENTIFICACIÓN | Cas10d | contiene un dominio homólogo al dominio palm de polimerasas de ácidos nucleicos y ciclasas de nucleótidos | [85] [86] | ||
| ES DECIR | Cse1, Cse2 | ||||
| SI | Csy1, Csy2, Csy3 | No determinado | [77] | ||
| IG [Nota 1] | GSU0054 | [87] | |||
| III | - | Cas10 | Homólogo de Cas10d y Cse1. Se une al ARN objetivo de CRISPR y promueve la estabilidad del complejo de interferencia | [86] [88] | |
| III-A | Csm2 | No determinado | [77] | ||
| III-B | Cmr5 | No determinado | [77] | ||
| III-C | Cas10 o Csx11 | [77] [88] | |||
| III-D | Csx10 | [77] | |||
| III-E | [87] | ||||
| III-F | [87] | ||||
| IV | - | Csf1 | [87] | ||
| IV-A | [87] | ||||
| IV-B | [87] | ||||
| IV-C | [87] | ||||
| 2 | II | - | Cas9 | Las nucleasas RuvC y HNH juntas producen DSB y por separado pueden producir roturas de una sola hebra. Asegura la adquisición de espaciadores funcionales durante la adaptación. | [89] [90] |
| II-A | Csn2 | Proteína de unión al ADN en forma de anillo. Involucrado en la adaptación cebada en el sistema CRISPR Tipo II. | [91] | ||
| II-B | Cas4 | Endonucleasa que trabaja con cas1 y cas2 para generar secuencias espaciadoras | [92] | ||
| II-C | Caracterizado por la ausencia de Csn2 o Cas4 | [93] | |||
| V | - | Cas12 | Nuclease RuvC. Carece de HNH. | [78] [94] | |
| Virginia | Cas12a (Cpf1) | [87] | |||
| VB | Cas12b (C2c1) | [87] | |||
| VC | Cas12c (C2c3) | [87] | |||
| enfermedad venérea | Cas12d (CasY) | [87] | |||
| VE | Cas12e (CasX) | [87] | |||
| VF | Cas12f (Cas14, C2c10) | [87] | |||
| VG | Cas12g | [87] | |||
| VH | Cas12h | [87] | |||
| VI | Cas12i | [87] | |||
| VK [Nota 2] | Cas12k (C2c5) | [87] | |||
| VU | C2c4, C2c8, C2c9 | [87] | |||
| VI | - | Cas13 | ARNasa guiada por ARN | [78] [95] | |
| VÍA | Cas13a (C2c2) | [87] | |||
| VI-B | Cas13b | [87] | |||
| VI-C | Cas13c | [87] | |||
| VI-D | Cas13d | [87] |
Mecanismo [ editar ]
La inmunidad CRISPR-Cas es un proceso natural de bacterias y arqueas. [96] CRISPR-Cas previene la infección por bacteriófagos, la conjugación y la transformación natural al degradar los ácidos nucleicos extraños que ingresan a la célula. [34]
Adquisición de espaciador [ editar ]
Cuando un bacteriófago invade un microbio , la primera etapa de la respuesta inmune es capturar el ADN del fago e insertarlo en un locus CRISPR en forma de espaciador. Cas1 y Cas2 se encuentran en ambos tipos de sistemas inmunológicos CRISPR-Cas, lo que indica que están involucrados en la adquisición de espaciadores. Los estudios de mutación confirmaron esta hipótesis, mostrando que la eliminación de cas1 o cas2 detuvo la adquisición del espaciador, sin afectar la respuesta inmune CRISPR. [97] [98] [99] [100] [101]
Se han caracterizado múltiples proteínas Cas1 y se han resuelto sus estructuras. [102] [103] [104] Las proteínas Cas1 tienen diversas secuencias de aminoácidos . Sin embargo, sus estructuras cristalinas son similares y todas las proteínas Cas1 purificadas son nucleasas / integrasas dependientes de metales que se unen al ADN de manera independiente de la secuencia. [80] proteínas Representante CAS2 se han caracterizado y poseer ya sea (hebra individual) ssRNA- [105] o (doble cadena) dsDNA- [106] [107] específica endoribonuclease actividad.
En el sistema IE de E. coli, Cas1 y Cas2 forman un complejo donde un dímero Cas2 une dos dímeros Cas1. [108] En este complejo, Cas2 desempeña un papel de andamiaje no enzimático, [108] uniendo fragmentos bicatenarios de ADN invasor, mientras que Cas1 une los flancos monocatenarios del ADN y cataliza su integración en matrices CRISPR. [109] [110] [111] Por lo general, se agregan nuevos espaciadores al comienzo del CRISPR junto a la secuencia líder, lo que crea un registro cronológico de las infecciones virales. [112] En E. coli, una proteína similar a una histona llamada factor de integración del huésped ( IHF), que se une a la secuencia líder, es responsable de la precisión de esta integración. [113] IHF también mejora la eficiencia de integración en el sistema de tipo IF de Pectobacterium atrosepticum . [114] pero en otros sistemas pueden ser necesarios diferentes factores de hospedaje [115]
Motivos adyacentes al protospacer [ editar ]
El análisis bioinformático de regiones de genomas de fagos que se escindieron como espaciadores (denominados protoespaciadores) reveló que no se seleccionaron al azar, sino que se encontraron adyacentes a secuencias de ADN cortas (3-5 pb) denominadas motivos adyacentes protoespaciadores (PAM). El análisis de los sistemas CRISPR-Cas mostró que los PAM son importantes para los sistemas de tipo I y II, pero no los de tipo III durante la adquisición. [30] [116] [117] [118] [119] [120] En los sistemas tipo I y tipo II, los protoespaciadores se extirpan en posiciones adyacentes a una secuencia PAM, con el otro extremo del espaciador cortado utilizando un mecanismo de regla, manteniendo así la regularidad del tamaño del espaciador en la matriz CRISPR. [121] [122]La conservación de la secuencia PAM difiere entre los sistemas CRISPR-Cas y parece estar vinculada evolutivamente a Cas1 y la secuencia líder . [120] [123]
Se agregan nuevos espaciadores a una matriz CRISPR de manera direccional, [28] ocurriendo preferentemente, [76] [116] [117] [124] [125] pero no exclusivamente, adyacentes [119] [122] a la secuencia líder. El análisis del sistema de tipo IE de E. coli demostró que se copia la primera repetición directa adyacente a la secuencia líder, con el espaciador recién adquirido insertado entre la primera y la segunda repeticiones directas. [100] [121]
La secuencia de PAM parece ser importante durante la inserción del espaciador en sistemas de tipo IE. Esa secuencia contiene un nucleótido final fuertemente conservado (nt) adyacente al primer nt del protoespaciador. Este nt se convierte en la base final en la primera repetición directa. [101] [126] [127] Esto sugiere que la maquinaria de adquisición del espaciador genera voladizos monocatenarios en la penúltima posición de la repetición directa y en el PAM durante la inserción del espaciador. Sin embargo, no todos los sistemas CRISPR-Cas parecen compartir este mecanismo ya que las PAM en otros organismos no muestran el mismo nivel de conservación en la posición final. [123] Es probable que en esos sistemas, se genere un extremo romo al final de la repetición directa y el protoespaciador durante la adquisición.
Variantes de inserción [ editar ]
El análisis de CRISPR de Sulfolobus solfataricus reveló más complejidades al modelo canónico de inserción de espaciadores, ya que uno de sus seis loci CRISPR insertó nuevos espaciadores al azar a lo largo de su matriz CRISPR, en lugar de insertar los más cercanos a la secuencia líder. [122]
Varios CRISPR contienen muchos espaciadores para el mismo fago. El mecanismo que causa este fenómeno fue descubierto en el sistema tipo IE de E. coli . Se detectó una mejora significativa en la adquisición del espaciador donde los espaciadores ya apuntan al fago, incluso los desajustes con el protoespaciador. Este 'cebado' requiere que las proteínas Cas involucradas tanto en la adquisición como en la interferencia interactúen entre sí. Los espaciadores recién adquiridos que resultan del mecanismo de cebado siempre se encuentran en la misma hebra que el espaciador de cebado. [101] [126] [127] Esta observación llevó a la hipótesis de que la maquinaria de adquisición se desliza a lo largo del ADN extraño después de cebar para encontrar un nuevo protoespaciador. [127]
Biogénesis [ editar ]
CRISPR-RNA (crRNA), que luego guía la nucleasa Cas hacia el objetivo durante el paso de interferencia, debe generarse a partir de la secuencia CRISPR. El crRNA se transcribe inicialmente como parte de una única transcripción larga que abarca gran parte de la matriz CRISPR. [26] Esta transcripción luego es escindida por proteínas Cas para formar crRNA. El mecanismo para producir crRNAs difiere entre los sistemas CRISPR / Cas. En los sistemas de tipo IE y tipo IF, las proteínas Cas6e y Cas6f, respectivamente, reconocen los bucles de tallo [128] [129] [130] creados por el emparejamiento de repeticiones idénticas que flanquean el crRNA. [131] Estas proteínas Cas escinden la transcripción más larga en el borde de la región pareada, dejando un solo crRNA junto con un pequeño remanente de la región de repetición pareada.
Los sistemas de tipo III también usan Cas6, sin embargo, sus repeticiones no producen bucles de vástago. En cambio, la escisión se produce por la transcripción más larga que envuelve el Cas6 para permitir la escisión justo aguas arriba de la secuencia repetida. [132] [133] [134]
Los sistemas de tipo II carecen del gen Cas6 y en su lugar utilizan RNaseIII para la escisión. Los sistemas funcionales de tipo II codifican un ARN extrapequeño que es complementario a la secuencia repetida, conocido como ARNcr trans-activador (tracrRNA). [38] La transcripción del tracrRNA y el transcrito primario de CRISPR da como resultado el apareamiento de bases y la formación de dsRNA en la secuencia repetida, que posteriormente es el objetivo de RNaseIII para producir crRNA. A diferencia de los otros dos sistemas, el crRNA no contiene el espaciador completo, que en su lugar está truncado en un extremo. [89]
Los ARNr se asocian con las proteínas Cas para formar complejos de ribonucleótidos que reconocen ácidos nucleicos extraños. Los ARNr no muestran preferencia entre las cadenas codificantes y no codificantes, lo que es indicativo de un sistema de dirección de ADN guiado por ARN. [5] [37] [97] [101] [135] [136] [137] El complejo de tipo IE (comúnmente denominado Cascade) requiere cinco proteínas Cas unidas a un solo crRNA. [138] [139]
Interferencia [ editar ]
Durante la etapa de interferencia en los sistemas de tipo I, la secuencia de PAM se reconoce en la hebra complementaria de crRNA y se requiere junto con la hibridación de crRNA. En los sistemas de tipo I, el apareamiento de bases correcto entre el crRNA y el protoespaciador señala un cambio conformacional en Cascade que recluta a Cas3 para la degradación del ADN.
Los sistemas de tipo II se basan en una única proteína multifuncional, Cas9 , para el paso de interferencia. [89] Cas9 requiere que tanto el crRNA como el tracrRNA funcionen y escinde el ADN utilizando sus dominios de endonucleasa duales HNH y RuvC / RNaseH. El emparejamiento de bases entre el PAM y el genoma del fago es necesario en los sistemas de tipo II. Sin embargo, el PAM se reconoce en la misma hebra que el crRNA (la hebra opuesta a los sistemas de tipo I).
Los sistemas de tipo III, como el tipo I, requieren seis o siete proteínas Cas que se unen a los crRNA. [140] [141] Los sistemas de tipo III analizados de S. solfataricus y P. furiosus se dirigen al ARNm de los fagos en lugar del genoma del ADN del fago, [81] [141] lo que puede hacer que estos sistemas sean excepcionalmente capaces de dirigirse a fagos basados en ARN genomas. [80] También se encontró que los sistemas de tipo III se dirigen al ADN además del ARN utilizando una proteína Cas diferente en el complejo, Cas10. [142] Se demostró que la escisión del ADN depende de la transcripción. [143]
El mecanismo para distinguir el ADN propio del extraño durante la interferencia está integrado en los ARNcr y, por lo tanto, es probable que sea común a los tres sistemas. A lo largo del proceso de maduración distintivo de cada tipo principal, todos los crRNA contienen una secuencia espaciadora y una parte de la repetición en uno o ambos extremos. Es la secuencia de repetición parcial la que evita que el sistema CRISPR-Cas se dirija al cromosoma, ya que el emparejamiento de bases más allá de la secuencia espaciadora se señala a sí mismo y evita la escisión del ADN. [144] enzimas CRISPR guiadas-RNA se clasifican como enzimas de restricción de tipo V .
Evolución [ editar ]
| Proteína asociada a CRISPR | |||||||||
|---|---|---|---|---|---|---|---|---|---|
estructura cristalina de una proteína asociada a crispr de Thermus thermophilus | |||||||||
| Identificadores | |||||||||
| Símbolo | CRISPR_assoc | ||||||||
| Pfam | PF08798 | ||||||||
| Clan pfam | CL0362 | ||||||||
| InterPro | IPR010179 | ||||||||
| CDD | cd09727 | ||||||||
| |||||||||
| Proteína Cas2 asociada a CRISPR | |||||||||
|---|---|---|---|---|---|---|---|---|---|
estructura cristalina de una proteína hipotética tt1823 de Thermus thermophilus | |||||||||
| Identificadores | |||||||||
| Símbolo | CRISPR_Cas2 | ||||||||
| Pfam | PF09827 | ||||||||
| InterPro | IPR019199 | ||||||||
| CDD | cd09638 | ||||||||
| |||||||||
| Proteína Cse1 asociada a CRISPR | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | CRISPR_Cse1 | ||||||||
| Pfam | PF09481 | ||||||||
| InterPro | IPR013381 | ||||||||
| CDD | cd09729 | ||||||||
| |||||||||
Se cree que los genes cas en los módulos adaptador y efector del sistema CRISPR-Cas han evolucionado a partir de dos módulos ancestrales diferentes. Un elemento similar a un transposón llamado casposón que codifica la integrasa similar a Cas1 y potencialmente otros componentes del módulo de adaptación se insertó junto al módulo efector ancestral, que probablemente funcionó como un sistema inmunológico innato independiente. [145] Los genes cas1 y cas2 altamente conservados del módulo adaptador evolucionaron a partir del módulo ancestral, mientras que una variedad de genes cas efectores de clase 1 evolucionaron a partir del módulo efector ancestral. [146] La evolución de estos diversos genes cas del módulo efector de clase 1 fue guiada por varios mecanismos, como los eventos de duplicación. [147]Por otro lado, cada tipo de módulo efector de clase 2 surgió a partir de inserciones independientes posteriores de elementos genéticos móviles. [148] Estos elementos genéticos móviles tomaron el lugar de los módulos efectores de genes múltiples para crear módulos efectores de un solo gen que producen proteínas grandes que realizan todas las tareas necesarias del módulo efector. [148] Las regiones espaciadoras de los sistemas CRISPR-Cas se toman directamente de elementos genéticos móviles extraños y, por lo tanto, su evolución a largo plazo es difícil de rastrear. [149] Se ha descubierto que la evolución no aleatoria de estas regiones espaciadoras depende en gran medida del medio ambiente y de los elementos genéticos móviles extraños que contiene. [150]
CRISPR / Cas puede inmunizar bacterias contra ciertos fagos y así detener la transmisión. Por esta razón, Koonin describió CRISPR / Cas como un mecanismo de herencia lamarckiano . [151] Sin embargo, esto fue discutido por un crítico que señaló: "Debemos recordar [a Lamarck] por el bien que contribuyó a la ciencia, no por cosas que se parecen a su teoría sólo superficialmente. De hecho, pensar en CRISPR y otros fenómenos como sólo lamarckianos oscurece la forma simple y elegante en que realmente funciona la evolución ". [152] Pero a medida que se han realizado estudios más recientes, se ha hecho evidente que las regiones espaciadoras adquiridas de los sistemas CRISPR-Cas son de hecho una forma de evolución lamarckiana porque son mutaciones genéticas que se adquieren y luego se transmiten. [153]Por otro lado, la evolución de la maquinaria del gen Cas que facilita el sistema evoluciona a través de la evolución darwiniana clásica. [153]
Coevolución [ editar ]
El análisis de las secuencias CRISPR reveló la coevolución de los genomas del huésped y del virus. [154] Las proteínas Cas9 están altamente enriquecidas en bacterias patógenas y comensales . La regulación génica mediada por CRISPR / Cas puede contribuir a la regulación de genes bacterianos endógenos, particularmente durante la interacción con huéspedes eucariotas. Por ejemplo, Francisella novicida usa un ARN único, pequeño, asociado a CRISPR / Cas (scaRNA) para reprimir una transcripción endógena que codifica una lipoproteína bacteriana que es crítica para que F. novicida amortigüe la respuesta del huésped y promueva la virulencia. [155]
El modelo básico de la evolución de CRISPR son los espaciadores recién incorporados que impulsan a los fagos a mutar sus genomas para evitar la respuesta inmune bacteriana, creando diversidad tanto en las poblaciones de fagos como de hospedadores. Para resistir una infección por fagos, la secuencia del espaciador CRISPR debe corresponder perfectamente a la secuencia del gen del fago diana. Los fagos pueden seguir infectando a sus huéspedes si se producen mutaciones puntuales en el espaciador. [144] Se requiere un rigor similar en PAM o la cepa bacteriana permanece sensible a los fagos. [117] [144]
Tarifas [ editar ]
Un estudio de 124 cepas de S. thermophilus mostró que el 26% de todos los espaciadores eran únicos y que diferentes loci CRISPR mostraban diferentes tasas de adquisición de espaciadores. [116] Algunos loci CRISPR evolucionan más rápidamente que otros, lo que permitió determinar las relaciones filogenéticas de las cepas. Un análisis genómico comparativo mostró que E. coli y S. enterica evolucionan mucho más lentamente que S. thermophilus . Las cepas de este último que divergieron hace 250 mil años todavía contenían el mismo complemento espaciador. [156]
El análisis metagenómico de dos biopelículas de drenaje ácido de minas mostró que uno de los CRISPR analizados contenía deleciones extensas y adiciones de espaciadores en comparación con el otro biofilm, lo que sugiere una mayor actividad / prevalencia de fagos en una comunidad que en la otra. [76] En la cavidad oral, un estudio temporal determinó que del 7 al 22% de los espaciadores se compartieron durante 17 meses dentro de un individuo, mientras que menos del 2% se compartieron entre los individuos. [125]
Desde el mismo entorno, se rastreó una única cepa utilizando cebadores de PCR específicos para su sistema CRISPR. Los resultados de nivel amplio de presencia / ausencia de espaciador mostraron una diversidad significativa. Sin embargo, este CRISPR agregó 3 espaciadores durante 17 meses, [125] lo que sugiere que incluso en un entorno con una diversidad CRISPR significativa, algunos loci evolucionan lentamente.
Los CRISPR se analizaron a partir de los metagenomas producidos para el proyecto del microbioma humano . [157] Aunque la mayoría eran específicos del sitio del cuerpo, algunos dentro de un sitio del cuerpo se comparten ampliamente entre las personas. Uno de estos loci se originó a partir de especies de estreptococos y contenía aproximadamente 15.000 espaciadores, el 50% de los cuales eran únicos. Al igual que en los estudios específicos de la cavidad bucal, algunos mostraron poca evolución con el tiempo. [157]
La evolución de CRISPR se estudió en quimiostatos utilizando S. thermophilus para examinar directamente las tasas de adquisición del espaciador. En una semana, las cepas de S. thermophilus adquirieron hasta tres espaciadores cuando se desafiaron con un solo fago. [158] Durante el mismo intervalo, el fago desarrolló polimorfismos de un solo nucleótido que se fijaron en la población, lo que sugiere que el direccionamiento había impedido la replicación del fago en ausencia de estas mutaciones. [158]
Otro experimento de S. thermophilus mostró que los fagos pueden infectar y replicarse en huéspedes que solo tienen un espaciador de dirección. Otro mostró que los huéspedes sensibles pueden existir en entornos con altos títulos de fagos. [159] Los estudios de quimiostato y de observación sugieren muchos matices de CRISPR y la (co) evolución de fagos.
Identificación [ editar ]
Los CRISPR se distribuyen ampliamente entre bacterias y arqueas [85] y muestran algunas similitudes de secuencia. [131] Su característica más notable son sus espaciadores repetidos y sus repeticiones directas. Esta característica hace que los CRISPR sean fácilmente identificables en secuencias largas de ADN, ya que el número de repeticiones disminuye la probabilidad de una coincidencia falsa positiva. [160]
El análisis de CRISPR en datos metagenómicos es más desafiante, ya que los loci CRISPR normalmente no se ensamblan, debido a su naturaleza repetitiva oa través de la variación de tensión, lo que confunde los algoritmos de ensamblaje. Cuando hay muchos genomas de referencia disponibles, se puede utilizar la reacción en cadena de la polimerasa (PCR) para amplificar matrices CRISPR y analizar el contenido del espaciador. [116] [125] [161] [162] [163] [164] Sin embargo, este enfoque proporciona información solo para CRISPR específicamente dirigidos y para organismos con suficiente representación en bases de datos públicas para diseñar una reacción en cadena de la polimerasa confiable(PCR) cebadores. Se pueden usar cebadores específicos de repetición degenerados para amplificar espaciadores CRISPR directamente de muestras ambientales; Los amplicones que contienen dos o tres espaciadores se pueden ensamblar computacionalmente para reconstruir matrices CRISPR largas. [164]
La alternativa es extraer y reconstruir matrices CRISPR a partir de datos metagenómicos de escopeta. Esto es computacionalmente más difícil, particularmente con tecnologías de secuenciación de segunda generación (por ejemplo, 454, Illumina), ya que las longitudes de lectura cortas evitan que aparezcan más de dos o tres unidades repetidas en una sola lectura. La identificación CRISPR en lecturas sin procesar se ha logrado mediante la identificación puramente de novo [165] o mediante el uso de secuencias repetidas directas en matrices CRISPR parcialmente ensambladas de contigs (segmentos de ADN superpuestos que juntos representan una región de consenso de ADN) [157] y secuencias repetidas directas de publicó genomas [166] como un gancho para identificar repeticiones directas en lecturas individuales.
Uso por fagos [ editar ]
Otra forma de que las bacterias se defiendan de la infección por fagos es tener islas cromosómicas . Un subtipo de islas cromosómicas llamado isla cromosómica inducible por fagos (PICI) se escinde de un cromosoma bacteriano tras la infección por fagos y puede inhibir la replicación del fago. [167]Los PICI son inducidos, escindidos, replicados y finalmente empaquetados en pequeñas cápsides por ciertos fagos estafilocócicos templados. Los PICI utilizan varios mecanismos para bloquear la reproducción de fagos. En el primer mecanismo, Ppi codificado por PICI bloquea diferencialmente la maduración del fago al unirse o interactuar específicamente con el fago TerS, por lo tanto bloquea la formación del complejo del fago TerS / TerL responsable del empaquetamiento del ADN del fago. En el segundo mecanismo, PICI CpmAB redirige la proteína morfogenética de la cápside del fago para hacer que el 95% de la cápside del tamaño de SaPI y el ADN del fago puedan empaquetar solo 1/3 de su genoma en esta pequeña cápside y, por lo tanto, convertirse en fagos no viables. [168]El tercer mecanismo involucra dos proteínas, PtiA y PtiB, que se dirigen a la LtrC, que es responsable de la producción de proteínas de virión y lisis. Este mecanismo de interferencia está modulado por una proteína moduladora, PtiM, que se une a una de las proteínas mediadoras de interferencia, PtiA, y por lo tanto logra el nivel de interferencia requerido. [169]
Un estudio mostró que el fago lítico ICP1, que se dirige específicamente al serogrupo O1 de Vibrio cholerae , ha adquirido un sistema CRISPR / Cas que se dirige a un elemento similar a PICI de V. cholera . El sistema tiene 2 loci CRISPR y 9 genes Cas. Parece ser homólogo al sistema IF que se encuentra en Yersinia pestis . Además, al igual que el sistema bacteriano CRISPR / Cas, ICP1 CRISPR / Cas puede adquirir nuevas secuencias, lo que permite que el fago y el huésped co-evolucionen. [170]
Se demostró que ciertos virus de arqueas llevan matrices mini-CRISPR que contienen uno o dos espaciadores. Se ha demostrado que los espaciadores dentro de las matrices CRISPR transmitidas por virus se dirigen a otros virus y plásmidos, lo que sugiere que las matrices mini-CRISPR representan un mecanismo de exclusión de superinfección heterotípica y participan en conflictos intervirales. [164]
Aplicaciones [ editar ]
Edición de genes CRISPR [ editar ]
La tecnología CRISPR se ha aplicado en las industrias alimentaria y agrícola para diseñar cultivos probióticos e inmunizar cultivos industriales (para yogur, por ejemplo) contra infecciones. También se utiliza en cultivos para mejorar el rendimiento, la tolerancia a la sequía y el valor nutricional. [171]
A finales de 2014 se habían publicado unos 1000 artículos de investigación que mencionaban CRISPR. [172] [173] La tecnología se había utilizado para inactivar funcionalmente genes en líneas celulares y células humanas, para estudiar Candida albicans , para modificar levaduras utilizadas para producir biocombustibles y para modificar genéticamente cepas de cultivos . [173] CRISPR también se puede utilizar para cambiar mosquitos para que no puedan transmitir enfermedades como la malaria. [174] Los enfoques basados en CRISPR que utilizan Cas12a se han utilizado recientemente en la modificación con éxito de un gran número de especies de plantas. [175]
En julio de 2019, CRISPR se utilizó para tratar experimentalmente a un paciente con un trastorno genético. La paciente era una mujer de 34 años con anemia drepanocítica . [176]
En febrero de 2020, se avanzó en los tratamientos contra el VIH con un 60-80% del ADN eliminado en ratones y algunos estaban completamente libres del virus después de ediciones que involucraron CRISPR y LASER ART . [177]
En marzo de 2020, se inyectó virus modificado con CRISPR en el ojo de un paciente en un intento de tratar la amaurosis congénita de Leber . [178]
En el futuro, la edición de genes CRISPR podría potencialmente usarse para crear nuevas especies o revivir especies extintas de otras estrechamente relacionadas. [179]
Las reevaluaciones basadas en CRISPR de las afirmaciones de las relaciones entre genes y enfermedades han llevado al descubrimiento de anomalías potencialmente importantes. [180]
CRISPR como herramienta de diagnóstico [ editar ]
Las nucleasas asociadas a CRISPR han demostrado ser útiles como herramienta para las pruebas moleculares debido a su capacidad para apuntar específicamente a secuencias de ácido nucleico en un fondo elevado de secuencias no diana. En 2016, la nucleasa Cas9 se utilizó para agotar las secuencias de nucleótidos no deseadas en bibliotecas de secuenciación de próxima generación, mientras que solo requería 250 picogramos de entrada de ARN inicial. [181] A partir de 2017, las nucleasas asociadas a CRISPR también se utilizaron para pruebas de diagnóstico directo de ácidos nucleicos, hasta la sensibilidad de una sola molécula. [182] [183]
Al acoplar los diagnósticos basados en CRISPR a procesos enzimáticos adicionales, es posible la detección de moléculas más allá de los ácidos nucleicos. Un ejemplo de tecnología acoplada es la elaboración de perfiles de transcripción in vitro (SPRINT) basada en SHERLOCK. SPRINT se puede utilizar para detectar una variedad de sustancias, como metabolitos en muestras de pacientes o contaminantes en muestras ambientales, con alto rendimiento o con dispositivos portátiles en el punto de atención. [184] Las plataformas CRISPR / Cas también se están explorando para la detección [185] [186] [187] [188] [189] y la inactivación del nuevo coronavirus, SARS-CoV-2. [190]
Ver también [ editar ]
- Anti-CRISPR
- Herramientas CRISPR / Cas
- Edición de genes CRISPR
- El diario CRISPR
- DRACO
- Nocaut genético
- Genética
- Pantallas de eliminación de CRISPR-Cas9 en todo el genoma
- Glosario de genética
- Human Nature (documental de 2019)
- Edición principal
- ARNi
- ARNip
- Ensayo de nucleasa de topógrafo
- Biología sintética
- Dedo de zinc
Notas [ editar ]
- ^ El subtipo IG se conocía anteriormente como subtipo IU . [77]
- ^ El subtipo VK se conocía anteriormente como subtipo VU 5. [87]
Referencias [ editar ]
- ^ Mulepati S, Héroux A, Bailey S (2014). "Estructura cristalina de un complejo de vigilancia guiado por CRISPR RNA unido a un objetivo de ssDNA" . Ciencia . 345 (6203): 1479–1484. Código Bibliográfico : 2014Sci ... 345.1479M . doi : 10.1126 / science.1256996 . PMC 4427192 . PMID 25123481 .
- ↑ a b Barrangou R (2015). "Los roles de los sistemas CRISPR-Cas en la inmunidad adaptativa y más allá". Opinión actual en inmunología . 32 : 36–41. doi : 10.1016 / j.coi.2014.12.008 . PMID 25574773 .
- ^ Redman M, King A, Watson C, King D (agosto de 2016). "¿Qué es CRISPR / Cas9?" . Archivos de enfermedades en la infancia. Edición Educación y Práctica . 101 (4): 213–215. doi : 10.1136 / archdischild-2016-310459 . PMC 4975809 . PMID 27059283 .
- ^ a b Barrangou R , Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, et al. (Marzo de 2007). "CRISPR proporciona resistencia adquirida frente a virus en procariotas". Ciencia . 315 (5819): 1709–1712. Código Bibliográfico : 2007Sci ... 315.1709B . doi : 10.1126 / science.1138140 . hdl : 20.500.11794 / 38902 . PMID 17379808 . S2CID 3888761 . ( se requiere registro )
- ↑ a b Marraffini LA, Sontheimer EJ (diciembre de 2008). "La interferencia de CRISPR limita la transferencia horizontal de genes en estafilococos al dirigirse al ADN" . Ciencia . 322 (5909): 1843–1845. Código Bibliográfico : 2008Sci ... 322.1843M . doi : 10.1126 / science.1165771 . PMC 2695655 . PMID 19095942 .
- ^ Hille F, Richter H, Wong SP, Bratovič M, Ressel S, Charpentier E (marzo de 2018). "La biología de CRISPR-Cas: hacia adelante y hacia atrás". Celular . 172 (6): 1239-1259. doi : 10.1016 / j.cell.2017.11.032 . hdl : 21.11116 / 0000-0003-FC0D-4 . PMID 29522745 . S2CID 3777503 .
- ^ Horvath P, Barrangou R (enero de 2010). "CRISPR / Cas, el sistema inmunológico de bacterias y arqueas". Ciencia . 327 (5962): 167-170. Código Bibliográfico : 2010Sci ... 327..167H . doi : 10.1126 / science.1179555 . PMID 20056882 . S2CID 17960960 .
- ↑ Zhang F, Wen Y, Guo X (2014). "CRISPR / Cas9 para la edición del genoma: avances, implicaciones y desafíos" . Genética molecular humana . 23 (R1): R40–6. doi : 10.1093 / hmg / ddu125 . PMID 24651067 .
- ^ CRISPR-CAS9, TALENS y ZFNS: la batalla en la edición de genes https://www.ptglab.com/news/blog/crispr-cas9-talens-and-zfns-the-battle-in-gene-editing/
- ^ a b c d e Hsu PD, Lander ES, Zhang F (junio de 2014). "Desarrollo y aplicaciones de CRISPR-Cas9 para la ingeniería del genoma" . Celular . 157 (6): 1262-1278. doi : 10.1016 / j.cell.2014.05.010 . PMC 4343198 . PMID 24906146 .
- ^ "Comunicado de prensa: el Premio Nobel de Química 2020" . Fundación Nobel . Consultado el 7 de octubre de 2020 .
- ^ Wu KJ, Peltier E (7 de octubre de 2020). "Premio Nobel de Química otorgado a 2 científicos por su trabajo en la edición del genoma: Emmanuelle Charpentier y Jennifer A. Doudna desarrollaron la herramienta Crispr, que puede alterar el ADN de animales, plantas y microorganismos con alta precisión" . The New York Times . Consultado el 7 de octubre de 2020 .
- ↑ a b Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A (diciembre de 1987). "Secuencia de nucleótidos del gen iap, responsable de la conversión de isoenzimas de fosfatasa alcalina en Escherichia coli, e identificación del producto génico" . Revista de bacteriología . 169 (12): 5429–5433. doi : 10.1128 / jb.169.12.5429-5433.1987 . PMC 213968 . PMID 3316184 .
- ^ van Soolingen D, de Haas PE, Hermans PW, Groenen PM, van Embden JD (agosto de 1993). "Comparación de varios elementos repetitivos del ADN como marcadores genéticos para la diferenciación de cepas y epidemiología de Mycobacterium tuberculosis" . Revista de microbiología clínica . 31 (8): 1987–1995. doi : 10.1128 / JCM.31.8.1987-1995.1993 . PMC 265684 . PMID 7690367 .
- ^ Groenen PM, Bunschoten AE, van Soolingen D, van Embden JD (diciembre de 1993). "Naturaleza del polimorfismo de ADN en el grupo de repetición directa de Mycobacterium tuberculosis; aplicación para la diferenciación de cepas mediante un nuevo método de tipificación". Microbiología molecular . 10 (5): 1057–1065. doi : 10.1111 / j.1365-2958.1993.tb00976.x . PMID 7934856 . S2CID 25304723 .
- ↑ a b c Mojica FJ, Montoliu L (2016). "Sobre el origen de la tecnología CRISPR-Cas: de procariotas a mamíferos". Tendencias en microbiología . 24 (10): 811–820. doi : 10.1016 / j.tim.2016.06.005 . PMID 27401123 .
- ↑ a b Mojica FJ, Rodriguez-Valera F (2016). "El descubrimiento de CRISPR en arqueas y bacterias" (PDF) . La revista FEBS . 283 (17): 3162–3169. doi : 10.1111 / febs.13766 . hdl : 10045/57676 . PMID 27234458 . S2CID 42827598 .
- ^ Mojica FJ, Díez-Villaseñor C, Soria E, Juez G (abril de 2000). "Importancia biológica de una familia de repeticiones espaciadas regularmente en los genomas de Archaea, bacterias y mitocondrias" . Microbiología molecular . 36 (1): 244–246. doi : 10.1046 / j.1365-2958.2000.01838.x . PMID 10760181 .
- ^ Barrangou R , van der Oost J (2013). Sistemas CRISPR-Cas: inmunidad adaptativa mediada por ARN en bacterias y arqueas . Heidelberg: Springer. pag. 6. ISBN 978-3-642-34656-9.
- ^ Tang TH, Bachellerie JP, Rozhdestvensky T, Bortolin ML, Huber H, Drungowski M; et al. (2002). "Identificación de 86 candidatos para pequeños ARN no mensajeros del archaeon Archaeoglobus fulgidus" . Proc Natl Acad Sci USA . 99 (11): 7536–41. Código bibliográfico : 2002PNAS ... 99.7536T . doi : 10.1073 / pnas.112047299 . PMC 124276 . PMID 12032318 . CS1 maint: multiple names: authors list (link)
- ^ Charpentier E, Richter H, van der Oost J, White MF (mayo de 2015). "Vías de biogénesis de guías de ARN en inmunidad adaptativa CRISPR-Cas de arqueas y bacterias" . Reseñas de Microbiología FEMS . 39 (3): 428–441. doi : 10.1093 / femsre / fuv023 . PMC 5965381 . PMID 25994611 .
- ^ Romero DA, Magill D, Millen A, Horvath P, Fremaux C (noviembre de 2020). "Interacciones fago-huésped de lactococos y estreptococos lácteos: una perspectiva industrial en un paisaje de fagos en evolución" . Reseñas de Microbiología FEMS . 44 (6): 909–932. doi : 10.1093 / femsre / fuaa048 . PMID 33016324 .
- ^ Molteni M, Huckins G (1 de agosto de 2020). "La guía WIRED de Crispr" . Conde Nast. Revista cableada.
- ^ Jansen R, Embden JD, Gaastra W, Schouls LM (marzo de 2002). "Identificación de genes que se asocian con repeticiones de ADN en procariotas". Microbiología molecular . 43 (6): 1565-1575. doi : 10.1046 / j.1365-2958.2002.02839.x . PMID 11952905 . S2CID 23196085 .
- ↑ a b Horvath P, Barrangou R (enero de 2010). "CRISPR / Cas, el sistema inmunológico de bacterias y arqueas". Ciencia . 327 (5962): 167-170. Código Bibliográfico : 2010Sci ... 327..167H . doi : 10.1126 / Science.1179555 . PMID 20056882 . S2CID 17960960 .
- ↑ a b Marraffini LA, Sontheimer EJ (marzo de 2010). "Interferencia CRISPR: inmunidad adaptativa dirigida por ARN en bacterias y arqueas" . Nature Reviews Genética . 11 (3): 181-190. doi : 10.1038 / nrg2749 . PMC 2928866 . PMID 20125085 .
- ^ Grissa I, Vergnaud G, Pourcel C (mayo de 2007). "La base de datos CRISPRdb y herramientas para visualizar CRISPRs y generar diccionarios de espaciadores y repeticiones" . BMC Bioinformática . 8 : 172. doi : 10.1186 / 1471-2105-8-172 . PMC 1892036 . PMID 17521438 .
- ↑ a b Pourcel C, Salvignol G, Vergnaud G (marzo de 2005). "Los elementos CRISPR en Yersinia pestis adquieren nuevas repeticiones mediante la captación preferencial del ADN del bacteriófago y proporcionan herramientas adicionales para estudios evolutivos". Microbiología . 151 (Pt 3): 653–663. doi : 10.1099 / mic.0.27437-0 . PMID 15758212 .
- ↑ a b Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (febrero de 2005). "Las secuencias que intervienen de repeticiones procariotas espaciadas regularmente derivan de elementos genéticos extraños". Revista de evolución molecular . 60 (2): 174-182. Código bibliográfico : 2005JMolE..60..174M . doi : 10.1007 / s00239-004-0046-3 . PMID 15791728 . S2CID 27481111 .
- ↑ a b Bolotin A, Quinquis B, Sorokin A, Ehrlich SD (agosto de 2005). "Las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) tienen espaciadores de origen extracromosómico" . Microbiología . 151 (Parte 8): 2551-2561. doi : 10.1099 / mic.0.28048-0 . PMID 16079334 .
- ^ Morange M (junio de 2015). "Lo que nos cuenta la historia XXXVII. CRISPR-Cas: El descubrimiento de un sistema inmunológico en procariotas" . Revista de Biociencias . 40 (2): 221-223. doi : 10.1007 / s12038-015-9532-6 . PMID 25963251 .
- ^ Lander ES (enero de 2016). "Los Héroes de CRISPR" . Celular . 164 (1–2): 18–28. doi : 10.1016 / j.cell.2015.12.041 . PMID 26771483 .
- ^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (marzo de 2006). "Un sistema inmune basado en interferencia de ARN putativo en procariotas: análisis computacional de la maquinaria enzimática predicha, analogías funcionales con ARNi eucariota y mecanismos de acción hipotéticos" . Biology Direct . 1 : 7. doi : 10.1186 / 1745-6150-1-7 . PMC 1462988 . PMID 16545108 .
- ↑ a b c Marraffini LA (octubre de 2015). "Inmunidad CRISPR-Cas en procariotas". Naturaleza . 526 (7571): 55–61. Código Bibcode : 2015Natur.526 ... 55M . doi : 10.1038 / nature15386 . PMID 26432244 . S2CID 3718361 .
- ^ Pennisi E (agosto de 2013). "La locura de CRISPR". Enfoque de noticias. Ciencia . 341 (6148): 833–836. Código bibliográfico : 2013Sci ... 341..833P . doi : 10.1126 / science.341.6148.833 . PMID 23970676 .
- ^ Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, Dickman MJ, Makarova KS, Koonin EV , van der Oost J (agosto de 2008). "Pequeños ARN CRISPR guían la defensa antiviral en procariotas" . Ciencia . 321 (5891): 960–964. Código Bibliográfico : 2008Sci ... 321..960B . doi : 10.1126 / science.1159689 . PMC 5898235 . PMID 18703739 .
- ^ a b Garneau JE, Dupuis MÈ, Villion M, Romero DA, Barrangou R , Boyaval P, et al. (Noviembre de 2010). "El sistema inmunológico bacteriano CRISPR / Cas escinde el ADN plasmídico y bacteriófago". Naturaleza . 468 (7320): 67–71. Bibcode : 2010Natur.468 ... 67G . CiteSeerX 10.1.1.451.9645 . doi : 10.1038 / nature09523 . PMID 21048762 . S2CID 205222849 .
- ^ a b Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E (marzo de 2011). "Maduración del ARN CRISPR por ARN pequeño transcodificado y factor de acogida ARNasa III" . Naturaleza . 471 (7340): 602–607. Código Bibliográfico : 2011Natur.471..602D . doi : 10.1038 / nature09886 . PMC 3070239 . PMID 21455174 .
- ^ Barrangou R (noviembre de 2015). "Diversidad de sistemas inmunes CRISPR-Cas y máquinas moleculares" . Biología del genoma . 16 : 247. doi : 10.1186 / s13059-015-0816-9 . PMC 4638107 . PMID 26549499 .
- ^ Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA , Charpentier E (agosto de 2012). "Una endonucleasa de ADN guiada por ARN dual programable en inmunidad bacteriana adaptativa" . Ciencia . 337 (6096): 816–821. Código bibliográfico : 2012Sci ... 337..816J . doi : 10.1126 / science.1225829 . PMC 6286148 . PMID 22745249 .
- ^ Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F (febrero de 2013). "Ingeniería del genoma multiplexado mediante sistemas CRISPR / Cas" . Ciencia . 339 (6121): 819–823. Código bibliográfico : 2013Sci ... 339..819C . doi : 10.1126 / science.1231143 . PMC 3795411 . PMID 23287718 .
- ^ Mali P, Yang L, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM (febrero de 2013). "Ingeniería del genoma humano guiada por ARN a través de Cas9" . Ciencia . 339 (6121): 823–826. Código bibliográfico : 2013Sci ... 339..823M . doi : 10.1126 / science.1232033 . PMC 3712628 . PMID 23287722 .
- ^ DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM (abril de 2013). "Ingeniería del genoma en Saccharomyces cerevisiae utilizando sistemas CRISPR-Cas" . Investigación de ácidos nucleicos . 41 (7): 4336–4343. doi : 10.1093 / nar / gkt135 . PMC 3627607 . PMID 23460208 .
- ^ Zhang GC, Kong II, Kim H, Liu JJ, Cate JH, Jin YS (diciembre de 2014). "Construcción de un mutante auxotrófico cuádruple de una cepa de saccharomyces cerevisiae poliploide industrial mediante el uso de nucleasa Cas9 guiada por ARN" . Microbiología aplicada y ambiental . 80 (24): 7694–7701. doi : 10.1128 / AEM.02310-14 . PMC 4249234 . PMID 25281382 .
- ^ Liu JJ, Kong II, Zhang GC, Jayakody LN, Kim H, Xia PF, Kwak S, Sung BH, Sohn JH, Walukiewicz HE, Rao CV, Jin YS (abril de 2016). "Ingeniería metabólica del probiótico Saccharomyces boulardii " . Microbiología aplicada y ambiental . 82 (8): 2280–2287. doi : 10.1128 / AEM.00057-16 . PMC 4959471 . PMID 26850302 .
- ^ Vyas VK, Barrasa MI, Fink GR (2015). "El sistema CRISPR de Candida albicans permite la ingeniería genética de genes esenciales y familias de genes" . Avances científicos . 1 (3): e1500248. Código bibliográfico : 2015SciA .... 1E0248V . doi : 10.1126 / sciadv.1500248 . PMC 4428347 . PMID 25977940 .
- ↑ Ng H, Dean N (2017). " Candida albicans por mayor expresión de ARN de guía única" . mSphere . 2 (2): e00385–16. doi : 10.1128 / mSphere.00385-16 . PMC 5397569 . PMID 28435892 .
- ^ Hwang WY, Fu Y, Reyon D, Maeder ML, Tsai SQ, Sander JD, Peterson RT, Yeh JR, Joung JK (marzo de 2013). "Edición eficiente del genoma en pez cebra utilizando un sistema CRISPR-Cas" . Biotecnología de la naturaleza . 31 (3): 227–229. doi : 10.1038 / nbt.2501 . PMC 3686313 . PMID 23360964 .
- ^ Gratz SJ, Cummings AM, Nguyen JN, Hamm DC, Donohue LK, Harrison MM, Wildonger J, O'Connor-Giles KM (agosto de 2013). "Ingeniería del genoma de Drosophila con la nucleasa Cas9 guiada por ARN CRISPR" . Genética . 194 (4): 1029–1035. doi : 10.1534 / genetics.113.152710 . PMC 3730909 . PMID 23709638 .
- ^ Bassett AR, Tibbit C, Ponting CP, Liu JL (julio de 2013). "Mutagénesis dirigida altamente eficiente de Drosophila con el sistema CRISPR / Cas9" . Informes de celda . 4 (1): 220–228. doi : 10.1016 / j.celrep.2013.06.020 . PMC 3714591 . PMID 23827738 .
- ^ Yan H, Opachaloemphan C, Mancini G, Yang H, Gallitto M, Mlejnek J, Leibholz A, Haight K, Ghaninia M, Huo L, Perry M, Slone J, Zhou X, Traficante M, Penick CA, Dolezal K, Gokhale K, Stevens K, Fetter-Pruneda I, Bonasio R, Zwiebel LJ, Berger SL, Liebig J, Reinberg D, Desplan C (agosto de 2017). "Una mutación orco diseñada produce un comportamiento social aberrante y un desarrollo neuronal defectuoso en las hormigas" . Celular . 170 (4): 736–747.e9. doi : 10.1016 / j.cell.2017.06.051 . PMC 5587193 . PMID 28802043 .
- ^ Trible W, Olivos-Cisneros L, McKenzie SK, Saragosti J, Chang NC, Matthews BJ, Oxley PR, Kronauer DJ (agosto de 2017). "Orco mutagénesis causa pérdida de glomérulos del lóbulo de la antena y comportamiento social deteriorado en hormigas" . Celular . 170 (4): 727–735.e10. doi : 10.1016 / j.cell.2017.07.001 . PMC 5556950 . PMID 28802042 .
- ^ Kistler KE, Vosshall LB, Matthews BJ (abril de 2015). "Ingeniería del genoma con CRISPR-Cas9 en el mosquito Aedes aegypti" . Informes de celda . 11 (1): 51–60. doi : 10.1016 / j.celrep.2015.03.009 . PMC 4394034 . PMID 25818303 .
- ^ Friedland AE, Tzur YB, Esvelt KM, Colaiácovo MP, Church GM, Calarco JA (agosto de 2013). "Edición hereditaria del genoma en C. elegans a través de un sistema CRISPR-Cas9" . Métodos de la naturaleza . 10 (8): 741–743. doi : 10.1038 / nmeth.2532 . PMC 3822328 . PMID 23817069 .
- ^ Jiang W, Zhou H, Bi H, Fromm M, Yang B, Weeks DP (noviembre de 2013). "Demostración de la modificación de genes dirigidos mediada por CRISPR / Cas9 / sgRNA en Arabidopsis, tabaco, sorgo y arroz" . Investigación de ácidos nucleicos . 41 (20): e188. doi : 10.1093 / nar / gkt780 . PMC 3814374 . PMID 23999092 .
- ^ Wang H, Yang H, Shivalila CS, Dawlaty MM, Cheng AW, Zhang F , Jaenisch R (mayo de 2013). "Generación de un paso de ratones portadores de mutaciones en múltiples genes por ingeniería genómica mediada por CRISPR / Cas" . Celular . 153 (4): 910–918. doi : 10.1016 / j.cell.2013.04.025 . PMC 3969854 . PMID 23643243 .
- ^ Soni D, Wang DM, Regmi SC, Mittal M, Vogel SM, Schlüter D, Tiruppathi C (mayo de 2018). "La función deubiquitinasa de A20 mantiene y repara la barrera endotelial después de una lesión vascular pulmonar" . Descubrimiento de la muerte celular . 4 (60): 60. doi : 10.1038 / s41420-018-0056-3 . PMC 5955943 . PMID 29796309 .
- ^ Guo X, Li XJ (julio de 2015). "Edición dirigida del genoma en embriones de primates" . Investigación celular . 25 (7): 767–768. doi : 10.1038 / cr.2015.64 . PMC 4493275 . PMID 26032266 .
- ^ Baltimore D, Berg P, Botchan M, Carroll D, Charo RA, Church G, Corn JE, Daley GQ, Doudna JA , Fenner M, Greely HT, Jinek M, Martin GS, Penhoet E, Puck J, Sternberg SH, Weissman JS, Yamamoto KR (abril de 2015). "Biotecnología. Un camino prudente para la ingeniería genómica y la modificación de genes de la línea germinal" . Ciencia . 348 (6230): 36–38. Código bibliográfico : 2015Sci ... 348 ... 36B . doi : 10.1126 / science.aab1028 . PMC 4394183 . PMID 25791083 .
- ^ Larson MH, Gilbert LA, Wang X, Lim WA, Weissman JS, Qi LS (noviembre de 2013). "Interferencia CRISPR (CRISPRi) para el control específico de secuencia de la expresión génica" . Protocolos de la naturaleza . 8 (11): 2180–2196. doi : 10.1038 / nprot.2013.132 . PMC 3922765 . PMID 24136345 .
- ^ Liang P, Xu Y, Zhang X, Ding C, Huang R, Zhang Z, et al. (Mayo de 2015). "Edición de genes mediada por CRISPR / Cas9 en cigotos tripronucleares humanos" . Proteínas y células . 6 (5): 363–372. doi : 10.1007 / s13238-015-0153-5 . PMC 4417674 . PMID 25894090 .
- ^ Yan MY, Yan HQ, Ren GX, Zhao JP, Guo XP, Sun YC (septiembre de 2017). "Recombineering asistido por CRISPR-Cas12a en bacterias" . Microbiología aplicada y ambiental . 83 (17). doi : 10.1128 / AEM.00947-17 . PMC 5561284 . PMID 28646112 .
- ^ Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F (octubre de 2015). "Cpf1 es una única endonucleasa guiada por ARN de un sistema CRISPR-Cas de clase 2" . Celular . 163 (3): 759–771. doi : 10.1016 / j.cell.2015.09.038 . PMC 4638220 . PMID 26422227 .
- ^ Fonfara I, Richter H, Bratovič M, Le Rhun A, Charpentier E (abril de 2016). "La enzima Cpf1 de escisión de ADN asociada a CRISPR también procesa el ARN de CRISPR precursor". Naturaleza . 532 (7600): 517–521. Código Bib : 2016Natur.532..517F . doi : 10.1038 / nature17945 . PMID 27096362 . S2CID 2271552 .
- ^ Kim H, Kim ST, Ryu J, Kang BC, Kim JS y Kim SG (febrero de 2017). "Edición del genoma vegetal libre de ADN mediada por CRISPR / Cpf1" . Comunicaciones de la naturaleza . 8 (14406): 14406. Bibcode : 2017NatCo ... 814406K . doi : 10.1038 / ncomms14406 . PMC 5316869 . PMID 28205546 .
- ^ "Nucleasa Cpf1" . abmgood.com . Consultado el 14 de diciembre de 2017 .
- ^ Chen JS, Ma E, Harrington LB, Da Costa M, Tian X, Palefsky JM, Doudna JA (abril de 2018). "La unión de la diana CRISPR-Cas12a desencadena una actividad indiscriminada de ADNasa monocatenaria" . Ciencia . 360 (6387): 436–439. doi : 10.1126 / science.aar6245 . PMID 29449511 .
- ^ Broughton JP, Deng X, Yu G, Fasching CL, Servellita V, Singh J, et al. (Julio de 2020). "Detección de SARS-CoV-2 basada en CRISPR-Cas12" . Biotecnología de la naturaleza . 38 (7): 870–874. doi : 10.1038 / s41587-020-0513-4 . PMID 32300245 .
- ^ Nguyen LT, Smith BM, Jain PK (septiembre de 2020). "La mejora de la actividad trans-escisión de Cas12a con crRNA modificado permite la detección de ácido nucleico amplificado" . Comunicaciones de la naturaleza . 11 (1): 4906. doi : 10.1038 / s41467-020-18615-1 . PMID 32999292 .
- ^ Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DB, et al. (Agosto de 2016). "C2c2 es un efector CRISPR dirigido a ARN guiado por ARN programable de un solo componente" . Ciencia . 353 (6299): aaf5573. doi : 10.1126 / science.aaf5573 . PMC 5127784 . PMID 27256883 .
- ^ Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, et al. (Abril de 2017). "Detección de ácidos nucleicos con CRISPR-Cas13a / C2c2" . Ciencia . 356 (6336): 438–442. Código bibliográfico : 2017Sci ... 356..438G . doi : 10.1126 / science.aam9321 . PMC 5526198 . PMID 28408723 .
- ^ Gootenberg JS, Abudayyeh OO, Kellner MJ, Joung J, Collins JJ, Zhang F (abril de 2018). "Plataforma de detección de ácidos nucleicos multiplexada y portátil con Cas13, Cas12a y Csm6" . Ciencia . 360 (6387): 439–444. Código bibliográfico : 2018Sci ... 360..439G . doi : 10.1126 / science.aaq0179 . PMC 5961727 . PMID 29449508 .
- ^ Iwasaki RS, Batey RT (septiembre de 2020). "SPRINT: una plataforma basada en Cas13a para la detección de moléculas pequeñas" . Investigación de ácidos nucleicos . 48 (17): e101. doi : 10.1093 / nar / gkaa673 . PMC 7515716 . PMID 32797156 .
- ^ Hille F, Charpentier E (noviembre de 2016). "CRISPR-Cas: biología, mecanismos y relevancia" . Transacciones filosóficas de la Royal Society de Londres. Serie B, Ciencias Biológicas . 371 (1707): 20150496. doi : 10.1098 / rstb.2015.0496 . PMC 5052741 . PMID 27672148 .
- ↑ a b c Barrangou R , Marraffini LA (abril de 2014). "Sistemas CRISPR-Cas: actualización de procariotas a inmunidad adaptativa" . Célula molecular . 54 (2): 234–244. doi : 10.1016 / j.molcel.2014.03.011 . PMC 4025954 . PMID 24766887 .
- ^ a b c Tyson GW, Banfield JF (enero de 2008). "CRISPRs de rápida evolución implicados en la resistencia adquirida de microorganismos a virus". Microbiología ambiental . 10 (1): 200–207. doi : 10.1111 / j.1462-2920.2007.01444.x . PMID 17894817 .
- ^ a b c d e f g h i Makarova KS, Wolf YI, Alkhnbashi OS, Costa F, Shah SA, Saunders SJ, et al. (Noviembre de 2015). "Una clasificación evolutiva actualizada de los sistemas CRISPR-Cas" . Reseñas de la naturaleza. Microbiología . 13 (11): 722–736. doi : 10.1038 / nrmicro3569 . PMC 5426118 . PMID 26411297 .
- ^ a b c Wright AV, Nuñez JK, Doudna JA (enero de 2016). "Biología y aplicaciones de sistemas CRISPR: aprovechar la caja de herramientas de la naturaleza para la ingeniería del genoma" . Celular . 164 (1–2): 29–44. doi : 10.1016 / j.cell.2015.12.035 . PMID 26771484 .
- ^ Westra ER, Dowling AJ, Broniewski JM, van Houte S (noviembre de 2016). "Evolución y Ecología de CRISPR" . Revisión anual de ecología, evolución y sistemática . 47 (1): 307–331. doi : 10.1146 / annurev-ecolsys-121415-032428 .
- ↑ a b c Wiedenheft B, Sternberg SH, Doudna JA (febrero de 2012). "Sistemas de silenciamiento genético guiados por ARN en bacterias y arqueas". Naturaleza . 482 (7385): 331–338. Código bibliográfico : 2012Natur.482..331W . doi : 10.1038 / nature10886 . PMID 22337052 . S2CID 205227944 .
- ^ a b Deng L, Garrett RA, Shah SA, Peng X, She Q (marzo de 2013). "Un mecanismo de interferencia novedoso por un módulo tipo IIIB CRISPR-Cmr en Sulfolobus" . Microbiología molecular . 87 (5): 1088–1099. doi : 10.1111 / mmi.12152 . PMID 23320564 .
- ^ Sinkunas T, Gasiunas G, Fremaux C, Barrangou R , Horvath P, Siksnys V (abril de 2011). "Cas3 es una ADN nucleasa monocatenaria y helicasa dependiente de ATP en el sistema inmunológico CRISPR / Cas" . El diario EMBO . 30 (7): 1335-1342. doi : 10.1038 / emboj.2011.41 . PMC 3094125 . PMID 21343909 .
- ^ Huo Y, Nam KH, Ding F, Lee H, Wu L, Xiao Y, Farchione MD, Zhou S, Rajashankar K, Kurinov I, Zhang R, Ke A (septiembre de 2014). "Las estructuras de CRISPR Cas3 ofrecen conocimientos mecánicos sobre el desenrollamiento y degradación del ADN activado en cascada" . Naturaleza Biología Molecular y Estructural . 21 (9): 771–777. doi : 10.1038 / nsmb.2875 . PMC 4156918 . PMID 25132177 .
- ^ Brendel J, Stoll B, Lange SJ, Sharma K, Lenz C, Stachler AE, et al. (Marzo del 2014). "Se requiere un complejo de proteínas Cas 5, 6 y 7 para la biogénesis y la estabilidad de las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (crispr) derivadas de rnas (crrnas) en Haloferax volcanii" . La Revista de Química Biológica . 289 (10): 7164–77. doi : 10.1074 / jbc.M113.508184 . PMC 3945376 . PMID 24459147 .
- ↑ a b Chylinski K, Makarova KS, Charpentier E, Koonin EV (junio de 2014). "Clasificación y evolución de sistemas CRISPR-Cas tipo II" . Investigación de ácidos nucleicos . 42 (10): 6091–6105. doi : 10.1093 / nar / gku241 . PMC 4041416 . PMID 24728998 .
- ↑ a b Makarova KS, Aravind L, Wolf YI, Koonin EV (julio de 2011). "Unificación de familias de proteínas Cas y un escenario simple para el origen y evolución de los sistemas CRISPR-Cas" . Biology Direct . 6 : 38. doi : 10.1186 / 1745-6150-6-38 . PMC 3150331 . PMID 21756346 .
- ^ a b c d e f g h i j k l m n o p q r s t u v w Makarova KS, Wolf YI, Iranzo J, Shmakov SA, Alkhnbashi OS, Brouns SJJ, Charpentier E, Cheng D, Haft DH , Horvath P, Moineau S, Mojica FJM, Scott D, Shah SA, Siksnys V, Charranes MP, Venclovas Č, Blanco MF, Yakunin AF, Yan W, Zhang F, Garrett RA, Backofen R, van der Oost J, Barrangou R , Koonin EV. (2019). "Clasificación evolutiva de los sistemas CRISPR-Cas: una explosión de clase 2 y variantes derivadas" . Nature Reviews Microbiología . 18(2): 67–83. doi : 10.1038 / s41579-019-0299-x . hdl : 10045/102627 . PMID 31857715 . S2CID 209420490 .CS1 maint: uses authors parameter (link)
- ↑ a b Mogila I, Kazlauskiene M, Valinskyte S, Tamulaitiene G, Tamulaitis G, Siksnys V (marzo de 2019). "Disección genética del complejo Csm del sistema CRISPR-Cas tipo III-A revela roles de subunidades individuales" . Informes de celda . 26 (10): 2753–2765.e4. doi : 10.1016 / j.celrep.2019.02.029 . PMID 30840895 .
- ↑ a b c Gasiunas G, Barrangou R , Horvath P, Siksnys V (septiembre de 2012). "El complejo de ribonucleoproteína Cas9-crRNA media la escisión del ADN específico para la inmunidad adaptativa en bacterias" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (39): E2579-2586. Código bibliográfico : 2012PNAS..109E2579G . doi : 10.1073 / pnas.1208507109 . PMC 3465414 . PMID 22949671 .
- ^ Heler R, Samai P, Modell JW, Weiner C, Goldberg GW, Bikard D, Marraffini LA (marzo de 2015). "Cas9 especifica objetivos virales funcionales durante la adaptación CRISPR-Cas" . Naturaleza . 519 (7542): 199–202. Código bibliográfico : 2015Natur.519..199H . doi : 10.1038 / nature14245 . PMC 4385744 . PMID 25707807 .
- ^ Nam KH, Kurinov I, Ke A (septiembre de 2011). "La estructura cristalina de la proteína Csn2 asociada a repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) reveló una actividad de unión al ADN bicatenario dependiente de Ca2 +" . La Revista de Química Biológica . 286 (35): 30759–30768. doi : 10.1074 / jbc.M111.256263 . PMC 3162437 . PMID 21697083 .
- ^ Lee H, Dhingra Y, Sashital DG (abril de 2019). "El complejo Cas4-Cas1-Cas2 media el procesamiento preciso del prespacer durante la adaptación CRISPR" . eLife . 8 . doi : 10.7554 / eLife.44248 . PMC 6519985 . PMID 31021314 .
- ^ Chylinski K, Le Rhun A, Charpentier E (mayo de 2013). "Las familias tracrRNA y Cas9 de sistemas de inmunidad tipo II CRISPR-Cas" . Biología del ARN . 10 (5): 726–737. doi : 10.4161 / rna.24321 . PMC 3737331 . PMID 23563642 .
- ^ Makarova KS, Zhang F, Koonin EV (enero de 2017). "Instantánea: Sistemas CRISPR-Cas Clase 2" . Celular . 168 (1–2): 328–328.e1. doi : 10.1016 / j.cell.2016.12.038 . PMID 28086097 .
- ^ Cox DB, Gootenberg JS, Abudayyeh OO, Franklin B, Kellner MJ, Joung J, Zhang F (noviembre de 2017). "Edición de ARN con CRISPR-Cas13" . Ciencia . 358 (6366): 1019–1027. Código bibliográfico : 2017Sci ... 358.1019C . doi : 10.1126 / science.aaq0180 . PMC 5793859 . PMID 29070703 .
- ^ Azangou-Khyavy, M. et al. (2020) 'CRISPR / Cas: De la edición de genes tumorales a la inmunoterapia del cáncer basada en células T', Frontiers in Immunology, 11. doi: 10.3389 / fimmu.2020.02062.
- ↑ a b Aliyari R, Ding SW (enero de 2009). "Inmunidad viral basada en ARN iniciada por la familia Dicer de receptores inmunes del huésped" . Revisiones inmunológicas . 227 (1): 176–188. doi : 10.1111 / j.1600-065X.2008.00722.x . PMC 2676720 . PMID 19120484 .
- ^ Dugar G, Herbig A, Förstner KU, Heidrich N, Reinhardt R, Nieselt K, Sharma CM (mayo de 2013). "Los mapas de transcriptomas de alta resolución revelan características reguladoras específicas de la cepa de múltiples aislados de Campylobacter jejuni" . PLOS Genetics . 9 (5): e1003495. doi : 10.1371 / journal.pgen.1003495 . PMC 3656092 . PMID 23696746 .
- ^ Hatoum-Aslan A, Maniv I, Marraffini LA (diciembre de 2011). "La longitud del ARN (crRNA) de repeticiones palindrómicas cortas agrupadas maduras, regularmente interespaciadas se mide mediante un mecanismo de regla anclado en el sitio de procesamiento del precursor" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (52): 21218–21222. Código bibliográfico : 2011PNAS..10821218H . doi : 10.1073 / pnas.1112832108 . PMC 3248500 . PMID 22160698 .
- ^ a b Yosef I, Goren MG, Qimron U (julio de 2012). "Proteínas y elementos de ADN esenciales para el proceso de adaptación CRISPR en Escherichia coli " . Investigación de ácidos nucleicos . 40 (12): 5569–5576. doi : 10.1093 / nar / gks216 . PMC 3384332 . PMID 22402487 .
- ↑ a b c d Swarts DC, Mosterd C, van Passel MW, Brouns SJ (2012). "La interferencia CRISPR dirige la adquisición del espaciador específico de la hebra" . PLOS ONE . 7 (4): e35888. Código bibliográfico : 2012PLoSO ... 735888S . doi : 10.1371 / journal.pone.0035888 . PMC 3338789 . PMID 22558257 .
- ^ Babu M , Beloglazova N, Flick R, Graham C, Skarina T, Nocek B, et al. (Enero de 2011). "Una función dual del sistema CRISPR-Cas en la inmunidad antivirus bacteriana y la reparación del ADN" . Microbiología molecular . 79 (2): 484–502. doi : 10.1111 / j.1365-2958.2010.07465.x . PMC 3071548 . PMID 21219465 .
- ^ Han D, Lehmann K, Krauss G (junio de 2009). "SSO1450: una proteína CAS1 de Sulfolobus solfataricus P2 con alta afinidad por el ARN y el ADN" . Cartas FEBS . 583 (12): 1928–1932. doi : 10.1016 / j.febslet.2009.04.047 . PMID 19427858 . S2CID 22279972 .
- ^ Wiedenheft B, Zhou K, Jinek M, Coyle SM, Ma W, Doudna JA (junio de 2009). "Base estructural para la actividad DNasa de una proteína conservada implicada en la defensa del genoma mediada por CRISPR". Estructura . 17 (6): 904–912. doi : 10.1016 / j.str.2009.03.019 . PMID 19523907 .
- ^ Beloglazova N, Brown G, Zimmerman MD, Proudfoot M, Makarova KS, Kudritska M, et al. (Julio de 2008). "Una nueva familia de endoribonucleasas específicas de secuencia asociadas con las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas" . La Revista de Química Biológica . 283 (29): 20361–20371. doi : 10.1074 / jbc.M803225200 . PMC 2459268 . PMID 18482976 .
- ^ Samai P, Smith P, Shuman S (diciembre de 2010). "Estructura de una proteína Cas2 asociada a CRISPR de Desulfovibrio vulgaris" . Acta Crystallographica Sección F . 66 (Pt 12): 1552-1556. doi : 10.1107 / S1744309110039801 . PMC 2998353 . PMID 21139194 .
- ^ Nam KH, Ding F, Haitjema C, Huang Q, DeLisa MP, Ke A (octubre de 2012). "Actividad de endonucleasa bicatenaria en Bacillus halodurans agrupadas repeticiones palindrómicas cortas regularmente interespaciadas (CRISPR) - proteína Cas2 asociada" . La Revista de Química Biológica . 287 (43): 35943–35952. doi : 10.1074 / jbc.M112.382598 . PMC 3476262 . PMID 22942283 .
- ↑ a b Nuñez JK, Kranzusch PJ, Noeske J, Wright AV, Davies CW, Doudna JA (junio de 2014). "La formación del complejo Cas1-Cas2 media la adquisición del espaciador durante la inmunidad adaptativa CRISPR-Cas" . Naturaleza Biología Molecular y Estructural . 21 (6): 528–534. doi : 10.1038 / nsmb.2820 . PMC 4075942 . PMID 24793649 .
- ^ Nuñez JK, Lee AS, Engelman A, Doudna JA (marzo de 2015). "Adquisición de espaciador mediada por integrasa durante la inmunidad adaptativa CRISPR-Cas" . Naturaleza . 519 (7542): 193–198. Código Bibliográfico : 2015Natur.519..193N . doi : 10.1038 / nature14237 . PMC 4359072 . PMID 25707795 .
- ^ Wang J, Li J, Zhao H, Sheng G, Wang M, Yin M, Wang Y (noviembre de 2015). "Base estructural y mecanicista de la adquisición de espaciadores dependientes de PAM en sistemas CRISPR-Cas" . Celular . 163 (4): 840–853. doi : 10.1016 / j.cell.2015.10.008 . PMID 26478180 .
- ^ Nuñez JK, Harrington LB, Kranzusch PJ, Engelman AN, Doudna JA (noviembre de 2015). "Captura de ADN extraño durante la inmunidad adaptativa CRISPR-Cas" . Naturaleza . 527 (7579): 535–538. Código Bibliográfico : 2015Natur.527..535N . doi : 10.1038 / nature15760 . PMC 4662619 . PMID 26503043 .
- ^ Sorek R, Lawrence CM, Wiedenheft B (2013). "Sistemas inmunes adaptativos mediados por CRISPR en bacterias y arqueas" . Revisión anual de bioquímica . 82 (1): 237–266. doi : 10.1146 / annurev-biochem-072911-172315 . PMID 23495939 .
- ^ Nuñez JK, Bai L, Harrington LB, Hinder TL, Doudna JA (junio de 2016). "La memoria inmunológica CRISPR requiere un factor de host para la especificidad" . Célula molecular . 62 (6): 824–833. doi : 10.1016 / j.molcel.2016.04.027 . PMID 27211867 .
- ^ Fagerlund RD, Wilkinson ME, Klykov O, Barendregt A, Pearce FG, Kieper SN, Maxwell HW, Capolupo A, Heck AJ, Krause KL, Bostina M, Scheltema RA, Staals RH, Fineran PC (junio de 2017). "Captura e integración de espaciadores mediante un complejo de adaptación CRISPR tipo IF Cas1-Cas2–3" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 114 (26): E5122-E5128. doi : 10.1073 / pnas.1618421114 . PMC 5495228 . PMID 28611213 .
- ^ Rollie C, Graham S, Rouillon C, White MF (febrero de 2018). "Procesamiento de prespacer e integración específica en un sistema CRISPR Tipo IA" . Investigación de ácidos nucleicos . 46 (3): 1007–1020. doi : 10.1093 / nar / gkx1232 . PMC 5815122 . PMID 29228332 .
- ^ a b c d Horvath P, Romero DA, Coûté-Monvoisin AC, Richards M, Deveau H, Moineau S, et al. (Febrero de 2008). "Diversidad, actividad y evolución de loci CRISPR en Streptococcus thermophilus " . Revista de bacteriología . 190 (4): 1401-1412. doi : 10.1128 / JB.01415-07 . PMC 2238196 . PMID 18065539 .
- ^ a b c Deveau H, Barrangou R , Garneau JE, Labonté J, Fremaux C, Boyaval P, Romero DA, Horvath P, Moineau S (febrero de 2008). "Respuesta de fagos a la resistencia codificada por CRISPR en Streptococcus thermophilus " . Revista de bacteriología . 190 (4): 1390-1400. doi : 10.1128 / JB.01412-07 . PMC 2238228 . PMID 18065545 .
- ^ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Almendros C (marzo de 2009). "Las secuencias de motivos cortos determinan los objetivos del sistema de defensa CRISPR procariota" . Microbiología . 155 (Pt 3): 733–740. doi : 10.1099 / mic.0.023960-0 . PMID 19246744 .
- ↑ a b Lillestøl RK, Shah SA, Brügger K, Redder P, Phan H, Christiansen J, Garrett RA (abril de 2009). "Familias CRISPR del género Crenarchaeal Sulfolobus: transcripción bidireccional y propiedades dinámicas" . Microbiología molecular . 72 (1): 259–272. doi : 10.1111 / j.1365-2958.2009.06641.x . PMID 19239620 . S2CID 36258923 .
- ↑ a b Shah SA, Hansen NR, Garrett RA (febrero de 2009). "Distribución de coincidencias espaciador CRISPR en virus y plásmidos de crenarchaeal acidotermophiles e implicaciones para su mecanismo inhibitorio". Transacciones de la sociedad bioquímica . 37 (Pt 1): 23-28. doi : 10.1042 / BST0370023 . PMID 19143596 . S2CID 19093261 .
- ↑ a b Díez-Villaseñor C, Guzmán NM, Almendros C, García-Martínez J, Mojica FJ (mayo de 2013). "Los plásmidos informadores de integración CRISPR-espaciador revelan distintas especificidades de adquisición genuinas entre las variantes CRISPR-Cas IE de Escherichia coli " . Biología del ARN . 10 (5): 792–802. doi : 10.4161 / rna.24023 . PMC 3737337 . PMID 23445770 .
- ↑ a b c Erdmann S, Garrett RA (septiembre de 2012). "Captación selectiva e hiperactiva de ADN extraño por sistemas inmunes adaptativos de un arqueón a través de dos mecanismos distintos" . Microbiología molecular . 85 (6): 1044–1056. doi : 10.1111 / j.1365-2958.2012.08171.x . PMC 3468723 . PMID 22834906 .
- ↑ a b Shah SA, Erdmann S, Mojica FJ, Garrett RA (mayo de 2013). "Motivos de reconocimiento de protospacer: identidades mixtas y diversidad funcional" . Biología del ARN . 10 (5): 891–899. doi : 10.4161 / rna.23764 . PMC 3737346 . PMID 23403393 .
- ^ Andersson AF, Banfield JF (mayo de 2008). "Dinámica de poblaciones de virus y resistencia a virus adquiridos en comunidades microbianas naturales". Ciencia . 320 (5879): 1047–1050. Código Bibliográfico : 2008Sci ... 320.1047A . doi : 10.1126 / science.1157358 . PMID 18497291 . S2CID 26209623 .
- ^ a b c d Pride DT, Sun CL, Salzman J, Rao N, Loomer P, Armitage GC, et al. (Enero de 2011). "El análisis de CRISPR estreptocócicos de la saliva humana revela una diversidad de secuencia sustancial dentro y entre sujetos a lo largo del tiempo" . Investigación del genoma . 21 (1): 126-136. doi : 10.1101 / gr.111732.110 . PMC 3012920 . PMID 21149389 .
- ↑ a b Goren MG, Yosef I, Auster O, Qimron U (octubre de 2012). "Definición experimental de un duplicón palindrómico corto agrupados regularmente interespaciados en Escherichia coli ". Revista de Biología Molecular . 423 (1): 14–16. doi : 10.1016 / j.jmb.2012.06.037 . PMID 22771574 .
- ↑ a b c Datsenko KA, Pougach K, Tikhonov A, Wanner BL, Severinov K, Semenova E (julio de 2012). "La memoria molecular de infecciones previas activa el sistema de inmunidad bacteriana adaptativa CRISPR / Cas" . Comunicaciones de la naturaleza . 3 : 945. Bibcode : 2012NatCo ... 3..945D . doi : 10.1038 / ncomms1937 . PMID 22781758 .
- ^ Gesner EM, Schellenberg MJ, Garside EL, George MM, Macmillan AM (junio de 2011). "Reconocimiento y maduración de ARN efectores en una vía de interferencia CRISPR". Naturaleza Biología Molecular y Estructural . 18 (6): 688–692. doi : 10.1038 / nsmb.2042 . PMID 21572444 . S2CID 677704 .
- ^ Sashital DG, Jinek M, Doudna JA (junio de 2011). "Un cambio conformacional inducido por ARN requerido para la escisión del ARN CRISPR por la endoribonucleasa Cse3". Naturaleza Biología Molecular y Estructural . 18 (6): 680–687. doi : 10.1038 / nsmb.2043 . PMID 21572442 . S2CID 5538195 .
- ^ Haurwitz RE, Jinek M, Wiedenheft B, Zhou K, Doudna JA (septiembre de 2010). "Procesamiento de ARN específico de secuencia y estructura por una endonucleasa CRISPR" . Ciencia . 329 (5997): 1355-1358. Código Bibliográfico : 2010Sci ... 329.1355H . doi : 10.1126 / science.1192272 . PMC 3133607 . PMID 20829488 .
- ↑ a b Kunin V, Sorek R, Hugenholtz P (2007). "Conservación evolutiva de secuencia y estructuras secundarias en repeticiones CRISPR" . Biología del genoma . 8 (4): R61. doi : 10.1186 / gb-2007-8-4-r61 . PMC 1896005 . PMID 17442114 .
- ^ Carte J, Wang R, Li H, Charranes RM, Charranes MP (diciembre de 2008). "Cas6 es una endoribonucleasa que genera ARN guía para la defensa invasora en procariotas" . Genes y desarrollo . 22 (24): 3489–3496. doi : 10.1101 / gad.1742908 . PMC 2607076 . PMID 19141480 .
- ^ Wang R, Preamplume G, Charranes MP, Charranes RM, Li H (febrero de 2011). "Interacción de la riboendonucleasa Cas6 con ARN CRISPR: reconocimiento y escisión" . Estructura . 19 (2): 257–264. doi : 10.1016 / j.str.2010.11.014 . PMC 3154685 . PMID 21300293 .
- ^ Niewoehner O, Jinek M, Doudna JA (enero de 2014). "Evolución del reconocimiento y procesamiento de ARN CRISPR por endonucleasas Cas6" . Investigación de ácidos nucleicos . 42 (2): 1341-1353. doi : 10.1093 / nar / gkt922 . PMC 3902920 . PMID 24150936 .
- ^ Semenova E, Jore MM, Datsenko KA, Semenova A, Westra ER, Wanner B, et al. (Junio de 2011). "La interferencia del ARN de repetición palindrómica corta (CRISPR) agrupada regularmente interespaciada está gobernada por una secuencia de semillas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (25): 10098–10103. Código Bibliográfico : 2011PNAS..10810098S . doi : 10.1073 / pnas.1104144108 . PMC 3121866 . PMID 21646539 .
- ^ Gudbergsdottir S, Deng L, Chen Z, Jensen JV, Jensen LR, She Q, Garrett RA (enero de 2011). "Propiedades dinámicas de los sistemas Sulfolobus CRISPR / Cas y CRISPR / Cmr cuando se desafían con genes virales y plasmídicos transmitidos por vectores y protoespaciadores" . Microbiología molecular . 79 (1): 35–49. doi : 10.1111 / j.1365-2958.2010.07452.x . PMC 3025118 . PMID 21166892 .
- ^ Manica A, Zebec Z, Teichmann D, Schleper C (abril de 2011). "Actividad in vivo de defensa de virus mediada por CRISPR en una arqueona hipertermofílica". Microbiología molecular . 80 (2): 481–491. doi : 10.1111 / j.1365-2958.2011.07586.x . PMID 21385233 . S2CID 41442419 .
- ^ Jore MM, Lundgren M, van Duijn E, Bultema JB, Westra ER, Waghmare SP, et al. (Mayo de 2011). "Base estructural para el reconocimiento de ADN guiado por ARN CRISPR por Cascade" (PDF) . Naturaleza Biología Molecular y Estructural . 18 (5): 529–536. doi : 10.1038 / nsmb.2019 . PMID 21460843 . S2CID 10987554 .
- ^ Wiedenheft B, Lander GC, Zhou K, Jore MM, Brouns SJ, van der Oost J, Doudna JA , Nogales E (septiembre de 2011). "Estructuras del complejo de vigilancia guiada por ARN de un sistema inmunológico bacteriano" . Naturaleza . 477 (7365): 486–489. Código Bib : 2011Natur.477..486W . doi : 10.1038 / nature10402 . PMC 4165517 . PMID 21938068 .
- ^ Zhang J, Rouillon C, Kerou M, Reeks J, Brugger K, Graham S, Reimann J, Cannone G, Liu H, Albers SV, Naismith JH, Spagnolo L, White MF (febrero de 2012). "Estructura y mecanismo del complejo CMR para la inmunidad antiviral mediada por CRISPR" . Célula molecular . 45 (3): 303–313. doi : 10.1016 / j.molcel.2011.12.013 . PMC 3381847 . PMID 22227115 .
- ↑ a b Hale CR, Zhao P, Olson S, Duff MO, Graveley BR, Wells L, Terns RM, Terns MP (noviembre de 2009). "Escisión de ARN guiada por ARN por un complejo proteico CRISPR ARN-Cas" . Celular . 139 (5): 945–956. doi : 10.1016 / j.cell.2009.07.040 . PMC 2951265 . PMID 19945378 .
- ^ Estrella MA, Kuo FT, Bailey S (2016). "Escisión de ADN activado por ARN por el complejo efector tipo III-B CRISPR-Cas" . Genes y desarrollo . 30 (4): 460–470. doi : 10.1101 / gad.273722.115 . PMC 4762430 . PMID 26848046 .
- ^ Samai P, Pyenson N, Jiang W, Goldberg GW, Hatoum-Aslan A, Marraffini LA (2015). "Escisión de ADN y ARN co-transcripcional durante la inmunidad tipo III CRISPR-Cas" . Celular . 161 (5): 1164-1174. doi : 10.1016 / j.cell.2015.04.027 . PMC 4594840 . PMID 25959775 .
- ↑ a b c Marraffini LA, Sontheimer EJ (enero de 2010). "Discriminación propia versus no propia durante la inmunidad dirigida por ARN CRISPR" . Naturaleza . 463 (7280): 568–571. Código Bibliográfico : 2010Natur.463..568M . doi : 10.1038 / nature08703 . PMC 2813891 . PMID 20072129 .
- ^ Krupovic M, Béguin P, Koonin EV (agosto de 2017). "Casposons: elementos genéticos móviles que dieron lugar a la maquinaria de adaptación CRISPR-Cas" . Opinión actual en microbiología . 38 : 36–43. doi : 10.1016 / j.mib.2017.04.004 . PMC 5665730 . PMID 28472712 .
- ^ Koonin EV , Makarova KS (mayo de 2013). "CRISPR-Cas: evolución de un sistema de inmunidad adaptativa basado en ARN en procariotas" . Biología del ARN . 10 (5): 679–686. doi : 10.4161 / rna.24022 . PMC 3737325 . PMID 23439366 .
- ^ Koonin EV , Makarova KS, Zhang F (junio de 2017). "Diversidad, clasificación y evolución de los sistemas CRISPR-Cas" . Opinión actual en microbiología . 37 : 67–78. doi : 10.1016 / j.mib.2017.05.008 . PMC 5776717 . PMID 28605718 .
- ^ a b Shmakov S, Smargon A, Scott D, Cox D, Pyzocha N, Yan W, Abudayyeh OO, Gootenberg JS, Makarova KS, Wolf YI, Severinov K, Zhang F , Koonin EV (marzo de 2017). "Diversidad y evolución de los sistemas CRISPR-Cas clase 2" . Reseñas de la naturaleza. Microbiología . 15 (3): 169-182. doi : 10.1038 / nrmicro.2016.184 . PMC 5851899 . PMID 28111461 .
- ^ Kupczok A, Bollback JP (febrero de 2013). "Modelos probabilísticos para la evolución del contenido del espaciador CRISPR" . Biología Evolutiva BMC . 13 (1): 54. doi : 10.1186 / 1471-2148-13-54 . PMC 3704272 . PMID 23442002 .
- ^ Sternberg SH, Richter H, Charpentier E, Qimron U (marzo de 2016). "Adaptación en sistemas CRISPR-Cas". Célula molecular . 61 (6): 797–808. doi : 10.1016 / j.molcel.2016.01.030 . hdl : 21.11116 / 0000-0003-E74E-2 . PMID 26949040 .
- ^ Koonin EV , Wolf YI (noviembre de 2009). "¿La evolución es darwiniana o / o lamarckiana?" . Biology Direct . 4 : 42. doi : 10.1186 / 1745-6150-4-42 . PMC 2781790 . PMID 19906303 .
- ^ Weiss A (octubre de 2015). "Ilusiones lamarckianas" . Tendencias en Ecología y Evolución . 30 (10): 566–568. doi : 10.1016 / j.tree.2015.08.003 . PMID 26411613 .
- ^ a b Koonin EV , Wolf YI (febrero de 2016). "Cómo lamarckiana es la inmunidad CRISPR-Cas: el continuo de los mecanismos de evolución" . Biology Direct . 11 (1): 9. doi : 10.1186 / s13062-016-0111-z . PMC 4765028 . PMID 26912144 .
- ^ Heidelberg JF, Nelson WC, Schoenfeld T, Bhaya D (2009). Ahmed N (ed.). "Guerra de gérmenes en una comunidad de alfombrillas microbianas: CRISPR proporcionan información sobre la evolución conjunta de los genomas virales y del huésped" . PLOS ONE . 4 (1): e4169. Código bibliográfico : 2009PLoSO ... 4.4169H . doi : 10.1371 / journal.pone.0004169 . PMC 2612747 . PMID 19132092 .
- ^ Sampson TR, Saroj SD, Llewellyn AC, Tzeng YL, Weiss DS (mayo de 2013). "Un sistema CRISPR / Cas media la evasión inmune innata bacteriana y la virulencia" . Naturaleza . 497 (7448): 254-257. Código bibliográfico : 2013Natur.497..254S . doi : 10.1038 / nature12048 . PMC 3651764 . PMID 23584588 .
- ^ Touchon M, Rocha EP (junio de 2010). Randau L (ed.). "El pequeño, lento y especializado CRISPR y anti-CRISPR de Escherichia y Salmonella" . PLOS ONE . 5 (6): e11126. Código Bibliográfico : 2010PLoSO ... 511126T . doi : 10.1371 / journal.pone.0011126 . PMC 2886076 . PMID 20559554 .
- ↑ a b c Rho M, Wu YW, Tang H, Doak TG, Ye Y (2012). "Diversos CRISPR en evolución en microbiomas humanos" . PLOS Genetics . 8 (6): e1002441. doi : 10.1371 / journal.pgen.1002441 . PMC 3374615 . PMID 22719260 .
- ↑ a b Sun CL, Barrangou R , Thomas BC, Horvath P, Fremaux C, Banfield JF (febrero de 2013). "Mutaciones de fagos en respuesta a la diversificación CRISPR en una población bacteriana". Microbiología ambiental . 15 (2): 463–470. doi : 10.1111 / j.1462-2920.2012.02879.x . PMID 23057534 .
- ^ Kuno S, Sako Y, Yoshida T (mayo de 2014). "Diversificación de CRISPR dentro de genotipos coexistentes en una población natural de la cianobacteria microcystis aeruginosa formadora de flores". Microbiología . 160 (Parte 5): 903–916. doi : 10.1099 / mic.0.073494-0 . PMID 24586036 .
- ^ Sorek R, Kunin V, Hugenholtz P (marzo de 2008). "CRISPR: un sistema generalizado que proporciona resistencia adquirida contra fagos en bacterias y arqueas" . Reseñas de la naturaleza. Microbiología . 6 (3): 181–186. doi : 10.1038 / nrmicro1793 . PMID 18157154 . S2CID 3538077 .
Tabla 1: Recursos web para el análisis CRISPR
- ^ Orgullo DT, Salzman J, Relman DA (septiembre de 2012). "Las comparaciones de viromas y repeticiones palindrómicas cortas agrupadas regularmente interespaciadas en la saliva humana revelan adaptaciones bacterianas a los virus salivales" . Microbiología ambiental . 14 (9): 2564-2576. doi : 10.1111 / j.1462-2920.2012.02775.x . PMC 3424356 . PMID 22583485 .
- ^ Held NL, Herrera A, Whitaker RJ (noviembre de 2013). "Reordenamiento de loci espaciador de repetición CRISPR en Sulfolobus islandicus". Microbiología ambiental . 15 (11): 3065–3076. doi : 10.1111 / 1462-2920.12146 . PMID 23701169 .
- ^ Held NL, Herrera A, Cadillo-Quiroz H, Whitaker RJ (septiembre de 2010). "Diversidad asociada a CRISPR dentro de una población de Sulfolobus islandicus" . PLOS ONE . 5 (9): e12988. Código bibliográfico : 2010PLoSO ... 512988H . doi : 10.1371 / journal.pone.0012988 . PMC 2946923 . PMID 20927396 .
- ^ a b c Medvedeva S, Liu Y, Koonin EV, Severinov K, Prangishvili D, Krupovic M (noviembre de 2019). "Las matrices mini-CRISPR transmitidas por virus están involucradas en conflictos intervirales" . Comunicaciones de la naturaleza . 10 (1): 5204. Bibcode : 2019NatCo..10.5204M . doi : 10.1038 / s41467-019-13205-2 . PMC 6858448 . PMID 31729390 .
- ^ Skennerton CT, Imelfort M, Tyson GW (mayo de 2013). "Crass: identificación y reconstrucción de CRISPR a partir de datos metagenómicos sin ensamblar" . Investigación de ácidos nucleicos . 41 (10): e105. doi : 10.1093 / nar / gkt183 . PMC 3664793 . PMID 23511966 .
- ^ Stern A, Mick E, Tirosh I, Sagy O, Sorek R (octubre de 2012). "La orientación de CRISPR revela un depósito de fagos comunes asociados con el microbioma intestinal humano" . Investigación del genoma . 22 (10): 1985–1994. doi : 10.1101 / gr.138297.112 . PMC 3460193 . PMID 22732228 .
- ^ Novick RP, Christie GE, Penadés JR (agosto de 2010). "Las islas cromosómicas relacionadas con fagos de bacterias Gram-positivas" . Nature Reviews Microbiología . 8 (8): 541–551. doi : 10.1038 / nrmicro2393 . PMC 3522866 . PMID 20634809 .
- ^ Ram G, Chen J, Kumar K, Ross HF, Ubeda C, Damle PK, Lane KD, Penadés JR, Christie GE, Novick RP (octubre de 2012). "La interferencia de la isla de patogenicidad estafilocócica con la reproducción del fago auxiliar es un paradigma de parasitismo molecular" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (40): 16300–16305. Código Bibliográfico : 2012PNAS..10916300R . doi : 10.1073 / pnas.1204615109 . PMC 3479557 . PMID 22991467 .
- ^ Ram G, Chen J, Ross HF, Novick RP (octubre de 2014). "Interferencia de isla de patogenicidad modulada con precisión con transcripción de genes de fagos tardíos" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 111 (40): 14536-14541. Código Bibliográfico : 2014PNAS..11114536R . doi : 10.1073 / pnas.1406749111 . PMC 4209980 . PMID 25246539 .
- ^ Seed KD, Lazinski DW, Calderwood SB, Camilli A (febrero de 2013). "Un bacteriófago codifica su propia respuesta adaptativa CRISPR / Cas para evadir la inmunidad innata del huésped" . Naturaleza . 494 (7438): 489–491. Código Bibliográfico : 2013Natur.494..489S . doi : 10.1038 / nature11927 . PMC 3587790 . PMID 23446421 .
- ^ "¿Qué es CRISPR y cómo funciona?" . Livescience.Tech . Consultado el 14 de diciembre de 2019 .
- ^ Doudna JA , Charpentier E (noviembre de 2014). "Edición del genoma. La nueva frontera de la ingeniería del genoma con CRISPR-Cas9". Ciencia . 346 (6213): 1258096. doi : 10.1126 / science.1258096 . PMID 25430774 . S2CID 6299381 .
- ↑ a b Ledford H (junio de 2015). "CRISPR, el disruptor" . Naturaleza . 522 (7554): 20–24. Bibcode : 2015Natur.522 ... 20L . doi : 10.1038 / 522020a . PMID 26040877 .
- ^ Alphey L (2016). "¿Pueden los impulsores del gen CRISPR-Cas9 frenar la malaria?" . Biotecnología de la naturaleza . 34 (2): 149-150. doi : 10.1038 / nbt.3473 . PMID 26849518 . S2CID 10014014 .
- ^ Bernabé-Orts JM, Casas-Rodrigo I, Minguet EG, Landolfi V, Garcia-Carpintero V, Gianoglio S, et al. (Abril de 2019). "Evaluación de la eficiencia de edición de genes mediada por Cas12a en plantas" . Revista de Biotecnología Vegetal . 17 (10): 1971–1984. doi : 10.1111 / pbi.13113 . PMC 6737022 . PMID 30950179 .
- ^ "En primer lugar, los médicos en Estados Unidos utilizan la herramienta CRISPR para tratar a pacientes con trastorno genético" . NPR.org . Consultado el 31 de julio de 2019 .
- ^ Abuso, Instituto Nacional de Drogas (14 de febrero de 2020). "La terapia antirretroviral combinada con la edición de genes CRISPR puede eliminar la infección por VIH en ratones" . Instituto Nacional de Abuso de Drogas . Consultado el 15 de noviembre de 2020 .
- ^ En primer lugar, los científicos utilizan una revolucionaria herramienta de edición de genes para editar el interior de un paciente
- ↑ The-Crispr (15 de julio de 2019). "Escuchar podcast de Radiolab CRISPR" . El Crispr . Consultado el 15 de julio de 2019 .
- ^ Ledford H (2017). "CRISPR estudia resultados turbios de investigación de genes más antiguos". Naturaleza . doi : 10.1038 / nature.2017.21763 . S2CID 90757972 .
- ^ Gu W, Crawford ED, O'Donovan BD, Wilson MR, Chow ED, Retallack H, DeRisi JL (marzo de 2016). "Agotamiento de secuencias abundantes por hibridación (DASH): utilizando Cas9 para eliminar especies no deseadas de alta abundancia en bibliotecas de secuenciación y aplicaciones de conteo molecular" . Biología del genoma . 17 (1): 41. doi : 10.1186 / s13059-016-0904-5 . PMC 4778327 . PMID 26944702 .
- ^ Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, et al. (Abril de 2017). "Detección de ácidos nucleicos con CRISPR-Cas13a / C2c2" . Ciencia . 356 (6336): 438–442. Código bibliográfico : 2017Sci ... 356..438G . doi : 10.1126 / science.aam9321 . PMC 5526198 . PMID 28408723 .
- ^ Chen JS, Ma E, Harrington LB, Da Costa M, Tian X, Palefsky JM, Doudna JA (abril de 2018). "La unión de la diana CRISPR-Cas12a desencadena una actividad indiscriminada de ADNasa monocatenaria" . Ciencia . 360 (6387): 436–439. Código bibliográfico : 2018Sci ... 360..436C . doi : 10.1126 / science.aar6245 . PMC 6628903 . PMID 29449511 .
- ^ Iwasaki RS, Batey RT (September 2020). "SPRINT: a Cas13a-based platform for detection of small molecules". Nucleic Acids Research. 48 (17): e101. doi:10.1093/nar/gkaa673. PMID 32797156.
- ^ Broughton JP, Deng X, Yu G, Fasching CL, Servellita V, Singh J, et al. (July 2020). "CRISPR-Cas12-based detection of SARS-CoV-2". Nature Biotechnology. 38 (7): 870–874. doi:10.1038/s41587-020-0513-4. PMID 32300245.
- ^ Joung J, Ladha A, Saito M, Kim NG, Woolley AE, Segel M, et al. (October 2020). "Detection of SARS-CoV-2 with SHERLOCK One-Pot Testing". The New England Journal of Medicine. 383 (15): 1492–1494. doi:10.1056/NEJMc2026172. PMC 7510942. PMID 32937062.
- ^ Dhamad AE, Abdal Rhida MA (2020). "COVID-19: molecular and serological detection methods". PeerJ. 8: e10180. doi:10.7717/peerj.10180. PMC 7547594. PMID 33083156.
- ^ Patchsung M, Jantarug K, Pattama A, Aphicho K, Suraritdechachai S, Meesawat P, et al. (December 2020). "Clinical validation of a Cas13-based assay for the detection of SARS-CoV-2 RNA". Nature Biomedical Engineering. 4 (12): 1140–1149. doi:10.1038/s41551-020-00603-x. PMID 32848209.
- ^ Nguyen LT, Smith BM, Jain PK (September 2020). "Enhancement of trans-cleavage activity of Cas12a with engineered crRNA enables amplified nucleic acid detection". Nature Communications. 11 (1): 4906. doi:10.1038/s41467-020-18615-1. PMID 32999292.
- ^ Konwarh R (September 2020). "Can CRISPR/Cas Technology Be a Felicitous Stratagem Against the COVID-19 Fiasco? Prospects and Hitches". Frontiers in Molecular Biosciences. 7: 557377. doi:10.3389/fmolb.2020.557377. PMC 7511716. PMID 33134311.
Further reading[edit]
- Doudna J, Mali P (23 March 2016). CRISPR-Cas: A Laboratory Manual. New York: Cold Spring Harbor Laboratory Press. ISBN 978-1-62182-131-1.
- Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J (August 2016). "Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems" (PDF). Science. 353 (6299): aad5147. doi:10.1126/science.aad5147. hdl:1721.1/113195. PMID 27493190. S2CID 11086282.
- Sander JD, Joung JK (April 2014). "CRISPR-Cas systems for editing, regulating and targeting genomes". Nature Biotechnology. 32 (4): 347–355. doi:10.1038/nbt.2842. PMC 4022601. PMID 24584096.
- Slaymaker IM, Gao L, Zetsche B, Scott DA, Yan WX, Zhang F (January 2016). "Rationally engineered Cas9 nucleases with improved specificity". Science. 351 (6268): 84–88. Bibcode:2016Sci...351...84S. doi:10.1126/science.aad5227. PMC 4714946. PMID 26628643.
- Terns RM, Terns MP (March 2014). "CRISPR-based technologies: prokaryotic defense weapons repurposed". Trends in Genetics. 30 (3): 111–118. doi:10.1016/j.tig.2014.01.003. PMC 3981743. PMID 24555991.
- Westra ER, Buckling A, Fineran PC (May 2014). "CRISPR-Cas systems: beyond adaptive immunity". Nature Reviews Microbiology. 12 (5): 317–326. doi:10.1038/nrmicro3241. PMID 24704746. S2CID 36575361.
- Andersson AF, Banfield JF (May 2008). "Virus population dynamics and acquired virus resistance in natural microbial communities". Science. 320 (5879): 1047–1050. Bibcode:2008Sci...320.1047A. doi:10.1126/science.1157358. PMID 18497291. S2CID 26209623.
- Hale C, Kleppe K, Terns RM, Terns MP (December 2008). "Prokaryotic silencing (psi)RNAs in Pyrococcus furiosus". RNA. 14 (12): 2572–2579. doi:10.1261/rna.1246808. PMC 2590957. PMID 18971321.
- van der Ploeg JR (June 2009). "Analysis of CRISPR in Streptococcus mutans suggests frequent occurrence of acquired immunity against infection by M102-like bacteriophages" (PDF). Microbiology. 155 (Pt 6): 1966–1976. doi:10.1099/mic.0.027508-0. PMID 19383692.
- van der Oost J, Brouns SJ (November 2009). "RNAi: prokaryotes get in on the act". Cell. 139 (5): 863–865. doi:10.1016/j.cell.2009.11.018. PMID 19945373. S2CID 11863610.
- Karginov FV, Hannon GJ (January 2010). "The CRISPR system: small RNA-guided defense in bacteria and archaea". Molecular Cell. 37 (1): 7–19. doi:10.1016/j.molcel.2009.12.033. PMC 2819186. PMID 20129051.
- Pul U, Wurm R, Arslan Z, Geissen R, Hofmann N, Wagner R (March 2010). "Identification and characterization of E. coli CRISPR-cas promoters and their silencing by H-NS". Molecular Microbiology. 75 (6): 1495–1512. doi:10.1111/j.1365-2958.2010.07073.x. PMID 20132443. S2CID 37529215.
- Díez-Villaseñor C, Almendros C, García-Martínez J, Mojica FJ (May 2010). "Diversity of CRISPR loci in Escherichia coli". Microbiology. 156 (Pt 5): 1351–1361. doi:10.1099/mic.0.036046-0. PMID 20133361.
- Deveau H, Garneau JE, Moineau S (2010). "CRISPR/Cas system and its role in phage-bacteria interactions". Annual Review of Microbiology. 64: 475–493. doi:10.1146/annurev.micro.112408.134123. PMID 20528693.
- Koonin EV, Makarova KS (December 2009). "CRISPR-Cas: an adaptive immunity system in prokaryotes". F1000 Biology Reports. 1: 95. doi:10.3410/B1-95. PMC 2884157. PMID 20556198.
- "The age of the red pen". The Economist. August 22, 2015. ISSN 0013-0613. Retrieved 2015-08-25.
- Ran AF, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (2013). "Genome engineering using the CRISPR-Cas9 system". Nature Protocols. 8 (11): 2281–2308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
External links[edit]
| Wikimedia Commons has media related to CRISPR. |
| Scholia has a topic profile for CRISPR. |
- Advanced Gene Editing: CRISPR-Cas9 Congressional Research Service
- Jennifer Doudna talk: Genome Engineering with CRISPR-Cas9: Birth of a Breakthrough Technology
- Overview of all the structural information available in the PDB for UniProt: Q46901 (CRISPR system Cascade subunit CasA) at the PDBe-KB.
- Overview of all the structural information available in the PDB for UniProt: P76632 (CRISPR system Cascade subunit CasB) at the PDBe-KB.
- Overview of all the structural information available in the PDB for UniProt: Q46899 (CRISPR system Cascade subunit CasC) at the PDBe-KB.
- Overview of all the structural information available in the PDB for UniProt: Q46898 (CRISPR system Cascade subunit CasD) at the PDBe-KB.
- Overview of all the structural information available in the PDB for UniProt: Q46897 (CRISPR system Cascade subunit CasE) at the PDBe-KB.