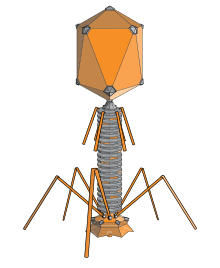
Bacteriófago

Un bacteriófago ( / b æ k t ɪər i oʊ f eɪ dʒ / ), también conocido informalmente como un fago ( / f eɪ dʒ / ), es un virus que infecta y se replica dentro de las bacterias y arqueas . El término se deriva de "bacteria" y del griego φαγεῖν ( phagein ), que significa "devorar". Los bacteriófagos están compuestos por proteínas que encapsulan un ADN o Genoma de ARN , y puede tener estructuras que son simples o elaboradas. Sus genomas pueden codificar tan solo cuatro genes (por ejemplo, MS2 ) y hasta cientos de genes . Los fagos se replican dentro de la bacteria después de la inyección de su genoma en su citoplasma .
Los bacteriófagos se encuentran entre las entidades más comunes y diversas de la biosfera . [2] Los bacteriófagos son virus ubicuos que se encuentran dondequiera que existan bacterias. Se estima que hay más de 10 31 bacteriófagos en el planeta, más que cualquier otro organismo de la Tierra, incluidas las bacterias, juntos. [3] Los virus son la entidad biológica más abundante en la columna de agua de los océanos del mundo y el segundo componente más grande de biomasa después de los procariotas , [4] donde se han encontrado hasta 9x10 8 viriones por mililitro en las capas microbianas de la superficie. [5] y hasta el 70% de las bacterias marinaspuede estar infectado por fagos. [6]
Los fagos se han utilizado desde finales del siglo XX como alternativa a los antibióticos en la ex Unión Soviética y Europa Central, así como en Francia. [7] [8] Se ven como una posible terapia contra cepas de muchas bacterias resistentes a múltiples fármacos (ver terapia con fagos ). [9] Por otro lado, se ha demostrado que los fagos de Inoviridae complican las biopelículas involucradas en la neumonía y la fibrosis quística y protegen a las bacterias de los medicamentos destinados a erradicar la enfermedad, promoviendo así la infección persistente. [10]
Clasificación
Los bacteriófagos se encuentran abundantemente en la biosfera, con diferentes genomas y estilos de vida. Los fagos son clasificados por el Comité Internacional de Taxonomía de Virus (ICTV) según su morfología y ácido nucleico.
| Pedido | Familia | Morfología | Ácido nucleico | Ejemplos de |
|---|---|---|---|---|
| Belfryvirales | Turriviridae | Envuelto, isométrico | ADN de doble cadena lineal | |
| Caudovirales | Ackermannviridae | Cola contráctil no envuelta | ADN de doble cadena lineal | |
| Autographiviridae | Cola no envuelta, no contráctil (corta) | ADN de doble cadena lineal | ||
| Chaseviridae | ADN de doble cadena lineal | |||
| Demerecviridae | ADN de doble cadena lineal | |||
| Drexlerviridae | ADN de doble cadena lineal | |||
| Guenliviridae | ADN de doble cadena lineal | |||
| Herelleviridae | Cola contráctil no envuelta | ADN de doble cadena lineal | ||
| Myoviridae | Cola contráctil no envuelta | ADN de doble cadena lineal | T4 , Mu , P1 , P2 | |
| Siphoviridae | Cola no envuelta, no contráctil (larga) | ADN de doble cadena lineal | λ , T5 , HK97 , N15 | |
| Podoviridae | Cola no envuelta, no contráctil (corta) | ADN de doble cadena lineal | T7 , T3 , Φ29 , P22 | |
| Rountreeviridae | ADN de doble cadena lineal | |||
| Salasmaviridae | ADN de doble cadena lineal | |||
| Schitoviridae | ADN de doble cadena lineal | |||
| Zobellviridae | ADN de doble cadena lineal | |||
| Halopanivirales | Sphaerolipoviridae | Envuelto, isométrico | ADN de doble cadena lineal | |
| Simuloviridae | Envuelto, isométrico | ADN de doble cadena lineal | ||
| Matshushitaviridae | Envuelto, isométrico | ADN de doble cadena lineal | ||
| Haloruvirales | Pleolipoviridae | Envuelto, pleomórfico | SsDNA circular, dsDNA circular o dsDNA lineal | |
| Kalamavirales | Tectiviridae | No envuelto, isométrico | ADN de doble cadena lineal | |
| Ligamenvirales | Lipothrixviridae | Envuelto, en forma de varilla | ADN de doble cadena lineal | Virus Acidianus filamentoso 1 |
| Rudiviridae | No envuelto, en forma de varilla | ADN de doble cadena lineal | Virus 1 con forma de bastoncillo de Sulfolobus islandicus | |
| Mindivirales | Cystoviridae | Envuelto, esférico | DsRNA lineal | Φ6 |
| Norzivirales | Atkinsviridae | No envuelto, isométrico | SsRNA lineal | |
| Duinviridae | No envuelto, isométrico | SsRNA lineal | ||
| Fiersviridae | No envuelto, isométrico | SsRNA lineal | MS2 , Qβ | |
| Solspiviridae | No envuelto, isométrico | SsRNA lineal | ||
| Petitvirales | Microviridae | No envuelto, isométrico | SsDNA circular | ΦX174 |
| Primavirales | Tristromaviridae | Envuelto, en forma de varilla | ADN de doble cadena lineal | |
| Timlovirales | Blumeviridae | No envuelto, isométrico | SsRNA lineal | |
| Steitzviridae | No envuelto, isométrico | SsRNA lineal | ||
| Tubulavirales | Inoviridae | No envuelto, filamentoso | SsDNA circular | M13 |
| Paulinoviridae | No envuelto, filamentoso | SsDNA circular | ||
| Plectroviridae | No envuelto, filamentoso | SsDNA circular | ||
| Vinavirales | Corticoviridae | No envuelto, isométrico | ADN ds circular | PM2 |
| Durnavirales | Picobirnaviridae (propuesta) | No envuelto, isométrico | DsRNA lineal | |
| Sin asignar | Ampullaviridae | Envuelto, en forma de botella | ADN de doble cadena lineal | |
| Autolykiviridae | No envuelto, isométrico | ADN de doble cadena lineal | ||
| Bicaudaviridae | Sin envolver, en forma de limón | ADN ds circular | ||
| Clavaviridae | No envuelto, en forma de varilla | ADN ds circular | ||
| Finnlakeviridae | No envuelto, isométrico | SsDNA circular | FLIP [11] | |
| Fuselloviridae | Sin envolver, en forma de limón | ADN ds circular | ||
| Globuloviridae | Envuelto, isométrico | ADN de doble cadena lineal | ||
| Guttaviridae | No envuelto, ovoide | ADN ds circular | ||
| Halspiviridae | Sin envolver, en forma de limón | ADN de doble cadena lineal | ||
| Plasmaviridae | Envuelto, pleomórfico | ADN ds circular | ||
| Portogloboviridae | Envuelto, isométrico | ADN ds circular | ||
| Thaspiviridae | Sin envolver, en forma de limón | ADN de doble cadena lineal | ||
| Spiraviridae | No envuelto, en forma de varilla | SsDNA circular |
Se ha sugerido que los miembros de Picobirnaviridae infectan bacterias, pero no mamíferos. [12]
También hay muchos géneros no asignados de la clase Leviviricetes : Chimpavirus , Hohglivirus , Mahrahvirus , Meihzavirus , Nicedsevirus , Sculuvirus , Skrubnovirus , Tetipavirus y Winunavirus que contienen genomas de ssRNA lineales [13] y el género Lilyvirus no asignado del orden Caudovirales que contiene un genoma de dsDNA lineal.
Historia
En 1896, Ernest Hanbury Hankin informó que algo en las aguas de los ríos Ganges y Yamuna en la India tenía una marcada acción antibacteriana contra el cólera y podía pasar a través de un filtro de porcelana muy fino. [14] En 1915, el bacteriólogo británico Frederick Twort , superintendente de la Brown Institution de Londres, descubrió un pequeño agente que infectaba y mataba bacterias. Creía que el agente debía ser uno de los siguientes:
- una etapa en el ciclo de vida de las bacterias
- una enzima producida por las propias bacterias, o
- un virus que creció y destruyó la bacteria [15]
La investigación de Twort se vio interrumpida por el inicio de la Primera Guerra Mundial , así como por la escasez de fondos y los descubrimientos de antibióticos.
Independientemente, el microbiólogo francocanadiense Félix d'Hérelle , que trabaja en el Instituto Pasteur de París , anunció el 3 de septiembre de 1917 que había descubierto "un microbio invisible y antagonista del bacilo de la disentería ". Para d'Hérelle, no había dudas sobre la naturaleza de su descubrimiento: "En un instante lo había entendido: lo que causaba mis manchas claras era de hecho un microbio invisible ... un virus parásito de las bacterias". [16] D'Hérelle llamó al virus bacteriófago, un devorador de bacterias (del griego phagein que significa "devorar"). También registró un relato dramático de un hombre que sufría de disentería y que los bacteriófagos le devolvieron la buena salud.[17] Fue D'Herelle quien realizó muchas investigaciones sobre bacteriófagos e introdujo el concepto de terapia con fagos . [18]
Premios Nobel otorgados por la investigación de fagos
En 1969, Max Delbrück , Alfred Hershey y Salvador Luria recibieron el Premio Nobel de Fisiología o Medicina por sus descubrimientos sobre la replicación de virus y su estructura genética. [19] Específicamente, el trabajo de Hershey, como colaborador del experimento Hershey-Chase en 1952, proporcionó evidencia convincente de que el ADN, no las proteínas, era el material genético de la vida. Delbrück y Luria llevaron a cabo el experimento de Luria-Delbrück que demostró estadísticamente que las mutaciones en bacterias ocurren al azar y, por lo tanto, siguen principios darwinianos más que lamarckianos .
Usos
Terapia de fagos
Se descubrió que los fagos eran agentes antibacterianos y se utilizaron en la ex República Soviética de Georgia (iniciada allí por Giorgi Eliava con la ayuda del co-descubridor de bacteriófagos, Félix d'Herelle ) durante las décadas de 1920 y 1930 para el tratamiento de infecciones bacterianas. Tuvieron un uso generalizado, incluido el tratamiento de los soldados del Ejército Rojo . Sin embargo, fueron abandonados para uso general en Occidente por varias razones:
- Los antibióticos se descubrieron y comercializaron ampliamente. Eran más fáciles de preparar, almacenar y recetar.
- Se llevaron a cabo ensayos médicos de fagos, pero una falta básica de comprensión planteó dudas sobre la validez de estos ensayos. [20]
- La publicación de la investigación en la Unión Soviética se realizó principalmente en los idiomas ruso o georgiano y durante muchos años no se siguió a nivel internacional.
El uso de fagos ha continuado desde el final de la Guerra Fría en Rusia, [21] Georgia y en otras partes de Europa Central y Oriental. La primera regulada, aleatorizado, doble ciego ensayo clínico se informó en el Journal of Wound Care en junio de 2009, que evaluó la seguridad y eficacia de un cóctel bacteriófago infectado tratar las úlceras venosas de la pierna en pacientes humanos. [22] La FDA aprobó el estudio como ensayo clínico de fase I. Los resultados del estudio demostraron la seguridad de la aplicación terapéutica de bacteriófagos, pero no mostraron eficacia. Los autores explicaron que el uso de ciertos productos químicos que son parte del cuidado estándar de heridas (por ejemplo, lactoferrinao plata) puede haber interferido con la viabilidad del bacteriófago. [22] Poco después de eso, otro ensayo clínico controlado en Europa Occidental (tratamiento de infecciones de oído causadas por Pseudomonas aeruginosa ) se informó en la revista Clinical Otolaryngology en agosto de 2009. [23] El estudio concluye que las preparaciones de bacteriófagos eran seguras y eficaces para el tratamiento. de las infecciones crónicas del oído en humanos. Además, se han realizado numerosos ensayos clínicos en animales y otros ensayos clínicos experimentales que evalúan la eficacia de los bacteriófagos para diversas enfermedades, como quemaduras y heridas infectadas e infecciones pulmonares asociadas a la fibrosis quística, entre otras. [23]
Mientras tanto, los investigadores de bacteriófagos han estado desarrollando virus modificados para superar la resistencia a los antibióticos y manipulando los genes del fago responsables de codificar las enzimas que degradan la matriz de la biopelícula, las proteínas estructurales del fago y las enzimas responsables de la lisis de la pared celular bacteriana. [5] [6] [7] Se han obtenido resultados que muestran que los fagos T4 que son de tamaño pequeño y de cola corta pueden ser útiles para detectar E. coli en el cuerpo humano. [24]
La eficacia terapéutica de un cóctel de fagos se evaluó en un modelo de ratón con infección nasal de A. baumannii multirresistente (MDR) . Los ratones tratados con el cóctel de fagos mostraron una tasa de supervivencia 2,3 veces mayor que los no tratados siete días después de la infección. [25] En 2017, un paciente con un páncreas comprometido por MDR A. baumannii recibió varios antibióticos, a pesar de esto, la salud del paciente continuó deteriorándose durante un período de cuatro meses. Sin antibióticos efectivos, el paciente fue sometido a terapia con fagos utilizando un cóctel de fagos que contenía nueve fagos diferentes que habían demostrado ser efectivos contra MDR A. baumannii.. Una vez en esta terapia, la trayectoria clínica descendente del paciente se revirtió y recuperó la salud. [26]
D'Herelle "aprendió rápidamente que los bacteriófagos se encuentran dondequiera que prosperen las bacterias: en las alcantarillas, en los ríos que atrapan los desechos de las tuberías y en las heces de los pacientes convalecientes". [27] Esto incluye ríos que tradicionalmente se pensaba que tenían poderes curativos, incluido el río Ganges de la India . [28]
Otro
Industria alimentaria : los fagos se han utilizado cada vez más para proteger los productos alimenticios y prevenir las bacterias dañinas . [29] Desde 2006, la Administración de Drogas y Alimentos de los Estados Unidos (FDA) y el Departamento de Agricultura de los Estados Unidos (USDA) han aprobado varios productos bacteriófagos. LMP-102 (Intralytix) fue aprobado para el tratamiento de productos cárnicos y de aves de corral listos para el consumo (RTE). En ese mismo año, la FDA aprobó LISTEX (desarrollado y producido por Micreos ) usando bacteriófagos en el queso para matar la bacteria Listeria monocytogenes , con el fin de otorgarles el estado generalmente reconocido como seguro (GRAS). [30]En julio de 2007, se aprobó el uso del mismo bacteriófago en todos los productos alimenticios. [31] En 2011, el USDA confirmó que LISTEX es un auxiliar de procesamiento de etiqueta limpia y está incluido en el USDA. [32] La investigación en el campo de la seguridad alimentaria continúa para ver si los fagos líticos son una opción viable para controlar otros patógenos transmitidos por los alimentos en varios productos alimenticios.
Diagnóstico : en 2011, la FDA aprobó el primer producto a base de bacteriófagos para uso diagnóstico in vitro. [33] La prueba de hemocultivo KeyPath MRSA / MSSA utiliza un cóctel de bacteriófagos para detectar Staphylococcus aureus en hemocultivos positivos y determinar la resistencia o susceptibilidad a la meticilina . La prueba arroja resultados en aproximadamente cinco horas, en comparación con dos o tres días para los métodos de prueba de susceptibilidad e identificación microbiana estándar. Fue la primera prueba acelerada de susceptibilidad a los antibióticos aprobada por la FDA. [34]
Contrarrestar las armas biológicas y las toxinas : las agencias gubernamentales de Occidente llevan varios años buscando ayuda en Georgia y la ex Unión Soviética para explotar los fagos para contrarrestar las armas biológicas y las toxinas, como el ántrax y el botulismo . [35]Los desarrollos continúan entre los grupos de investigación en los EE. UU. Otros usos incluyen la aplicación por aspersión en horticultura para proteger las plantas y los productos vegetales de la descomposición y la propagación de enfermedades bacterianas. Otras aplicaciones de los bacteriófagos son como biocidas para superficies ambientales, por ejemplo, en hospitales, y como tratamientos preventivos para catéteres y dispositivos médicos antes de su uso en entornos clínicos. Actualmente existe la tecnología para que los fagos se apliquen a superficies secas, por ejemplo, uniformes, cortinas o incluso suturas para cirugía. Los ensayos clínicos publicados en Clinical Otolaryngology [23] muestran éxito en el tratamiento veterinario de perros con otitis .
El método de detección e identificación de bacterias SEPTIC utiliza la emisión de iones y su dinámica durante la infección por fagos y ofrece una alta especificidad y velocidad de detección. [36]
La presentación de fagos es un uso diferente de fagos que involucra una biblioteca de fagos con un péptido variable ligado a una proteína de superficie. Cada genoma de fago codifica la variante de la proteína que se muestra en su superficie (de ahí el nombre), lo que proporciona un vínculo entre la variante del péptido y su gen codificante. Los fagos variantes de la biblioteca pueden seleccionarse a través de su afinidad de unión a una molécula inmovilizada (por ejemplo, toxina del botulismo) para neutralizarla. Los fagos seleccionados unidos se pueden multiplicar reinfectando una cepa bacteriana susceptible, lo que les permite recuperar los péptidos codificados en ellos para su posterior estudio. [37]
Descubrimiento de fármacos antimicrobianos : las proteínas de los fagos a menudo tienen actividad antimicrobiana y pueden servir como guías para los peptidomiméticos , es decir, fármacos que imitan a los péptidos. [38] La tecnología de fagos-ligandos utiliza proteínas de fagos para diversas aplicaciones, como la unión de bacterias y componentes bacterianos (por ejemplo, endotoxinas ) y la lisis de bacterias. [39]
Investigación básica : los bacteriófagos son organismos modelo importantes para estudiar los principios de la evolución y la ecología . [40]
Detrimentos
Industria láctea
Los bacteriófagos presentes en el medio ambiente pueden provocar fallos en la fermentación de los cultivos iniciadores de queso . Para evitar esto, se pueden utilizar cultivos iniciadores de cepas mixtas y regímenes de rotación de cultivos. [41] Se ha estudiado la ingeniería genética de microbios de cultivo, especialmente Lactococcus lactis y Streptococcus thermophilus , para el análisis genético y la modificación para mejorar la resistencia a los fagos . Esto se ha centrado especialmente en modificaciones de plásmidos y cromosomas recombinantes . [42] [29]
Replicación
Los bacteriófagos pueden tener un ciclo lítico o un ciclo lisogénico . Con fagos líticos como el fago T4 , las células bacterianas se abren (lisan) y se destruyen después de la replicación inmediata del virión. Tan pronto como se destruye la célula, la progenie del fago puede encontrar nuevos huéspedes para infectar. Los fagos líticos son más adecuados para la terapia con fagos . Algunos fagos líticos experimentan un fenómeno conocido como inhibición de la lisis, donde la progenie completa del fago no se lisará inmediatamente fuera de la célula si las concentraciones extracelulares del fago son altas. Este mecanismo no es idéntico al de los fagos templados que se vuelven inactivos y, por lo general, es temporal.
Por el contrario, el ciclo lisogénico no da como resultado la lisis inmediata de la célula huésped. Los fagos capaces de sufrir lisogenia se conocen como fagos templados . Su genoma viral se integrará con el ADN del huésped y se replicará junto con él, de manera relativamente inofensiva, o incluso puede establecerse como un plásmido . El virus permanece inactivo hasta que las condiciones del hospedador se deterioran, quizás debido al agotamiento de los nutrientes, luego, los fagos endógenos (conocidos como profagos) se vuelven activos. En este punto, inician el ciclo reproductivo, lo que resulta en la lisis de la célula huésped. Como el ciclo lisogénico permite que la célula huésped continúe sobreviviendo y reproduciéndose, el virus se replica en toda la descendencia de la célula. Un ejemplo de bacteriófago que se sabe que sigue el ciclo lisogénico y el ciclo lítico es el fago lambda de E. coli. [43]
A veces, los profagos pueden proporcionar beneficios a la bacteria huésped mientras están inactivos al agregar nuevas funciones al genoma bacteriano , en un fenómeno llamado conversión lisogénica . Algunos ejemplos son la conversión de cepas inofensivas de Corynebacterium diphtheriae o Vibrio cholerae por bacteriófagos, en cepas altamente virulentas que causan difteria o cólera , respectivamente. [44] [45] Se han propuesto estrategias para combatir ciertas infecciones bacterianas dirigidas a estos profagos que codifican toxinas. [46]
Apego y penetración
Las células bacterianas están protegidas por una pared celular de polisacáridos , que son importantes factores de virulencia que protegen a las células bacterianas contra las defensas inmunitarias del huésped y los antibióticos . [47] Para ingresar a una célula huésped, los bacteriófagos se unen a receptores específicos en la superficie de las bacterias, incluidos los lipopolisacáridos , los ácidos teicoicos , las proteínas o incluso los flagelos.. Esta especificidad significa que un bacteriófago puede infectar solo ciertas bacterias portadoras de receptores a los que pueden unirse, lo que a su vez, determina el rango de hospedadores del fago. Las enzimas que degradan polisacáridos, como las endolisinas, son proteínas asociadas a viriones que degradan enzimáticamente la capa externa capsular de sus huéspedes, en el paso inicial de un proceso de infección por fagos estrechamente programado. Las condiciones de crecimiento del hospedador también influyen en la capacidad del fago para unirse e invadirlo. [48] Como los viriones de fagos no se mueven de forma independiente, deben depender de encuentros aleatorios con los receptores correctos cuando están en solución, como sangre, circulación linfática, irrigación, agua del suelo, etc.
Los bacteriófagos de miovirus utilizan un movimiento similar a una jeringa hipodérmica para inyectar su material genético en la célula. Después de contactar con el receptor apropiado, las fibras de la cola se flexionan para acercar la placa base a la superficie de la celda. Esto se conoce como encuadernación reversible. Una vez adherida por completo, se inicia la unión irreversible y la cola se contrae, posiblemente con la ayuda de ATP , presente en la cola, [6] inyectando material genético a través de la membrana bacteriana. [49]La inyección se logra a través de una especie de movimiento de flexión en el eje yendo hacia un lado, contrayéndose más cerca de la celda y empujando hacia arriba. Los podovirus carecen de una vaina de la cola alargada como la de un miovirus, por lo que, en cambio, usan sus pequeñas fibras de la cola en forma de dientes para degradar una parte de la membrana celular antes de insertar su material genético.
Síntesis de proteínas y ácido nucleico.
En cuestión de minutos, los ribosomas bacterianos comienzan a traducir el ARNm viral en proteína. Para los fagos basados en ARN , la replicasa de ARN se sintetiza al principio del proceso. Las proteínas modifican la ARN polimerasa bacteriana para que transcriba preferentemente el ARNm viral. La síntesis normal de proteínas y ácidos nucleicos del huésped se interrumpe y, en su lugar, se ve obligado a fabricar productos virales. Estos productos pasan a formar parte de nuevos viriones dentro de la célula, proteínas auxiliares que contribuyen al ensamblaje de nuevos viriones o proteínas involucradas en la lisis celular . En 1972, Walter Fiers ( Universidad de Gante , Bélgica) fue el primero en establecer la secuencia completa de nucleótidos de un gen y, en 1976, del genoma viral del bacteriófago MS2 . [50] Algunos bacteriófagos de dsDNA codifican proteínas ribosómicas, que se cree que modulan la traducción de proteínas durante la infección por fagos. [51]
Ensamblaje de Virion
En el caso del fago T4 , la construcción de nuevas partículas de virus implica la asistencia de proteínas auxiliares que actúan catalíticamente durante la morfogénesis del fago . [52] Las placas base se ensamblan primero, y las colas se construyen sobre ellas después. Las cápsides de la cabeza, construidas por separado, se ensamblarán espontáneamente con las colas. Durante el ensamblaje del virión del fago T4 , las proteínas morfogenéticas codificadas por los genes del fago interactúan entre sí en una secuencia característica. Mantener un equilibrio apropiado en las cantidades de cada una de estas proteínas producidas durante la infección viral parece ser crítico para la morfogénesis normal del fago T4 . [53]El ADN se empaqueta de manera eficiente dentro de las cabezas. [54] Todo el proceso dura unos 15 minutos.
Liberación de viriones
Los fagos pueden liberarse por lisis celular, por extrusión o, en algunos casos, por gemación. La lisis, por fagos con cola, se logra mediante una enzima llamada endolisina , que ataca y descompone el peptidoglicano de la pared celular . Un tipo de fago completamente diferente, el fago filamentoso , hace que la célula huésped secrete continuamente nuevas partículas de virus. Los viriones liberados se describen como libres y, a menos que sean defectuosos, pueden infectar una nueva bacteria. La gemación está asociada con ciertos fagos de Mycoplasma . En contraste con la liberación de viriones, los fagos que muestran un ciclo lisogénico no matan al huésped, sino que se convierten en residentes a largo plazo como profagos .
Comunicación
La investigación en 2017 reveló que el bacteriófago Φ3T produce una proteína viral corta que indica a otros bacteriófagos que permanezcan inactivos en lugar de matar a la bacteria huésped. Arbitrium es el nombre que le dieron a esta proteína los investigadores que la descubrieron. [55] [56]
Estructura del genoma
Dados los millones de fagos diferentes en el medio ambiente, los genomas de los fagos se presentan en una variedad de formas y tamaños. Los fagos de ARN como MS2 tienen los genomas más pequeños, de solo unas pocas kilobases. Sin embargo, algunos fagos de ADN como el T4 pueden tener genomas grandes con cientos de genes; el tamaño y la forma de la cápside varía junto con el tamaño del genoma. [57] Los genomas de bacteriófagos más grandes alcanzan un tamaño de 735 kb. [58]
Los genomas de los bacteriófagos pueden tener un gran mosaico , es decir, el genoma de muchas especies de fagos parece estar compuesto por numerosos módulos individuales. Estos módulos se pueden encontrar en otras especies de fagos en diferentes disposiciones. Los micobacteriófagos , bacteriófagos con huéspedes micobacterianos , han proporcionado excelentes ejemplos de este mosaicismo. En estos micobacteriófagos, surtido genético puede ser el resultado de repetidos casos de recombinación específica de sitio y recombinación ilegítima (el resultado de fago adquisición genoma de secuencias genéticas huésped bacterianas). [60]Los mecanismos evolutivos que dan forma a los genomas de los virus bacterianos varían entre diferentes familias y dependen del tipo de ácido nucleico, las características de la estructura del virión y el modo del ciclo de vida viral. [61]
Algunos fagos de roseobacter marino contienen desoxiuridina (dU) en lugar de desoxitimidina (dT) en su ADN genómico. Existe alguna evidencia de que este componente inusual es un mecanismo para evadir los mecanismos de defensa bacterianos como las endonucleasas de restricción y los sistemas CRISPR / Cas que evolucionaron para reconocer y escindir secuencias dentro del fago invasor, inactivándolos así. Se sabe desde hace mucho tiempo que otros fagos utilizan nucleótidos inusuales. En 1963, Takahashi y Marmur identificaron un fago de Bacillus que tenía dU sustituyendo a dT en su genoma, [62] y en 1977, Kirnos et al. identificó un cianófago que contiene 2-aminoadenina (Z) en lugar de adenina (A).[63]
Biologia de sistemas
El campo de la biología de sistemas investiga las complejas redes de interacciones dentro de un organismo, generalmente utilizando herramientas computacionales y modelos. [64] Por ejemplo, un genoma de fago que entra en una célula huésped bacteriana puede expresar cientos de proteínas de fagos que afectarán la expresión de numerosos genes del huésped o el metabolismo del huésped . Todas estas interacciones complejas se pueden describir y simular en modelos informáticos. [64]
Por ejemplo, la infección de Pseudomonas aeruginosa por el fago templado PaP3 cambió la expresión del 38% (2160/5633) de los genes de su huésped. Muchos de estos efectos son probablemente indirectos, de ahí que el desafío sea identificar las interacciones directas entre bacterias y fagos. [sesenta y cinco]
Se han realizado varios intentos para cartografiar las interacciones proteína-proteína entre los fagos y su huésped. Por ejemplo, se descubrió que el bacteriófago lambda interactúa con su huésped, E. coli , mediante decenas de interacciones. Una vez más, la importancia de muchas de estas interacciones sigue sin estar clara, pero estos estudios sugieren que lo más probable es que haya varias interacciones clave y muchas interacciones indirectas cuyo papel permanece sin caracterizar. [66]
En el ambiente
La metagenómica ha permitido la detección en agua de bacteriófagos que antes no era posible. [67]
Además, los bacteriófagos se han utilizado en la modelización y el rastreo hidrológico en sistemas fluviales , especialmente donde se producen interacciones entre aguas superficiales y subterráneas . Se prefiere el uso de fagos al marcador de colorante más convencional porque se absorben significativamente menos cuando pasan a través de aguas subterráneas y se detectan fácilmente a concentraciones muy bajas. [68] El agua no contaminada puede contener aproximadamente 2 × 10 8 bacteriófagos por ml. [69]
Se cree que los bacteriófagos contribuyen en gran medida a la transferencia horizontal de genes en entornos naturales, principalmente mediante transducción , pero también mediante transformación . [70] Los estudios basados en la metagenómica también han revelado que los viromas de una variedad de entornos albergan genes de resistencia a los antibióticos, incluidos aquellos que podrían conferir resistencia a múltiples fármacos . [71]
Inhumanos
Aunque los fagos no infectan a los humanos, existen innumerables partículas de fagos en el cuerpo humano, dado nuestro extenso microbioma . Nuestra población de fagos se ha denominado fageoma humano, incluido el "fageoma intestinal sano" (HGP) y el "fageoma humano enfermo" (DHP). [72] Se ha estimado que el fageoma activo de un ser humano sano (es decir, profago integrado que se replica activamente en lugar de que no se replica ) comprende decenas a miles de virus diferentes. [73] El papel de los bacteriófagos en la salud humana sigue siendo poco conocido, pero los estudios preliminares indican que los bacteriófagos comunes se encuentran en promedio en el 62% de las personas sanas, mientras que su prevalencia se redujo en un 42% y un 54% en promedio en pacientes concolitis ulcerosa (CU) y enfermedad de Crohn (EC). [72] Esto indica que es posible que se requieran fagos para controlar la población de bacterias tanto normales como patógenas en el microbioma intestinal humano .
Bacteriófago comúnmente estudiado
Entre los innumerables fagos, solo unos pocos se han estudiado en detalle, incluidos algunos fagos de importancia histórica que se descubrieron en los primeros días de la genética microbiana. Estos, especialmente el fago T, ayudaron a descubrir principios importantes de la estructura y función de los genes.
- 186 fagos
- fago λ
- Φ6 fago
- Φ29 fago
- ΦX174
- Bacteriófago φCb5
- Fago G4
- Fago M13
- Fago MS2 (de 23 a 28 nm de tamaño) [74]
- Fago N4
- Fago P1
- Fago P2
- Fago P4
- Fago R17
- Fago T2
- Fago T4 ( genoma de 169 kpb , [75] 200 nm de largo [76] )
- Fago T7
- Fago T12
Ver también
- Bacterívoro
- CrAssphage
- CRISPR
- Virus de ADN
- Ecología de fagos
- Monografías de fagos (una lista completa de fagos y monografías asociadas a fagos, desde 1921 hasta el presente)
- Fagémido
- Polifago
- Virus de ARN
- Transducción
- Viriome
- Virofago , virus que infectan a otros virus
Referencias
- ↑ Padilla-Sanchez V (2021). "Modelo estructural del bacteriófago T4" . WikiJournal of Science . 4 (1): 5. doi : 10.15347 / WJS / 2021.005 .
- ^ a b McGrath S, van Sinderen D, eds. (2007). Bacteriófago: Genética y Biología Molecular (1ª ed.). Prensa Académica Caister. ISBN 978-1-904455-14-1.
- ^ LaFee S, Buschman H (25 de abril de 2017). "La nueva terapia con fagos salva al paciente con una infección bacteriana resistente a múltiples fármacos" . UC Health - UC San Diego . Consultado el 13 de mayo de 2018 .
- ^ Suttle CA (septiembre de 2005). "Virus en el mar". Naturaleza . 437 (7057): 356–61. Código Bibliográfico : 2005Natur.437..356S . doi : 10.1038 / nature04160 . PMID 16163346 . S2CID 4370363 .
- ↑ a b Wommack KE, Colwell RR (marzo de 2000). "Virioplancton: virus en ecosistemas acuáticos" . Revisiones de Microbiología y Biología Molecular . 64 (1): 69-114. doi : 10.1128 / MMBR.64.1.69-114.2000 . PMC 98987 . PMID 10704475 .
- ↑ a b c m Prescott L (1993). Microbiología . Wm. C. Editores Brown. ISBN 0-697-01372-3.
- ↑ a b Bunting J (1997). "El virus que cura" . BBC Horizon . BBC Worldwide Ltd. OCLC 224991186 . - Documental sobre la historia de la medicina fagosa en Rusia y Occidente.
- ^ Borrell B, Fishchetti V (agosto de 2012). "Charla de ciencia: factor fago". Scientific American . págs. 80–83. JSTOR 26016042 .
- ^ Keen EC (2012). "Terapia de fagos: concepto para curar" . Fronteras en microbiología . 3 : 238. doi : 10.3389 / fmicb.2012.00238 . PMC 3400130 . PMID 22833738 .
- ^ Sweere JM, Van Belleghem JD, Ishak H, Bach MS, Popescu M, Sunkari V, et al. (Marzo de 2019). "Los bacteriófagos desencadenan la inmunidad antiviral y previenen la eliminación de la infección bacteriana" . Ciencia . 363 (6434): eaat9691. doi : 10.1126 / science.aat9691 . PMC 6656896 . PMID 30923196 .
- ^ Laanto E, Mäntynen S, De Colibus L, Marjakangas J, Gillum A, Stuart DI, et al. (Agosto de 2017). "Virus encontrado en un lago boreal vincula virus ssDNA y dsDNA" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 114 (31): 8378–8383. doi : 10.1073 / pnas.1703834114 . PMC 5547622 . PMID 28716906 .
- ^ Krishnamurthy SR, Wang D (marzo de 2018). "Amplia conservación de sitios de unión ribosomal procarióticos en picobirnavirus conocidos y nuevos" . Virología . 516 : 108-114. doi : 10.1016 / j.virol.2018.01.006 . PMID 29346073 .
- ^ Callanan J, Stockdale SR, Adriaenssens EM, Kuhn JH (enero de 2021). "Cambiar el nombre de una clase (Leviviricetes - antes Allassoviricetes), cambiar el nombre de un orden (Norzivirales - antes Levivirales), crear un nuevo orden (Timlovirales) y ampliar la clase a un total de seis familias, 420 géneros y 883 especies" . ResearchGate . doi : 10.13140 / RG.2.2.25363.40481 .
- ↑ Hankin EH (1896). "L'action bactericide des eaux de la Jumna et du Gange sur le vibrion du cholera" . Annales de l'Institut Pasteur (en francés). 10 : 511-23.
- ^ Twort FW (1915). "Una investigación sobre la naturaleza de los virus ultramicroscópicos" . The Lancet . 186 (4814): 1241–43. doi : 10.1016 / S0140-6736 (01) 20383-3 .
- ↑ d'Hérelles F (1917). "Sur un microbio invisible antagonista des bacilles dysentériques" (PDF) . Comptes Rendus de l'Académie des Sciences de Paris . 165 : 373–5. Archivado (PDF) desde el original el 11 de mayo de 2011 . Consultado el 5 de septiembre de 2010 .
- ↑ d'Hérelles F (1949). "El bacteriófago" (PDF) . Noticias de ciencia . 14 : 44–59 . Consultado el 5 de septiembre de 2010 .
- ^ Keen EC (diciembre de 2012). "Felix d'Herelle y nuestro futuro microbiano". Microbiología del futuro . 7 (12): 1337–9. doi : 10.2217 / fmb.12.115 . PMID 23231482 .
- ^ "El Premio Nobel de Fisiología o Medicina 1969" . Fundación Nobel . Consultado el 28 de julio de 2007 .
- ^ Kutter E, De Vos D, Gvasalia G, Alavidze Z, Gogokhia L, Kuhl S, Abedon ST (enero de 2010). "Terapia de fagos en la práctica clínica: tratamiento de infecciones humanas". Biotecnología Farmacéutica Actual . 11 (1): 69–86. doi : 10.2174 / 138920110790725401 . PMID 20214609 . S2CID 31626252 .
- ^ Golovin S (2017). "Бактериофаги: убийцы в роли спасителей" [Bacteriófagos: asesinos como salvadores]. Наука и жизнь [ Nauka I Zhizn (Ciencia y vida) ] (en ruso) (6): 26–33.
- ↑ a b Rhoads DD, Wolcott RD, Kuskowski MA, Wolcott BM, Ward LS, Sulakvelidze A (junio de 2009). "Terapia con bacteriófagos de úlceras venosas de la pierna en humanos: resultados de un ensayo de seguridad de fase I". Revista de cuidado de heridas . 18 (6): 237–8, 240–3. doi : 10.12968 / jowc.2009.18.6.42801 . PMID 19661847 .
- ↑ a b c Wright A, Hawkins CH, Anggård EE, Harper DR (agosto de 2009). "Un ensayo clínico controlado de una preparación de bacteriófagos terapéuticos en la otitis crónica por Pseudomonas aeruginosa resistente a los antibióticos; un informe preliminar de eficacia" . Otorrinolaringología clínica . 34 (4): 349–57. doi : 10.1111 / j.1749-4486.2009.01973.x . PMID 19673983 .
- ^ Robu ME, Larson JD, Nasevicius A, Beiraghi S, Brenner C, Farber SA, Ekker SC (mayo de 2007). "Activación de p53 por tecnologías de derribo" . PLOS Genetics . 3 (5): e78. doi : 10.1371 / journal.pgen.0030078 . PMC 1877875 . PMID 17530925 .
- ^ Cha K, Oh HK, Jang JY, Jo Y, Kim WK, Ha GU, et al. (10 de abril de 2018). "Caracterización de dos nuevos bacteriófagos que infectan Acinetobacter baumannii multirresistente (MDR) y evaluación de su eficacia terapéutica in vivo " . Fronteras en microbiología . 9 : 696. doi : 10.3389 / fmicb.2018.00696 . PMC 5932359 . PMID 29755420 .
- ^ Schooley RT, Biswas B, Gill JJ, Hernandez-Morales A, Lancaster J, Lessor L, et al. (Octubre de 2017). "Desarrollo y uso de cócteles terapéuticos personalizados basados en bacteriófagos para tratar a un paciente con una infección diseminada por Acinetobacter baumannii resistente" . Agentes antimicrobianos y quimioterapia . 61 (10). doi : 10.1128 / AAC.00954-17 . PMC 5610518 . PMID 28807909 .
- ^ Kuchment A (2012), La cura olvidada: el pasado y el futuro de la terapia con fagos , Springer, p. 11, ISBN 978-1-4614-0250-3
- ^ Deresinski S (abril de 2009). "Terapia con bacteriófagos: explotación de pulgas más pequeñas" . Enfermedades Clínicas Infecciosas . 48 (8): 1096–101. doi : 10.1086 / 597405 . PMID 19275495 .
- ↑ a b O'Sullivan L, Bolton D, McAuliffe O, Coffey A (marzo de 2019). "Bacteriófagos en aplicaciones alimentarias: de enemigo a amigo". Revisión anual de ciencia y tecnología de los alimentos . Revisiones anuales . 10 (1): 151-172. doi : 10.1146 / annurev-food-032818-121747 . PMID 30633564 .
- ^ FDA de EE. UU. / CFSAN: Carta de respuesta de la agencia, Aviso GRAS No. 000198
- ^ (FDA / CFSAN de EE. UU .: Carta de respuesta de la agencia, Aviso GRAS No. 000218)
- ^ "Directiva 7120 del FSIS: ingredientes seguros y adecuados utilizados en la producción de productos cárnicos, de aves de corral y de huevo" (PDF) . Servicio de Inspección y Seguridad Alimentaria . Washington, DC: Departamento de Agricultura de los Estados Unidos. Archivado desde el original (PDF) el 18 de octubre de 2011.
- ^ "Notificación previa a la comercialización de la FDA 510 (k)" . Administración de Drogas y Alimentos de EE. UU.
- ^ "La FDA autoriza la primera prueba para diagnosticar y distinguir rápidamente MRSA y MSSA" . Administración de Drogas y Alimentos de EE. UU. 6 de mayo de 2011. Archivado desde el original el 5 de enero de 2012.
- ^ Vaisman D (25 de mayo de 2007). "Estudiando ántrax en un laboratorio de la era soviética - con financiación occidental" . The New York Times .
- ↑ Dobozi-King M, Seo S, Kim JU, Young R, Cheng M, Kish LB (2005). "Detección e identificación rápida de bacterias: detección de cascada de iones activados por fagos (SEPTIC)" (PDF) . Revista de Física y Química Biológicas . 5 : 3-7. doi : 10.4024 / 1050501.jbpc.05.01 . Archivado desde el original (PDF) el 26 de septiembre de 2018 . Consultado el 19 de diciembre de 2016 .
- ^ Smith GP, Petrenko VA (abril de 1997). "Visualización de fagos". Revisiones químicas . 97 (2): 391–410. doi : 10.1021 / cr960065d . PMID 11848876 .
- ^ Liu J, Dehbi M, Moeck G, Arhin F, Bauda P, Bergeron D, et al. (Febrero de 2004). "Descubrimiento de fármacos antimicrobianos a través de la genómica de bacteriófagos". Biotecnología de la naturaleza . 22 (2): 185–91. doi : 10.1038 / nbt932 . PMID 14716317 . S2CID 9905115 .
- ^ "Antecedentes tecnológicos Tecnología de ligando de fagos" . bioMérieux .
- ^ Keen EC (enero de 2014). "Compensaciones en historias de vida de bacteriófagos" . Bacteriófago . 4 (1): e28365. doi : 10.4161 / bact.28365 . PMC 3942329 . PMID 24616839 .
- ^ Atamer Z, Samtlebe M, Neve H, J Heller K, Hinrichs J (16 de julio de 2013). "Revisión: eliminación de bacteriófagos en suero y productos de suero" . Fronteras en microbiología . 4 : 191. doi : 10.3389 / fmicb.2013.00191 . PMC 3712493 . PMID 23882262 .
- ^ Coffey A, Ross RP (agosto de 2002). "Sistemas de resistencia a bacteriófagos en cepas iniciadoras de lácteos: análisis molecular a la aplicación". Antonie van Leeuwenhoek . Springer . 82 (1–4): 303–21. doi : 10.1023 / a: 1020639717181 . PMID 12369198 . S2CID 7217985 .
- ^ Mason KA, Losos JB, Singer SR, Raven PH, Johnson GB (2011). Biología . Nueva York: McGraw-Hill. pag. 533. ISBN 978-0-07-893649-4.
- ^ Mokrousov I (enero de 2009). "Corynebacterium diphtheriae: diversidad del genoma, estructura de la población y perspectivas de genotipado". Infección, Genética y Evolución . 9 (1): 1-15. doi : 10.1016 / j.meegid.2008.09.011 . PMID 19007916 .
- ^ Charles RC, Ryan ET (octubre de 2011). "Cólera en el siglo XXI". Opinión Actual en Enfermedades Infecciosas . 24 (5): 472–7. doi : 10.1097 / QCO.0b013e32834a88af . PMID 21799407 . S2CID 6907842 .
- ^ Keen EC (diciembre de 2012). "Paradigmas de patogénesis: dirigidos a los elementos genéticos móviles de la enfermedad" . Fronteras en microbiología celular e infecciosa . 2 : 161. doi : 10.3389 / fcimb.2012.00161 . PMC 3522046 . PMID 23248780 .
- ↑ Drulis-Kawa Z, Majkowska-Skrobek G, Maciejewska B (2015). "Bacteriófagos y proteínas derivadas de fagos - enfoques de aplicación" . Química Medicinal Actual . 22 (14): 1757–73. doi : 10.2174 / 0929867322666150209152851 . PMC 4468916 . PMID 25666799 .
- ^ Gabashvili IS, Khan SA, Hayes SJ, Serwer P (octubre de 1997). "Polimorfismo del bacteriófago T7". Revista de Biología Molecular . 273 (3): 658–67. doi : 10.1006 / jmbi.1997.1353 . PMID 9356254 .
- ^ Maghsoodi A, Chatterjee A, Andricioaei I, Perkins NC (diciembre de 2019). "Cómo funciona la maquinaria de inyección del fago T4, incluida la energía, las fuerzas y la vía dinámica" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 116 (50): 25097-25105. doi : 10.1073 / pnas.1909298116 . PMC 6911207 . PMID 31767752 .
- ^ Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, et al. (Abril de 1976). "Secuencia completa de nucleótidos del ARN del bacteriófago MS2: estructura primaria y secundaria del gen de la replicasa". Naturaleza . 260 (5551): 500–7. Código Bibliográfico : 1976Natur.260..500F . doi : 10.1038 / 260500a0 . PMID 1264203 . S2CID 4289674 .
- ^ Mizuno CM, Guyomar C, Roux S, Lavigne R, Rodriguez-Valera F, Sullivan MB, et al. (Febrero de 2019). "Numerosos virus cultivados y no cultivados codifican proteínas ribosómicas" . Comunicaciones de la naturaleza . 10 (1): 752. Bibcode : 2019NatCo..10..752M . doi : 10.1038 / s41467-019-08672-6 . PMC 6375957 . PMID 30765709 .
- ^ Snustad DP (agosto de 1968). "Interacciones de dominancia en células de Escherichia coli infectadas de forma mixta con mutantes de tipo salvaje y ámbar del bacteriófago T4D y sus posibles implicaciones en cuanto al tipo de función del producto genético: catalítica frente a estequiométrica". Virología . 35 (4): 550–63. doi : 10.1016 / 0042-6822 (68) 90285-7 . PMID 4878023 .
- ^ Floor E (febrero de 1970). "Interacción de genes morfogenéticos del bacteriófago T4". Revista de Biología Molecular . 47 (3): 293-306. doi : 10.1016 / 0022-2836 (70) 90303-7 . PMID 4907266 .
- ^ Petrov AS, Harvey SC (julio de 2008). "Empaquetado de ADN de doble hélice en cápsides virales: estructuras, fuerzas y energéticas" . Revista biofísica . 95 (2): 497–502. Código bibliográfico : 2008BpJ .... 95..497P . doi : 10.1529 / biophysj.108.131797 . PMC 2440449 . PMID 18487310 .
- ^ Callaway E (2017). "¿Hablas virus? Fagos atrapados enviando mensajes químicos" . Naturaleza . doi : 10.1038 / nature.2017.21313 .
- ^ Erez Z, Steinberger-Levy I, Shamir M, Doron S, Stokar-Avihail A, Peleg Y, et al. (Enero de 2017). "La comunicación entre virus guía las decisiones de lisis-lisogenia" . Naturaleza . 541 (7638): 488–493. Código Bib : 2017Natur.541..488E . doi : 10.1038 / nature21049 . PMC 5378303 . PMID 28099413 .
- ^ Negro LW, Thomas JA (2012). "Estructura del genoma condensado". Máquinas moleculares virales . Avances en Medicina y Biología Experimental. 726 . págs. 469–87. doi : 10.1007 / 978-1-4614-0980-9_21 . ISBN 978-1-4614-0979-3. PMC 3559133 . PMID 22297527 .
- ^ Al-Shayeb B, Sachdeva R, Chen LX, Ward F, Munk P, Devoto A, et al. (Febrero de 2020). "Clados de fagos enormes de todos los ecosistemas de la Tierra" . Naturaleza . 578 (7795): 425–431. Código Bib : 2020Natur.578..425A . doi : 10.1038 / s41586-020-2007-4 . PMC 7162821 . PMID 32051592 .
- ^ Häuser R, Blasche S, Dokland T, Haggård-Ljungquist E, von Brunn A, Salas M, et al. (2012). "Interacciones proteína-proteína bacteriófago" . Avances en la investigación de virus . 83 : 219–98. doi : 10.1016 / B978-0-12-394438-2.00006-2 . ISBN 9780123944382. PMC 3461333 . PMID 22748812 .
- ^ Morris P, Marinelli LJ, Jacobs-Sera D, Hendrix RW, Hatfull GF (marzo de 2008). "Caracterización genómica del micobacteriófago Giles: evidencia de la adquisición del ADN del huésped por fagos por recombinación ilegítima" . Revista de bacteriología . 190 (6): 2172–82. doi : 10.1128 / JB.01657-07 . PMC 2258872 . PMID 18178732 .
- ^ Krupovic M, Prangishvili D, Hendrix RW, Bamford DH (diciembre de 2011). "Genómica de virus bacterianos y arqueales: dinámica dentro de la virosfera procariota" . Revisiones de Microbiología y Biología Molecular . 75 (4): 610–35. doi : 10.1128 / MMBR.00011-11 . PMC 3232739 . PMID 22126996 .
- ^ Takahashi I, Marmur J (febrero de 1963). "Reemplazo de ácido timidílico por ácido desoxiuridílico en el ácido desoxirribonucleico de un fago transductor de Bacillus subtilis". Naturaleza . 197 (4869): 794–5. Código Bibliográfico : 1963Natur.197..794T . doi : 10.1038 / 197794a0 . PMID 13980287 . S2CID 4166988 .
- ^ Kirnos MD, Khudyakov IY, Alexandrushkina NI, Vanyushin BF (noviembre de 1977). "La 2-aminoadenina es una adenina que sustituye a una base en el ADN del cianófago S-2L". Naturaleza . 270 (5635): 369–70. Código bibliográfico : 1977Natur.270..369K . doi : 10.1038 / 270369a0 . PMID 413053 . S2CID 4177449 .
- ↑ a b Klipp E (2009). Biología de sistemas: un libro de texto . Weinheim: Wiley-VCH. ISBN 978-3-527-31874-2. OCLC 288986435 .
- ^ Zhao X, Chen C, Shen W, Huang G, Le S, Lu S, et al. (Enero de 2016). "Análisis transcriptómico global de interacciones entre Pseudomonas aeruginosa y bacteriófago PaP3" . Informes científicos . 6 : 19237. Código Bibliográfico : 2016NatSR ... 619237Z . doi : 10.1038 / srep19237 . PMC 4707531 . PMID 26750429 .
- ^ Blasche S, Wuchty S, Rajagopala SV, Uetz P (diciembre de 2013). "La red de interacción de proteínas del bacteriófago lambda con su huésped, Escherichia coli" . Revista de Virología . 87 (23): 12745–55. doi : 10.1128 / JVI.02495-13 . PMC 3838138 . PMID 24049175 .
- ^ Breitbart M , Salamon P, Andresen B, Mahaffy JM, Segall AM, Mead D, et al. (Octubre de 2002). "Análisis genómico de comunidades virales marinas no cultivadas" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (22): 14250–5. Código bibliográfico : 2002PNAS ... 9914250B . doi : 10.1073 / pnas.202488399 . PMC 137870 . PMID 12384570 .
- ^ Martín C (1988). "La aplicación de técnicas de trazadores de bacteriófagos en el agua del suroeste". Revista Agua y Medio Ambiente . 2 (6): 638–642. doi : 10.1111 / j.1747-6593.1988.tb01352.x .
- ^ Bergh O, Børsheim KY, Bratbak G, Heldal M (agosto de 1989). "Gran abundancia de virus encontrados en ambientes acuáticos". Naturaleza . 340 (6233): 467–8. Código Bibliográfico : 1989Natur.340..467B . doi : 10.1038 / 340467a0 . PMID 2755508 . S2CID 4271861 .
- ^ Keen EC, Bliskovsky VV, Malagon F, Baker JD, Prince JS, Klaus JS, Adhya SL (enero de 2017). Los bacteriófagos "novedosos" superpropagadores "promueven la transferencia horizontal de genes por transformación" . mBio . 8 (1): e02115–16. doi : 10.1128 / mBio.02115-16 . PMC 5241400 . PMID 28096488 .
- ^ Lekunberri I, Subirats J, Borrego CM, Balcázar JL (enero de 2017). "Explorando la contribución de los bacteriófagos a la resistencia a los antibióticos". Contaminación ambiental . 220 (Pt B): 981–984. doi : 10.1016 / j.envpol.2016.11.059 . hdl : 10256/14115 . PMID 27890586 .
- ↑ a b Manrique P, Bolduc B, Walk ST, van der Oost J, de Vos WM, Young MJ (septiembre de 2016). "Fageoma del intestino humano sano" . Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 113 (37): 10400-5. doi : 10.1073 / pnas.1601060113 . PMC 5027468 . PMID 27573828 .
- ^ Minot S, Sinha R, Chen J, Li H, Keilbaugh SA, Wu GD, et al. (Octubre de 2011). "El viroma del intestino humano: variación interindividual y respuesta dinámica a la dieta" . Investigación del genoma . 21 (10): 1616–25. doi : 10.1101 / gr.122705.111 . PMC 3202279 . PMID 21880779 .
- ^ Strauss JH, Sinsheimer RL (julio de 1963). "Purificación y propiedades del bacteriófago MS2 y de su ácido ribonucleico". Revista de Biología Molecular . 7 (1): 43–54. doi : 10.1016 / S0022-2836 (63) 80017-0 . PMID 13978804 .
- ^ Miller ES, Kutter E, Mosig G, Arisaka F, Kunisawa T, Rüger W (marzo de 2003). "Genoma del bacteriófago T4" . Revisiones de Microbiología y Biología Molecular . 67 (1): 86-156, índice. doi : 10.1128 / MMBR.67.1.86-156.2003 . PMC 150520 . PMID 12626685 .
- ^ Ackermann HW, Krisch HM (6 de abril de 2014). "Un catálogo de bacteriófagos de tipo T4". Archivos de Virología . 142 (12): 2329–45. doi : 10.1007 / s007050050246 . PMID 9672598 . S2CID 39369249 .
Bibliografía
- Hauser AR, Mecsas J, Moir DT (julio de 2016). "Más allá de los antibióticos: nuevos enfoques terapéuticos para las infecciones bacterianas" . Enfermedades Clínicas Infecciosas . 63 (1): 89–95. doi : 10.1093 / cid / ciw200 . PMC 4901866 . PMID 27025826 .
- Strathdee S, Patterson T (2019). El depredador perfecto . Libros Hachette . ISBN 978-0316418089.
enlaces externos
| Wikimedia Commons tiene medios relacionados con bacteriófagos . |
| Wikiquote tiene citas relacionadas con: Bacteriófago |
- Häusler T (2006). Virus vs superbacterias: ¿una solución a la crisis de los antibióticos? . Londres: Macmillan. ISBN 978-1-4039-8764-8.
- Abedon ST. "El Grupo de Ecología de Bacteriófagos" . La Universidad Estatal de Ohio. Archivado desde el original el 3 de junio de 2013.
- Tourterel C, Blouin Y. "Ilustraciones y genómica de bacteriófagos" . Sitio web del fago de Orsay .
- "QuipStories: Los bacteriófagos se afianzan en sus presas" (PDF) . PDBe .
- Flatow I (abril de 2008). "Uso de virus 'Phage' para ayudar a combatir la infección" . Podcast de Science Friday . NPR. Archivado desde el original el 17 de abril de 2008.
- "Animación de un bacteriófago T4 científicamente correcto dirigido a la bacteria E. coli" . YouTube.
- "Bacteriófago T4 dirigido a bacterias E. coli " . Animación de Hybrid Animation Medical . 21 de diciembre de 2009.
- Bacteriófagos: ¿Qué son? Presentación del profesor Graham Hatfull, Universidad de Pittsburgh en YouTube
- Bacteriophages
- Biology