Repetición en tándem de número variable
Una repetición en tándem de número variable (o VNTR ) es una ubicación en un genoma donde una secuencia de nucleótidos corta se organiza como una repetición en tándem . Estos se pueden encontrar en muchos cromosomas y, a menudo, muestran variaciones en la longitud (número de repeticiones) entre los individuos. Cada variante actúa como un alelo heredado , lo que permite su uso para la identificación personal o parental. Su análisis es útil en investigación genética y biológica , medicina forense y huellas dactilares de ADN .
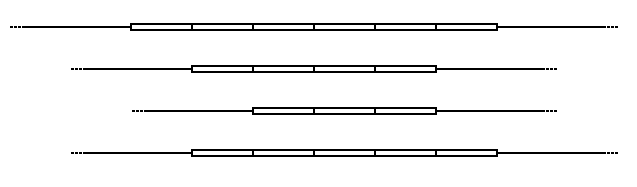
En el esquema anterior, los bloques rectangulares representan cada una de las secuencias de ADN repetidas en una ubicación particular de VNTR. Las repeticiones están en tándem, es decir, están agrupadas y orientadas en la misma dirección. Las repeticiones individuales pueden eliminarse (o agregarse) al VNTR a través de errores de recombinación o replicación , lo que da lugar a alelos con diferentes números de repeticiones. Las regiones flanqueantes son segmentos de secuencia no repetitiva (que se muestran aquí como líneas finas), que permiten extraer los bloques VNTR con enzimas de restricción y analizarlos mediante RFLP , o amplificarlos mediante la técnica de reacción en cadena de la polimerasa (PCR) y determinar su tamaño mediante electroforesis en gel . .
Los VNTR fueron una fuente importante de marcadores genéticos RFLP utilizados en el análisis de ligamiento (mapeo) de genomas diploides. Ahora que se han secuenciado muchos genomas , los VNTR se han vuelto esenciales para las investigaciones de delitos forenses , a través de la toma de huellas dactilares de ADN y la base de datos CODIS . Cuando se eliminan del ADN circundante mediante los métodos PCR o RFLP, y se determina su tamaño mediante electroforesis en gel o transferencia Southern , producen un patrón de bandas único para cada individuo. Cuando se prueba con un grupo de marcadores VNTR independientes, la probabilidad de que dos individuos no relacionados tengan el mismo patrón alelo es extremadamente baja. El análisis VNTR también se está utilizando para estudiardiversidad genética y patrones de reproducción en poblaciones de animales salvajes o domesticados. Como tales, los VNTR se pueden usar para distinguir cepas de patógenos bacterianos. En este contexto de análisis forense microbiano, estos ensayos suelen denominarse análisis VNTR de múltiples loci o MLVA .
El ADN repetitivo , que representa más del 40% del genoma humano, está organizado en una asombrosa variedad de patrones. Las repeticiones se identificaron primero mediante la extracción de Satellite DNA , que no revela cómo están organizadas. El uso de enzimas de restricción mostró que algunos bloques repetidos estaban intercalados en todo el genoma. La secuenciación del ADN mostró más tarde que otras repeticiones se agrupan en ubicaciones específicas, siendo las repeticiones en tándem más comunes que las repeticiones invertidas (que pueden interferir con la replicación del ADN). Los VNTR son la clase de repeticiones en tándem agrupadas que exhiben una variación alélica en sus longitudes.
Los VNTR son un tipo de minisatélite en el que el tamaño de la secuencia repetida es generalmente de diez a cien pares de bases. Los minisatélites son un tipo de secuencia repetida en tándem de ADN , lo que significa que las secuencias se repiten una tras otra sin otras secuencias o nucleótidos entre ellas. Los minisatélites se caracterizan por una secuencia repetida de entre diez y cien nucleótidos, y el número de veces que se repite la secuencia varía entre cinco y cincuenta veces. Las secuencias de minisatélites son más grandes que las de microsatélites, en el que la secuencia repetida es generalmente de 1 a 6 nucleótidos. Los dos tipos de secuencias repetidas son en tándem, pero están especificados por la longitud de la secuencia repetida. Los VNTR, por lo tanto, debido a que tienen secuencias repetidas de diez a cien nucleótidos en las que cada repetición es exactamente igual, se consideran minisatélites. Sin embargo, aunque todos los VNTR son minisatélites, no todos los minisatélites son VNTR. Los VNTR pueden variar en la cantidad de repeticiones de un individuo a otro, como cuando algunos minisatélites que no son VNTR tienen secuencias repetidas que se repiten la misma cantidad de veces en todos los individuos que contienen repeticiones en tándem en sus genomas. [1] [2]