
La acetilación (o en IUPAC nomenclatura ethanoylation ) es un orgánico esterificación reacción con ácido acético . Introduce un grupo funcional acetilo en un compuesto químico . Dichos compuestos se denominan ésteres de acetato o acetatos. La desacetilación es la reacción opuesta, la eliminación de un grupo acetilo de un compuesto químico.
Tanto las reacciones de acetilación como las de desacetilación ocurren dentro de las células vivas como metabolismo de fármacos , por enzimas en el hígado y otros órganos (p. Ej., El cerebro). Los productos farmacéuticos emplean con frecuencia la acetilación para permitir que dichos ésteres atraviesen la barrera hematoencefálica (y la placenta ), donde son desacetilados por enzimas ( carboxilesterasas ) de una manera similar a la acetilcolina . Ejemplos de productos farmacéuticos acetilados son diacetilmorfina (heroína), ácido acetilsalicílico (aspirina), THC-O-acetato y diacereína . Por el contrario, medicamentos como la isoniazidase acetilan en el hígado durante el metabolismo de los fármacos. Un fármaco que depende de tales transformaciones metabólicas para actuar se denomina profármaco .
Para reacciones de esterificación fuera de las células, se usa comúnmente anhídrido acético .
Acetilación de proteínas
La acetilación es una modificación importante de proteínas en biología celular ; y los estudios de proteómica han identificado miles de proteínas de mamíferos acetiladas. [1] [2] [3] La acetilación ocurre como una modificación cotraduccional y postraduccional de proteínas , por ejemplo, histonas , p53 y tubulinas . Entre estas proteínas, las proteínas de la cromatina y las enzimas metabólicas están muy representadas, lo que indica que la acetilación tiene un impacto considerable en la expresión y el metabolismo de los genes . En las bacterias , el 90% de las proteínas implicadas en el metabolismo central deSalmonella enterica están acetiladas. [4] [5]
Acetilación N-terminal

La acetilación N-terminal es una de las modificaciones covalentes cotraduccionales más comunes de proteínas en eucariotas , y es crucial para la regulación y función de diferentes proteínas. La acetilación N-terminal juega un papel importante en la síntesis, estabilidad y localización de proteínas. Aproximadamente el 85% de todas las proteínas humanas y el 68% de las levaduras están acetiladas en su extremo Nα. [6] Varias proteínas de procariotas y arqueas también se modifican mediante acetilación N-terminal.
La acetilación N-terminal es catalizada por un conjunto de complejos enzimáticos, las acetiltransferasas N-terminales (NAT). Los NAT transfieren un grupo acetilo de la acetil-coenzima A (Ac-CoA) al grupo α-amino del primer residuo de aminoácido de la proteína. Diferentes NAT son responsables de la acetilación de la proteína N-terminal naciente, y se encontró que la acetilación era irreversible hasta ahora. [7]
Acetiltransferasas N-terminales
Hasta la fecha, se han encontrado siete NAT diferentes en humanos: NatA, NatB, NatC, NatD, NatE, NatF y NatH. Cada uno de estos diferentes complejos enzimáticos es específico para diferentes aminoácidos o secuencias de aminoácidos, lo que se muestra en la siguiente tabla.
Tabla 1. Composición y especificidad de sustrato de los NAT.
| NAT | Subunidades (las subunidades catalíticas están en negrita ). | Sustratos |
|---|---|---|
| NatA | Naa10 (Ard1) Naa15 (Nat1) | Ser -, Ala -, Gly -, Thr-, Val -, Cys - N-termini |
| NatB | Naa20 (Nat3) Naa25 (Mdm20) | Met - Glu -, Met - Asp -, Met - Asn -, Met - Gln - N-terminal |
| NatC | Naa30 (Mak3) Naa35 (Mak10) Naa38 (Mak31) | Met - Leu -, Met -Ile-, Met -Trp-, Met - Phe - N-termini |
| NatD | Naa40 (Nat4) | Ser - Gly - Gly -, Ser - Gly - Arg - N-termini |
| NatE | Naa50 (Nat5) Naa10 (Ard1) Naa15 (Nat1) | Met -Leu-, Met - Ala -, Met - Lys -, Met - Met - N-terminal |
| NatF | Naa60 | Met - Lys -, Met - Leu -, Met -Ile-, Met -Trp-, Met - Phe - N-termini |
| NatH | Naa80 | Actina - N-terminal |
NatA

NatA se compone de dos subunidades, la subunidad catalítica Naa10 y la subunidad auxiliar Naa15. Las subunidades NatA son más complejas en eucariotas superiores que en eucariotas inferiores. Además de los genes NAA10 y NAA15 , los genes específicos de mamíferos NAA11 y NAA16 producen productos génicos funcionales, que forman diferentes complejos NatA activos. Estas cuatro proteínas forman cuatro posibles dímeros auxiliares catalíticos de hNatA. Sin embargo, Naa10 / Naa15 es la NatA más abundante. [9]
NatA acetila Ser , Ala -, Gly -, Thr-, Val - y Cys N-terminal después de la iniciador de metionina se elimina por la metionina amino-peptidasas. Estos aminoácidos se expresan con mayor frecuencia en el extremo N-terminal de las proteínas en eucariotas, por lo que NatA es el principal NAT correspondiente al número total de sus sustratos potenciales. [10]
Varios socios de interacción diferentes están involucrados en la acetilación N-terminal por NatA. La proteína que interactúa con la huntingtina K (HYPK) interactúa con hNatA en el ribosoma para afectar la acetilación N-terminal de un subconjunto de sustratos de NatA. Las subunidades hNaa10 y hNaa15 aumentarán la tendencia a la agregación de Huntingtin si se agota HYPK. También se ha encontrado que el factor inducible por hipoxia (HIF) -1α interactúa con hNaa10 para inhibir la activación de la actividad transcripcional de β-catenina mediada por hNaa10. [11]
NatB
Los complejos NatB están compuestos por la subunidad catalítica Naa20p y la subunidad auxiliar Naa25p, que se encuentran tanto en levaduras como en seres humanos. En la levadura , todas las subunidades de NatB están asociadas a ribosomas; pero en los seres humanos, se encuentra que las subunidades NatB están asociadas a ribosomas y no ribosomales. NatB acetila la metionina N-terminal de los sustratos comenzando con Met - Glu -, Met - Asp -, Met - Asn - o Met - Gln - N terminales.
NatC
El complejo NatC consta de una subunidad catalítica Naa30p y dos subunidades auxiliares Naa35p y Naa38p. Las tres subunidades se encuentran en el ribosoma de la levadura, pero también se encuentran en formas NAT no ribosómicas como Nat2. NaTC acetilatos complejos la metionina N-terminal de los sustratos Met - Leu -, Met -Ile-, Met Trp- o Met - Phe N-terminales.
NatD
NatD solo está compuesto con la unidad catalítica Naa40p y Naa40p y es conceptualmente diferente de los otros NAT. Al principio, solo se han identificado dos sustratos, H2A y H4, en levaduras y seres humanos. En segundo lugar, la especificidad de sustrato de Naa40p se encuentra dentro de los primeros 30-50 residuos que son bastante más grandes que la especificidad de sustrato de otros NAT. La acetilación de histonas por NatD está parcialmente asociada con ribosomas y los sustratos de aminoácidos son los mismos residuos N-terminales, lo que lo hace diferente de las lisina N-acetiltransferasas (KAT). [12]
NatE
El complejo NatE consta de la subunidad Naa50p y dos subunidades NatA, Naa10p y Naa15p. El extremo N de los sustratos de Naa50p es diferente de los acetilados por la actividad NatA de Naa10p. [13] El NAA50 en las plantas es esencial para controlar el crecimiento, el desarrollo y las respuestas al estrés de las plantas, y la función del NAA50 está muy conservada entre los seres humanos y las plantas. [14] [15] [16] [17]
NatF
NatF es un NAT que se compone de la enzima Naa60. Inicialmente, se pensó que NatF solo se encontraba en eucariotas superiores, ya que estaba ausente de la levadura. [18] Sin embargo, más tarde se descubrió que Naa60 se encuentra en todo el dominio eucariota, pero se perdió secundariamente en el linaje de los hongos. [19] En comparación con la levadura, NatF contribuye a una mayor abundancia de acetilación N-terminal en humanos. NATF acetilatos complejos la metionina N-terminal de los sustratos Met - Lys -, Met - Leu -, Met -Ile-, Met Trp- y Met - Phe N termini que se solapan en parte con NaTC y Nate. [6]Se ha demostrado que NatF tiene una localización organelar y acetila los extremos N-terminales citosólicos de las proteínas transmembrana. [20] La localización organellar de Naa60 está mediada por su único extremo C-terminal, que consta de dos hélices alfa que periféricamente asociado con la membrana y las interacciones mediatas con PI (4) P . [21]
NAA80 / NatH
NAA80 / NatH es una acetiltransferasa N-terminal que acetila específicamente el N-terminal de la actina . [22]
Función de acetilación N-terminal
Estabilidad de las proteínas
La acetilación N-terminal de proteínas puede afectar la estabilidad de las proteínas, pero los resultados y el mecanismo no eran muy claros hasta ahora. [23] Se creía que la acetilación N-terminal protege a las proteínas de la degradación, ya que se suponía que los N-terminales de Nα-acetilación bloquean la ubiquitinación N-terminal y la degradación de proteínas subsiguiente . [24] Sin embargo, varios estudios han demostrado que la proteína acetilada N-terminal tiene una tasa de degradación similar a la de las proteínas con un N-terminal no bloqueado. [25]
Localización de proteínas
Se ha demostrado que la acetilación N-terminal puede dirigir la localización de proteínas. Arl3p es una de las GTPasas 'tipo Arf' (Arl) , que es crucial para la organización del tráfico de membranas. [26] Requiere su grupo Nα-acetilo para dirigirse a la membrana de Golgi mediante la interacción con la proteína Sys1p que reside en la membrana de Golgi. Si Phe o Tyr se reemplaza por un Ala en el N-terminal de Arl3p, ya no puede localizarse en la membrana de Golgi, lo que indica que Arl3p necesita sus residuos N-terminales naturales que podrían acetilarse para una localización adecuada. [27]
Metabolismo y apoptosis
También se ha demostrado que la acetilación N-terminal de proteínas se relaciona con la regulación del ciclo celular y la apoptosis con experimentos de eliminación de proteínas. La caída del complejo NatA o NatC conduce a la inducción de apoptosis dependiente de p53 , lo que puede indicar que las proteínas antiapoptóticas eran menos funcionales o ya no funcionaban debido a la reducción de la acetilación del extremo N de la proteína. [28] Pero, por el contrario, la caspasa-2 , que es acetilada por NatA, puede interactuar con la proteína adaptadora RIP asociada a la proteína homóloga Ich-1 / Ced-3 con un dominio de muerte (RAIDD). Esto podría activar la caspasa-2 e inducir la apoptosis celular . [29]
Síntesis de proteínas
Las proteínas ribosómicas juegan un papel importante en la síntesis de proteínas, que también podrían acetilarse en el extremo N-terminal. La acetilación N-terminal de las proteínas del ribosoma puede tener un efecto sobre la síntesis de proteínas. Se observó una disminución del 27% y 23% en la tasa de síntesis de proteínas con las cepas de deleción NatA y NatB. Se observó una reducción de la fidelidad de la traducción en la cepa de deleción de NatA y se observó un defecto en el ribosoma en la cepa de deleción de NatB. [30]
Cáncer
Se ha sugerido que los NAT actúan como oncoproteínas y supresores de tumores en cánceres humanos, y la expresión de NAT puede aumentar y disminuir en las células cancerosas. La expresión ectópica de hNaa10p aumentó la proliferación celular y la regulación positiva del gen involucrado en la proliferación y el metabolismo de la supervivencia celular . La sobreexpresión de hNaa10p estaba en el cáncer de vejiga urinaria , cáncer de mama y carcinoma de cuello uterino . [31] Pero un alto nivel de expresión de hNaa10p también podría suprimir el crecimiento tumoral y un nivel reducido de hNaa10p expresado se asocia con un pronóstico precario, tumores grandes y más metástasis en los ganglios linfáticos.
Tabla 2. Descripción general de la expresión de subunidades de NatA en varios tejidos cancerosos [32]
| Subunidades nat | Tejido canceroso | Patrón de expresión |
|---|---|---|
| hNaa10 | cáncer de pulmón , cáncer de mama , cáncer colorrectal , carcinoma hepatocelular | alto en tumores |
| hNaa10 | cáncer de pulmón , cáncer de mama , cáncer de páncreas , cáncer de ovario | pérdida de heterocigosidad en tumores |
| hNaa10 | cáncer de mama , cáncer gástrico , cáncer de pulmón | alto en tumores primarios, pero bajo con metástasis en los ganglios linfáticos |
| hNaa10 | Cáncer de pulmón de células no pequeñas | bajo en tumores |
| hNaa15 | carcinoma papilar de tiroides , cáncer gástrico | alto en tumores |
| hNaa15 | neuroblastoma | alto en tumores en estadio avanzado |
| hNaa11 | carcinoma hepatocelular | pérdida de heterocigosidad en tumores |
Acetilación y desacetilación de lisina

Las proteínas se acetilan típicamente en residuos de lisina y esta reacción se basa en acetil-coenzima A como donante de grupo acetilo. En la acetilación y desacetilación de histonas, las proteínas de histonas se acetilan y desacetilan en residuos de lisina en la cola N-terminal como parte de la regulación génica . Normalmente, estas reacciones son catalizadas por enzimas con actividad histona acetiltransferasa (HAT) o histona desacetilasa (HDAC), aunque las HAT y HDAC también pueden modificar el estado de acetilación de proteínas que no son histonas. [33]
La regulación de factores de transcripción, proteínas efectoras, chaperonas moleculares y proteínas citoesqueléticas por acetilación y desacetilación es un importante mecanismo regulador postraduccional [34]. Estos mecanismos reguladores son análogos a la fosforilación y desfosforilación por la acción de quinasas y fosfatasas . El estado de acetilación de una proteína no solo puede modificar su actividad, sino que recientemente se ha sugerido que esta modificación postraduccional también puede dialogar con la fosforilación , metilación , ubiquitinación , sumoilación y otras para el control dinámico de la señalización celular. [35] El reglamento deLa proteína tubulina es un ejemplo de esto en las neuronas y astroglias de ratón. [36] [37] Una tubulina acetiltransferasa se encuentra en el axonema y acetila la subunidad α-tubulina en un microtúbulo ensamblado. Una vez desmontada, esta acetilación es eliminada por otra desacetilasa específica en el citosol celular. Por tanto, los microtúbulos axonémicos, que tienen una vida media larga, llevan una "acetilación característica", que está ausente en los microtúbulos citosólicos que tienen una vida media más corta.
En el campo de la epigenética , se ha demostrado que la acetilación (y desacetilación ) de histonas son mecanismos importantes en la regulación de la transcripción de genes. Las histonas, sin embargo, no son las únicas proteínas reguladas por acetilación postraduccional . Los siguientes son ejemplos de varias otras proteínas con funciones en la regulación de la transducción de señales, cuyas actividades también se ven afectadas por la acetilación y desacetilación.
p53
La proteína p53 es un supresor de tumores que juega un papel importante en las transacciones de señales en las células, especialmente en el mantenimiento de la estabilidad del genoma al prevenir la mutación. Por lo tanto, también se le conoce como "el guardián del genoma". También regula el ciclo celular y detiene el crecimiento celular activando un regulador del ciclo celular, p21 . En caso de daño severo del ADN , también inicia la muerte celular programada . de p53 está regulada negativamente por la oncoproteína Mdm2 . Los estudios sugirieron que Mdm2 formará un complejo con p53y evitar que se una a genes específicos que responden a p53. [38] [39]
Acetilación de p53

La acetilación de p53 es indispensable para su activación. Se ha informado que el nivel de acetilación de p53 aumentará significativamente cuando la célula sufra estrés. Se han observado sitios de acetilación en el dominio de unión al ADN (K164 y K120) y el terminal C. [40] Los sitios de acetilación demuestran una redundancia significativa: si solo un sitio de acetilación se inactiva por mutación a arginina, todavía se observa la expresión de p21 . Sin embargo, si se bloquean múltiples sitios de acetilación, la expresión de p21 y la supresión del crecimiento celular causada por p53 se pierden por completo. Además, la acetilación de p53 evita su unión al represor Mdm2 en el ADN. [41]Además, se sugiere que la acetilación de p53 es crucial para sus funciones proapoptóticas independientes de la transcripción . [42] Se investigó un sitio de acetilación del extremo C-terminal mediante simulaciones de dinámica molecular y espectroscopía de dicroísmo circular , y se sugirió que la acetilación cambia el conjunto estructural del extremo C-terminal. [43]
Implicaciones para la terapia del cáncer
Dado que la función principal de p53 es supresor de tumores , la idea de que la activación de p53 es una estrategia atractiva para el tratamiento del cáncer. Nutlin-3 [44] es una pequeña molécula diseñada para dirigirse a la interacción p53 y Mdm2 que impidió la desactivación de p53 . [45] Los informes también mostraron que la célula cancerosa bajo el tratamiento con Nutilin-3a, la acetilación de lys 382 se observó en el c-terminal de p53. [46] [47]
Microtúbulos

La estructura de los microtúbulos es un cilindro largo y hueco ensamblado dinámicamente a partir de dímeros de tubulina α / β . Desempeñan un papel esencial en el mantenimiento de la estructura de la célula, así como en los procesos celulares, por ejemplo, el movimiento de los orgánulos . [48] Además, los microtúbulos son responsables de formar el huso mitótico en las células eucariotas para transportar los cromosomas en la división celular . [49] [50]
Acetilación de tubulina
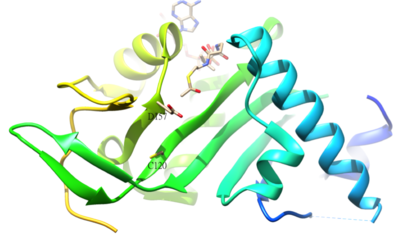
El residuo acetilado de α- tubulina es K40, que es catalizado por α-tubulina acetiltransferasa (α-TAT) en humanos. La acetilación de K40 en α-tubulina es un sello distintivo de los microtúbulos estables . Los residuos del sitio activo D157 y C120 de α-TAT1 son responsables de la catálisis debido a la forma complementaria de α-Tubulina. Además, algunas características estructurales únicas como las regiones de horquilla β4-β5 , bucle C-terminal y bucle α1-α2 son importantes para el reconocimiento molecular específico de la α-tubulina . [51] La reacción inversa de la acetilación es catalizada por la histona desacetilasa 6. [52]
Implicaciones para la terapia del cáncer
Dado que los microtúbulos desempeñan un papel importante en la división celular , especialmente en la fase G2 / M del ciclo celular , se han realizado intentos para impedir la función de los microtúbulos utilizando inhibidores de moléculas pequeñas, que se han utilizado con éxito en clínicas como terapias contra el cáncer. [53] Por ejemplo, los alcaloides y taxanos de la vinca se unen e inhiben selectivamente los microtúbulos , lo que lleva a la detención del ciclo celular. [54]La identificación de la estructura cristalina de la acetilación de la α-tubulina acetiltransferasa (α-TAT) también arroja luz sobre el descubrimiento de una molécula pequeña que podría modular la estabilidad o despolimerización de la tubulina . En otras palabras, al dirigirse a α-TAT, es posible evitar la acetilación de la tubulina y dar como resultado la desestabilización de la tubulina, que es un mecanismo similar para los agentes desestabilizadores de tubulina. [51]
STAT3
El transductor de señal y activador de la transcripción 3 ( STAT3 ) es un factor de transcripción que es fosforilado por quinasas asociadas al receptor , por ejemplo, tirosina quinasas de la familia Janus , y que se transloca al núcleo . STAT3 regula varios genes en respuesta a factores de crecimiento y citocinas y juega un papel importante en el crecimiento celular. Por lo tanto, STAT3 facilita la oncogénesis en una variedad de vías relacionadas con el crecimiento celular. Por otro lado, también juega un papel en el supresor de tumores . [55]
Acetilación de STAT3

La acetilación de Lys685 de STAT3 es importante para STAT3 homo-dimerización, que es esencial para la unión al ADN y la activación transcripcional de oncogenes . La acetilación de STAT3 es catalizada por la histona acetiltransferasa p300 y revertida por la histona desacetilasa tipo 1 . La acetilación de lisina de STAT3 también está elevada en las células cancerosas. [56]
Implicaciones terapéuticas para la terapia del cáncer
Dado que la acetilación de STAT3 es importante por su actividad oncogénica y el hecho de que el nivel de STAT3 acetilado es alto en las células cancerosas, se da a entender que apuntar a STAT3 acetilado para quimioprevención y quimioterapia es una estrategia prometedora. Esta estrategia está respaldada por el tratamiento del resveratrol , un inhibidor de la acetilación de STAT3, en la línea de células cancerosas que revierte la metilación aberrante de la isla CpG. [57]
Acetilación de madera
Desde principios del siglo XX, se investigó la acetilación de la madera como método para mejorar la durabilidad de la madera en cuanto a resistencia contra procesos de descomposición y mohos. Los beneficios secundarios incluyen la mejora de la estabilidad dimensional, la mejora de la dureza de la superficie y la ausencia de disminución de las propiedades mecánicas debido al tratamiento.
Las propiedades físicas de cualquier material están determinadas por su estructura química. La madera contiene una gran cantidad de grupos químicos llamados hidroxilos libres . Los grupos hidroxilo libres adsorben y liberan agua de acuerdo con los cambios en las condiciones climáticas a las que está expuesta la madera. Ésta es una explicación de por qué la madera se hincha y se encoge. También se cree que la digestión de la madera por enzimas se inicia en los sitios de hidroxilo libre, que es una de las principales razones por las que la madera es propensa a descomponerse.
La acetilación transforma los hidroxilos libres dentro de la madera en grupos acetilo. Esto se hace haciendo reaccionar la madera con anhídrido acético, que proviene del ácido acético (conocido como vinagre cuando está diluido). Cuando el grupo hidroxilo libre se transforma en un grupo acetilo, la capacidad de la madera para absorber agua se reduce en gran medida, lo que hace que la madera sea más estable dimensionalmente y, debido a que ya no es digerible, extremadamente duradera.
En 2007, Titan Wood, una empresa con sede en Londres, con instalaciones de producción en los Países Bajos, logró una comercialización rentable y comenzó la producción a gran escala de madera acetilada con el nombre comercial "Accoya".
Ver también
- Grupo acetoxi
- Acilación
- Amida
- Compendio de acetilación de proteína lisina
- Ester
- Glicosilación
- Lipidación
- Nitrosilación
- Síntesis orgánica
- Proteólisis
Referencias
- ^ Choudhary C, Kumar C, Gnad F, Nielsen ML, Rehman M, Walther TC, Olsen JV, Mann M (2009). "La acetilación de lisina se dirige a los complejos de proteínas y co-regula las principales funciones celulares". Ciencia . 325 (5942): 834–840. Código Bibliográfico : 2009Sci ... 325..834C . doi : 10.1126 / science.1175371 . PMID 19608861 . S2CID 206520776 .
- ^ Fritz KS, Galligan JJ, Hirschey MD, Verdin E, Petersen DR (2012). "Análisis de acetiloma mitocondrial en un modelo de ratón de lesión hepática inducida por alcohol utilizando ratones knockout SIRT3" . J. Proteome Res . 11 (3): 1633–1643. doi : 10.1021 / pr2008384 . PMC 3324946 . PMID 22309199 .
- ^ Brook T. "Acetilación de proteínas: mucho más que acetilación de histonas" . Cayman Chemical. Archivado desde el original el 28 de febrero de 2014.
- ^ Zhao S, Xu W, Jiang W, Yu W, Lin Y, Zhang T, Yao J, Zhou L, Zeng Y, Li H, Li Y, Shi J, An W, Hancock SM, He F, Qin L, Chin J, Yang P, Chen X, Lei Q, Xiong Y, Guan KL (2010). "Regulación del metabolismo celular por acetilación de lisina de proteínas" . Ciencia . 327 (5968): 1000–1004. Código Bibliográfico : 2010Sci ... 327.1000Z . doi : 10.1126 / science.1179689 . PMC 3232675 . PMID 20167786 .
- ^ Wang Q, Zhang Y, Yang C, Xiong H, Lin Y, Yao J, et al. (Febrero de 2010). "La acetilación de enzimas metabólicas coordina la utilización de la fuente de carbono y el flujo metabólico" . Ciencia . 327 (5968): 1004–7. Código Bibliográfico : 2010Sci ... 327.1004W . doi : 10.1126 / science.1179687 . PMC 4183141 . PMID 20167787 .
- ↑ a b Van Damme P, Hole K, Pimenta-Marques A, Helsens K, Vandekerckhove J, Martinho RG, Gevaert K, Arnesen T (2011). "NatF contribuye a un cambio evolutivo en la acetilación N-terminal de la proteína y es importante para la segregación cromosómica normal" . PLOS Genet . 7 (7): e1002169. doi : 10.1371 / journal.pgen.1002169 . PMC 3131286 . PMID 21750686 .
- ^ Starheim KK, Gevaert K, Arnesen T (2012). "Proteínas acetiltransferasas N-terminales: cuando el comienzo importa". Trends Biochem. Sci . 37 (4): 152-161. doi : 10.1016 / j.tibs.2012.02.003 . PMID 22405572 .
- ^ Liszczak G, Goldberg JM, Foyn H, Petersson EJ, Arnesen T, Marmorstein R (2013). "Base molecular para la acetilación N-terminal por el complejo NatA heterodimérico" . Nat. Struct. Mol. Biol . 20 (9): 1098-105. doi : 10.1038 / nsmb.2636 . PMC 3766382 . PMID 23912279 .
- ^ Starheim KK, Gromyko D, Velde R, Varhaug JE, Arnesen T (2009). "Composición y significado biológico de las acetiltransferasas Nalfa-terminales humanas" . Procedimientos de BMC . 3 Suppl 6 (Suppl 6): S3. doi : 10.1186 / 1753-6561-3-s6-s3 . PMC 2722096 . PMID 19660096 .
- ^ Arnesen T, Van Damme P, Polevoda B, Helsens K, Evjenth R, Colaert N, Varhaug JE, Vandekerckhove J, Lillehaug JR, Sherman F, Gevaert K (2009). "Los análisis de proteómica revelan la conservación evolutiva y la divergencia de acetiltransferasas N-terminales de levadura y humanos" . Proc. Natl. Acad. Sci. USA . 106 (20): 8157–8162. Código Bibliográfico : 2009PNAS..106.8157A . doi : 10.1073 / pnas.0901931106 . PMC 2688859 . PMID 19420222 .
- ^ Arnesen T, Starheim KK, Van Damme P, Evjenth R, Dinh H, Betts MJ, Ryningen A, Vandekerckhove J, Gevaert K, Anderson D (2010). "La proteína tipo chaperona HYPK actúa junto con NatA en la acetilación N-terminal cotraduccional y la prevención de la agregación de Huntingtin" . Mol. Celda. Biol . 30 (8): 1898-1909. doi : 10.1128 / mcb.01199-09 . PMC 2849469 . PMID 20154145 .
- ^ Hoyo K, Van Damme P, Dalva M, Aksnes H, Glomnes N, Varhaug JE, Lillehaug JR, Gevaert K, Arnesen T (2011). "La N-alfa-acetiltransferasa 40 humana (hNaa40p / hNatD) se conserva de la levadura y acetila en el extremo N las histonas H2A y H4" . PLOS ONE . 6 (9): e24713. Código bibliográfico : 2011PLoSO ... 624713H . doi : 10.1371 / journal.pone.0024713 . PMC 3174195 . PMID 21935442 .
- ^ Gautschi M, Just S, Mun A, Ross S, Rücknagel P, Dubaquié Y, et al. (Octubre de 2003). "La levadura N (alfa) -acetiltransferasa NatA se ancla cuantitativamente al ribosoma e interactúa con polipéptidos nacientes" . Biología Molecular y Celular . 23 (20): 7403-14. doi : 10.1128 / mcb.23.20.7403-7414.2003 . PMC 230319 . PMID 14517307 .
- ^ Hartman S (agosto de 2020). "El significado de un fin: N-terminal acetiltransferasa NAA50 controla el crecimiento de las plantas y las respuestas al estrés" . Fisiología vegetal . 183 (4): 1410–1411. doi : 10.1104 / pp.20.00794 . PMID 32747486 .
- ^ Armbruster L, Linster E, Boyer JB, Brünje A, Eirich J, Stephan I, et al. (Agosto de 2020). "NAA50 es una N α- acetiltransferasa enzimáticamente activa que es crucial para el desarrollo y la regulación de las respuestas al estrés" . Fisiología vegetal . 183 (4): 1502-1516. doi : 10.1104 / pp.20.00222 . PMID 32461302 .
- ^ Neubauer M, Innes RW (agosto de 2020). "La pérdida de la acetiltransferasa NAA50 induce el estrés del retículo endoplásmico y las respuestas inmunitarias y suprime el crecimiento" . Fisiología vegetal . 183 (4): 1838–1854. doi : 10.1104 / pp.20.00225 . PMID 32457093 .
- ^ Feng J, Hu J, Li Y, Li R, Yu H, Ma L (septiembre de 2020). "La acetiltransferasa N-terminal Naa50 regula el crecimiento de Arabidopsis y la respuesta al estrés osmótico". Fisiología vegetal y celular . 61 (9): 1565-1575. doi : 10.1093 / pcp / pcaa081 . PMID 32544241 .
- ^ Van Damme P, Hole K, Pimenta-Marques A, Helsens K, Vandekerckhove J, Martinho RG, et al. (Julio de 2011). "NatF contribuye a un cambio evolutivo en la acetilación N-terminal de la proteína y es importante para la segregación cromosómica normal" . PLoS Genetics . 7 (7): e1002169. doi : 10.1371 / journal.pgen.1002169 . PMC 3131286 . PMID 21750686 .
- ^ Rathore OS, Faustino A, Prudêncio P, Van Damme P, Cox CJ, Martinho RG (febrero de 2016). "Ausencia de diversificación de acetiltransferasa N-terminal durante la evolución de organismos eucariotas" . Informes científicos . 6 : 21304. Bibcode : 2016NatSR ... 621304R . doi : 10.1038 / srep21304 . PMC 4748286 . PMID 26861501 .
- ^ Aksnes H, Van Damme P, Goris M, Starheim KK, Marie M, Støve SI, et al. (Marzo de 2015). "Un organelar nα-acetiltransferasa, naa60, acetila los terminales N citosólicos de las proteínas transmembrana y mantiene la integridad de Golgi" . Informes de celda . 10 (8): 1362–74. doi : 10.1016 / j.celrep.2015.01.053 . PMID 25732826 .
- ^ Aksnes H, Goris M, Strømland Ø, Drazic A, Waheed Q, Reuter N, Arnesen T (abril de 2017). "Determinantes moleculares del anclaje N-terminal acetiltransferasa Naa60 a la membrana de Golgi" . La revista de química biológica . 292 (16): 6821–6837. doi : 10.1074 / jbc.M116.770362 . PMC 5399128 . PMID 28196861 .
- ^ Drazic A, Aksnes H, Marie M, Boczkowska M, Varland S, Timmerman E, Foyn H, Glomnes N, Rebowski G, Impens F, Gevaert K, Dominguez R y Arnesen T (2018). "NAA80 es la acetiltransferasa N-terminal de la actina y regula el ensamblaje del citoesqueleto y la motilidad celular" . Proc Natl Acad Sci USA . 115 (17): 4399–4404. doi : 10.1073 / pnas.1718336115 . PMC 5924898 . PMID 29581253 .
- ^ Hollebeke J, Van Damme P, Gevaert K (2012). "Acetilación N-terminal y otras funciones de Nα-acetiltransferasas". Biol. Chem . 393 (4): 291–8. doi : 10.1515 / hsz-2011-0228 . PMID 22718636 .
- ^ Hershko A, Heller H, Eytan E, Kaklij G, Rose IA (1984). "Papel del grupo alfa-amino de la proteína en la degradación de proteínas mediada por ubiquitina" . Proc. Natl. Acad. Sci. USA . 81 (22): 7021–5. Código Bibliográfico : 1984PNAS ... 81.7021H . doi : 10.1073 / pnas.81.22.7021 . PMC 392068 . PMID 6095265 .
- ^ Hwang CS, Shemorry A, Varshavsky A (2010). "La acetilación N-terminal de proteínas celulares crea señales de degradación específicas" . Ciencia . 327 (5968): 973–977. Código bibliográfico : 2010Sci ... 327..973H . doi : 10.1126 / science.1183147 . PMC 4259118 . PMID 20110468 .
- ^ Behnia R, Pánico B, Whyte JR, Munro S (2004). "La orientación de la GTPasa Arl3p similar a Arf al Golgi requiere acetilación N-terminal y la proteína de membrana Sys1p". Nat. Cell Biol . 6 (5): 405–413. doi : 10.1038 / ncb1120 . PMID 15077113 .
- ^ Starheim KK, Gromyko D, Evjenth R, Ryningen A, Varhaug JE, Lillehaug JR, Arnesen T (2009). "La caída del complejo C de acetiltransferasa N alfa-terminal humana conduce a la apoptosis dependiente de p53 y la localización aberrante de Arl8b humana" . Mol. Celda. Biol . 29 (13): 3569–3581. doi : 10.1128 / mcb.01909-08 . PMC 2698767 . PMID 19398576 .
- ^ Gromyko D, Arnesen T, Ryningen A, Varhaug JE, Lillehaug JR (2010). "El agotamiento de la acetiltransferasa A Nα-terminal humana induce la apoptosis dependiente de p53 y la inhibición del crecimiento independiente de p53" . En t. J. Cancer . 127 (12): 2777–2789. doi : 10.1002 / ijc.25275 . PMID 21351257 .
- ^ Yi CH, Pan H, Seebacher J, Jang IH, Hyberts SG, Heffron GJ, Vander Heiden MG, Yang R, Li F, Locasale JW, Sharfi H, Zhai B, Rodriguez-Mias R, Luithardt H, Cantley LC, Daley GQ, Asara JM, Gygi SP, Wagner G, Liu CF, Yuan J (2011). "La regulación metabólica de la proteína N-alfa-acetilación por Bcl-xL promueve la supervivencia celular" . Celular . 146 (4): 607–620. doi : 10.1016 / j.cell.2011.06.050 . PMC 3182480 . PMID 21854985 .
- ^ Kamita M, Kimura Y, Ino Y, Kamp RM, Polevoda B, Sherman F, Hirano H (2011). "N (α) -Acetilación de proteínas ribosomales de levadura y su efecto sobre la síntesis de proteínas". J Proteomics . 74 (4): 431–441. doi : 10.1016 / j.jprot.2010.12.007 . PMID 21184851 .
- ^ Yu M, Gong J, Ma M, Yang H, Lai J, Wu H, Li L, Li L, Tan D (2009). "Análisis inmunohistoquímico de detención humana defectuosa-1 expresada en cánceres in vivo" . Oncol. Rep . 21 (4): 909-15. doi : 10.3892 / o_00000303 . PMID 19287988 .
- ^ Kalvik TV, Arnesen T (2013). "Proteínas acetiltransferasas N-terminales en cáncer" . Oncogén . 32 (3): 269–276. doi : 10.1038 / onc.2012.82 . PMID 22391571 .
- ^ Sadoul K, Boyault C, Pabion M, Khochbin S (2008). "Regulación del recambio proteico por acetiltransferasas y desacetilasas". Biochimie . 90 (2): 306–12. doi : 10.1016 / j.biochi.2007.06.009 . PMID 17681659 .
- ^ Glozak MA, Sengupta N, Zhang X, Seto E (2005). "Acetilación y desacetilación de proteínas no histonas". Gene . 363 : 15-23. doi : 10.1016 / j.gene.2005.09.010 . PMID 16289629 .
- ↑ Yang XJ, Seto E (2008). "Acetilación de lisina: diafonía codificada con otras modificaciones postraduccionales" . Mol. Celular . 31 (4): 449–61. doi : 10.1016 / j.molcel.2008.07.002 . PMC 2551738 . PMID 18722172 .
- ^ Eddé B, Denoulet P, de Néchaud B, Koulakoff A, Berwald-Netter Y, Gros F (1989). "Modificaciones postraduccionales de tubulina en neuronas de cerebro de ratón cultivadas y astroglia". Biol. Celular . 65 (2): 109-117. doi : 10.1016 / 0248-4900 (89) 90018-x . PMID 2736326 .
- ^ Maruta H, Greer K, Rosenbaum JL (1986). "La acetilación de la alfa-tubulina y su relación con el montaje y desmontaje de microtúbulos" . J. Cell Biol . 103 (2): 571–579. doi : 10.1083 / jcb.103.2.571 . PMC 2113826 . PMID 3733880 .
- ^ Alberts B (marzo de 2002). Biología molecular de la célula . Garland Science. ISBN 0815332181.
- ^ Weinberg RA (2013). Biología del cáncer (2. ed.). [Sl]: Garland Science. ISBN 978-0815342205.
- ^ Brooks CL, Gu W (2011). "El impacto de la acetilación y desacetilación en la vía p53" . Célula de proteína . 2 (6): 456–462. doi : 10.1007 / s13238-011-1063-9 . PMC 3690542 . PMID 21748595 .
- ^ Tang Y, Zhao W, Chen Y, Zhao Y, Gu W (2008). "La acetilación es indispensable para la activación de p53" . Celular . 133 (4): 612–626. doi : 10.1016 / j.cell.2008.03.025 . PMC 2914560 . PMID 18485870 .
- ^ Yamaguchi H, Woods NT, Piluso LG, Lee HH, Chen J, Bhalla KN, Monteiro A, Liu X, Hung MC, Wang HG (2009). "La acetilación de p53 es crucial para sus funciones proapoptóticas independientes de la transcripción" . J. Biol. Chem . 284 (17): 11171–11183. doi : 10.1074 / jbc.M809268200 . PMC 2670122 . PMID 19265193 .
- ^ Iida S, Mashimo T, Kurosawa T, Hojo H, Muta H, Goto Y, et al. (Diciembre de 2016). "Variación del paisaje de energía libre del dominio C-terminal de p53 inducida por acetilación: muestreo conformacional mejorado" . Revista de Química Computacional . 37 (31): 2687–2700. doi : 10.1002 / jcc.24494 . PMC 5242334 . PMID 27735058 .
- ^ Vassilev LT, Vu BT, Graves B, Carvajal D, Podlaski F, Filipovic Z, Kong N, Kammlott U, Lukacs C, Klein C, Fotouhi N, Liu EA (2004). "Activación in vivo de la vía p53 por antagonistas de moléculas pequeñas de MDM2". Ciencia . 303 (5659): 844–848. Código bibliográfico : 2004Sci ... 303..844V . doi : 10.1126 / science.1092472 . PMID 14704432 .
- ^ Shangary S, Wang S (2009). "Inhibidores de moléculas pequeñas de la interacción proteína-proteína MDM2-p53 para reactivar la función p53: un enfoque novedoso para la terapia del cáncer" . Annu. Rev. Pharmacol. Toxicol . 49 (1): 223–241. doi : 10.1146 / annurev.pharmtox.48.113006.094723 . PMC 2676449 . PMID 18834305 .
- ↑ Zajkowicz A, Krześniak M, Matuszczyk I, Głowala-Kosińska M, Butkiewicz D, Rusin M (2013). "Nutlin-3a, un antagonista de MDM2 y activador de p53, ayuda a preservar el potencial replicativo de las células cancerosas tratadas con una dosis genotóxica de resveratrol" . Mol. Biol. Rep . 40 (8): 5013–5026. doi : 10.1007 / s11033-013-2602-7 . PMC 3723979 . PMID 23666059 .
- ^ Kumamoto K, Spillare EA, Fujita K, Horikawa I, Yamashita T, Appella E, Nagashima M, Takenoshita S, Yokota J, Harris CC (2008). "Nutlin-3a activa p53 para regular a la baja el inhibidor del crecimiento 2 y regular al alza la expresión de mir-34a, mir-34b y mir-34c, e inducir la senescencia" . Cancer Res . 68 (9): 3193–3203. doi : 10.1158 / 0008-5472.CAN-07-2780 . PMC 2440635 . PMID 18451145 .
- ^ Kreis T, Vale R, eds. (1999). Guía de proteínas citoesqueléticas y motoras (2ª ed.). Oxford [ua]: Universidad de Oxford. Prensa. ISBN 0198599560.
- ^ Lodish H (2013). Biología celular molecular (7ª ed.). Nueva York: WH Freeman and Co. ISBN 978-1429234139.
- ^ Fojo T, ed. (2008). El papel de los microtúbulos en la biología celular, neurobiología y oncología ([Online-Ausg.] Ed.). Totowa, Nueva Jersey: Humana Press. ISBN 978-1588292940.
- ↑ a b Friedmann DR, Aguilar A, Fan J, Nachury MV, Marmorstein R (2012). "Estructura de la α-tubulina acetiltransferasa, αTAT1 e implicaciones para la acetilación específica de tubulina" . Proc. Natl. Acad. Sci. USA . 109 (48): 19655–19660. Código bibliográfico : 2012PNAS..10919655F . doi : 10.1073 / pnas.1209357109 . PMC 3511727 . PMID 23071314 .
- ↑ Hubbert C, Guardiola A, Shao R, Kawaguchi Y, Ito A, Nixon A, Yoshida M, Wang XF, Yao TP (2002). "HDAC6 es una desacetilasa asociada a microtúbulos". Naturaleza . 417 (6887): 455–458. Código Bibliográfico : 2002Natur.417..455H . doi : 10.1038 / 417455a . PMID 12024216 .
- ^ Teresa Carlomagno, ed. (2009). Agentes aglutinantes de tubulina: conocimientos sintéticos, estructurales y mecanicistas . contribuciones de K.-H. Altmann. Berlín: Springer. ISBN 978-3540690368.
- ^ Zito TL, Lemke TL, Williams DA, Roche VG, William S, eds. (2013). Principios de química medicinal de Foye (7ª ed.). Filadelfia: Wolters Kluwer Health / Lippincott Williams & Wilkins. ISBN 978-1609133450.
- ^ Müller-Decker FM, Klingmüller Reino Unido (2009). Procesamiento de señales celulares: una introducción a los mecanismos moleculares de la transducción de señales . Nueva York: Garland Science. ISBN 978-0815342151.
- ^ Yuan ZL, Guan YJ, Chatterjee D, Chin YE (2005). "Dimerización Stat3 regulada por acetilación reversible de un solo residuo de lisina". Ciencia . 307 (5707): 269–273. Código bibliográfico : 2005Sci ... 307..269Y . doi : 10.1126 / science.1105166 . PMID 15653507 .
- ^ Lee H, Zhang P, Herrmann A, Yang C, Xin H, Wang Z, Hoon DS, Forman SJ, Jove R, Riggs AD, Yu H (2012). "STAT3 acetilado es crucial para la metilación de los promotores de genes supresores de tumores y la inhibición por resveratrol da como resultado la desmetilación" . Proc. Natl. Acad. Sci. USA . 109 (20): 7765–7769. Código bibliográfico : 2012PNAS..109.7765L . doi : 10.1073 / pnas.1205132109 . PMC 3356652 . PMID 22547799 .